+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10104 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of Azospirillum brasilense Glutamate Synthase in a4b4 oligomeric state. | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | glutamate synthesys / complex / oligomeric assemblies / FLAVOPROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報glutamate synthase (NADPH) / glutamate synthase (NADPH) activity / L-glutamate biosynthetic process / ammonia assimilation cycle / 3 iron, 4 sulfur cluster binding / 4 iron, 4 sulfur cluster binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Azospirillum brasilense (バクテリア) Azospirillum brasilense (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.9 Å | |||||||||
 データ登録者 データ登録者 | Swuec P | |||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2019 ジャーナル: J Mol Biol / 年: 2019タイトル: Cryo-EM Structures of Azospirillum brasilense Glutamate Synthase in Its Oligomeric Assemblies. 著者: Paolo Swuec / Antonio Chaves-Sanjuan / Carlo Camilloni / Maria Antonietta Vanoni / Martino Bolognesi /  要旨: Bacterial NADPH-dependent glutamate synthase (GltS) is a complex iron-sulfur flavoprotein that catalyzes the reductive synthesis of two L-Glu molecules from L-Gln and 2-oxo-glutarate. GltS functional ...Bacterial NADPH-dependent glutamate synthase (GltS) is a complex iron-sulfur flavoprotein that catalyzes the reductive synthesis of two L-Glu molecules from L-Gln and 2-oxo-glutarate. GltS functional unit hosts an α-subunit (αGltS) and a β-subunit (βGltS) that assemble in different αβ oligomers in solution. Here, we present the cryo-electron microscopy structures of Azospirillum brasilense GltS in four different oligomeric states (αβ, αβ, αβ and αβ, in the 3.5- to 4.1-Å resolution range). Our study provides a comprehensive GltS model that details the inter-protomeric assemblies and allows unequivocal location of the FAD cofactor and of two electron transfer [4Fe-4S] clusters within βGltS. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10104.map.gz emd_10104.map.gz | 104.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10104-v30.xml emd-10104-v30.xml emd-10104.xml emd-10104.xml | 24.6 KB 24.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10104_fsc.xml emd_10104_fsc.xml | 10.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10104.png emd_10104.png | 167.6 KB | ||
| マスクデータ |  emd_10104_msk_1.map emd_10104_msk_1.map | 111.5 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-10104.cif.gz emd-10104.cif.gz | 7.5 KB | ||
| その他 |  emd_10104_additional.map.gz emd_10104_additional.map.gz emd_10104_half_map_1.map.gz emd_10104_half_map_1.map.gz emd_10104_half_map_2.map.gz emd_10104_half_map_2.map.gz | 87.5 MB 87.7 MB 87.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10104 http://ftp.pdbj.org/pub/emdb/structures/EMD-10104 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10104 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10104 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10104.map.gz / 形式: CCP4 / 大きさ: 111.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10104.map.gz / 形式: CCP4 / 大きさ: 111.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.426 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_10104_msk_1.map emd_10104_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
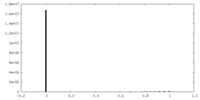
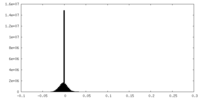
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_10104_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
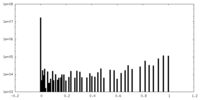
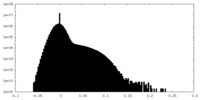
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_10104_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
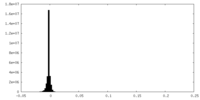
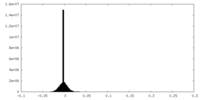
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_10104_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
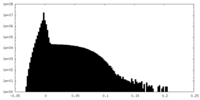
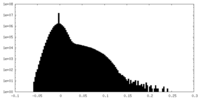
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Glutamate Synthase complex in a4b4 oligomeric state
| 全体 | 名称: Glutamate Synthase complex in a4b4 oligomeric state |
|---|---|
| 要素 |
|
-超分子 #1: Glutamate Synthase complex in a4b4 oligomeric state
| 超分子 | 名称: Glutamate Synthase complex in a4b4 oligomeric state / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Azospirillum brasilense (バクテリア) Azospirillum brasilense (バクテリア) |
-分子 #1: Glutamate synthase [NADPH] large chain
| 分子 | 名称: Glutamate synthase [NADPH] large chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO / EC番号: glutamate synthase (NADPH) |
|---|---|
| 由来(天然) | 生物種:  Azospirillum brasilense (バクテリア) Azospirillum brasilense (バクテリア) |
| 分子量 | 理論値: 166.224734 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTTELNQGEQ FVADFRANAA ALTTANAYNP EDEHDACGVG FIAAIDGKPR RSVVEKGIEA LKAVWHRGAV DADGKTGDGA GIHVAVPQK FFKDHVKVIG HRAPDNKLAV GQVFLPRISL DAQEACRCIV ETEILAFGYY IYGWRQVPIN VDIIGEKANA T RPEIEQII ...文字列: MTTELNQGEQ FVADFRANAA ALTTANAYNP EDEHDACGVG FIAAIDGKPR RSVVEKGIEA LKAVWHRGAV DADGKTGDGA GIHVAVPQK FFKDHVKVIG HRAPDNKLAV GQVFLPRISL DAQEACRCIV ETEILAFGYY IYGWRQVPIN VDIIGEKANA T RPEIEQII VGNNKGVSDE QFELDLYIIR RRIEKAVKGE QINDFYICSL SARSIIYKGM FLAEQLTTFY PDLLDERFES DF AIYHQRY STNTFPTWPL AQPFRMLAHN GEINTVKGNV NWMKAHETRM EHPAFGTHMQ DLKPVIGVGL SDSGSLDTVF EVM VRAGRT APMVKMMLVP QALTSSQTTP DNHKALIQYC NSVMEPWDGP AALAMTDGRW VVGGMDRNGL RPMRYTITTD GLII GGSET GMVKIDETQV IEKGRLGPGE MIAVDLQSGK LYRDRELKDH LATLKPWDKW VQNTTHLDEL VKTASLKGEP SDMDK AELR RRQQAFGLTM EDMELILHPM VEDGKEAIGS MGDDSPIAVL SDKYRGLHHF FRQNFSQVTN PPIDSLRERR VMSLKT RLG NLGNILDEDE TQTRLLQLES PVLTTAEFRA MRDYMGDTAA EIDATFPVDG GPEALRDALR RIRQETEDAV RGGATHV IL TDEAMGPARA AIPAILATGA VHTHLIRSNL RTFTSLNVRT AEGLDTHYFA VLIGVGATTV NAYLAQEAIA ERHRRGLF G SMPLEKGMAN YKKAIDDGLL KIMSKMGISV ISSYRGGGNF EAIGLSRALV AEHFPAMVSR ISGIGLNGIQ KKVLEQHAT AYNEEVVALP VGGFYRFRKS GDRHGWEGGV IHTLQQAVTN DSYTTFKKYS EQVNKRPPMQ LRDLLELRST KAPVPVDEVE SITAIRKRF ITPGMSMGAL SPEAHGTLNV AMNRIGAKSD SGEGGEDPAR FRPDKNGDNW NSAIKQVASG RFGVTAEYLN Q CRELEIKV AQGAKPGEGG QLPGFKVTEM IARLRHSTPG VMLISPPPHH DIYSIEDLAQ LIYDLKQINP DAKVTVKLVS RS GIGTIAA GVAKANADII LISGNSGGTG ASPQTSIKFA GLPWEMGLSE VHQVLTLNRL RHRVRLRTDG GLKTGRDIVI AAM LGAEEF GIGTASLIAM GCIMVRQCHS NTCPVGVCVQ DDKLRQKFVG TPEKVVNLFT FLAEEVREIL AGLGFRSLNE VIGR TDLLH QVSRGAEHLD DLDLNPRLAQ VDPGENARYC TLQGRNEVPD TLDARIVADA RPLFEEGEKM QLAYNARNTQ RAIGT RLSS MVTRKFGMFG LQPGHITIRL RGTAGQSLGA FAVQGIKLEV MGDANDYVGK GLSGGTIVVR PTTSSPLETN KNTIIG NTV LYGATAGKLF AAGQAGERFA VRNSGATVVV EGCGSNGCEY MTGGTAVILG RVGDNFAAGM TGGMAYVYDL DDSLPLY IN DESVIFQRIE VGHYESQLKH LIEEHVTETQ SRFAAEILND WAREVTKFWQ VVPKEMLNRL EVPVHLPKAI SAE UniProtKB: Glutamate synthase [NADPH] large chain |
-分子 #2: Glutamate synthase [NADPH] small chain
| 分子 | 名称: Glutamate synthase [NADPH] small chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 4 / 光学異性体: LEVO / EC番号: glutamate synthase (NADPH) |
|---|---|
| 由来(天然) | 生物種:  Azospirillum brasilense (バクテリア) Azospirillum brasilense (バクテリア) |
| 分子量 | 理論値: 52.425109 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MANQRMLGFV HTAQRMPDKR PAAERRQDFA EIYARFSDER ANEQANRCSQ CGVPFCQVHC PVSNNIPDWL KLTSEGRLEE AYEVSQATN NFPEICGRIC PQDRLCEGNC VIEQSTHGAV TIGSVEKYIN DTAWDQGWVK PRTPSRELGL SVGVIGAGPA G LAAAEELR ...文字列: MANQRMLGFV HTAQRMPDKR PAAERRQDFA EIYARFSDER ANEQANRCSQ CGVPFCQVHC PVSNNIPDWL KLTSEGRLEE AYEVSQATN NFPEICGRIC PQDRLCEGNC VIEQSTHGAV TIGSVEKYIN DTAWDQGWVK PRTPSRELGL SVGVIGAGPA G LAAAEELR AKGYEVHVYD RYDRMGGLLV YGIPGFKLEK SVVERRVKLL ADAGVIYHPN FEVGRDASLP ELRRKHVAVL VA TGVYKAR DIKAPGSGLG NIVAALDYLT TSNKVSLGDT VEAYENGSLN AAGKHVVVLG GGDTAMDCVR TAIRQGATSV KCL YRRDRK NMPGSQREVA HAEEEGVEFI WQAAPEGFTG DTVVTGVRAV RIHLGVADAT GRQTPQVIEG SEFTVQADLV IKAL GFEPE DLPNAFDEPE LKVTRWGTLL VDHRTKMTNM DGVFAAGDIV RGASLVVWAI RDGRDAAEGI HAYAKAKAEA PVAVA AE UniProtKB: Glutamate synthase [NADPH] small chain |
-分子 #3: FLAVIN MONONUCLEOTIDE
| 分子 | 名称: FLAVIN MONONUCLEOTIDE / タイプ: ligand / ID: 3 / コピー数: 4 / 式: FMN |
|---|---|
| 分子量 | 理論値: 456.344 Da |
| Chemical component information |  ChemComp-FMN: |
-分子 #4: FE3-S4 CLUSTER
| 分子 | 名称: FE3-S4 CLUSTER / タイプ: ligand / ID: 4 / コピー数: 4 / 式: F3S |
|---|---|
| 分子量 | 理論値: 295.795 Da |
| Chemical component information |  ChemComp-F3S: |
-分子 #5: IRON/SULFUR CLUSTER
| 分子 | 名称: IRON/SULFUR CLUSTER / タイプ: ligand / ID: 5 / コピー数: 8 / 式: SF4 |
|---|---|
| 分子量 | 理論値: 351.64 Da |
| Chemical component information |  ChemComp-FS1: |
-分子 #6: FLAVIN-ADENINE DINUCLEOTIDE
| 分子 | 名称: FLAVIN-ADENINE DINUCLEOTIDE / タイプ: ligand / ID: 6 / コピー数: 4 / 式: FAD |
|---|---|
| 分子量 | 理論値: 785.55 Da |
| Chemical component information |  ChemComp-FAD: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)