+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0262 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | III2-IV2 mitochondrial respiratory supercomplex from S. cerevisiae | |||||||||
 マップデータ マップデータ | The sharpened map of the III2IV2 supercomplex | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報matrix side of mitochondrial inner membrane / protein processing involved in protein targeting to mitochondrion / mitochondrial cytochrome c oxidase assembly / mitochondrial respiratory chain complex III assembly / Respiratory electron transport / mitochondrial respirasome assembly / Mitochondrial protein degradation / : / : / cytochrome-c oxidase ...matrix side of mitochondrial inner membrane / protein processing involved in protein targeting to mitochondrion / mitochondrial cytochrome c oxidase assembly / mitochondrial respiratory chain complex III assembly / Respiratory electron transport / mitochondrial respirasome assembly / Mitochondrial protein degradation / : / : / cytochrome-c oxidase / : / quinol-cytochrome-c reductase / cellular respiration / ubiquinol-cytochrome-c reductase activity / mitochondrial electron transport, cytochrome c to oxygen / cytochrome-c oxidase activity / mitochondrial electron transport, ubiquinol to cytochrome c / mitochondrial crista / electron transport coupled proton transport / enzyme regulator activity / ATP synthesis coupled electron transport / proton transmembrane transport / nuclear periphery / mitochondrial membrane / aerobic respiration / mitochondrial intermembrane space / metalloendopeptidase activity / 2 iron, 2 sulfur cluster binding / oxidoreductase activity / mitochondrial inner membrane / copper ion binding / heme binding / mitochondrion / proteolysis / zinc ion binding / membrane / metal ion binding / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
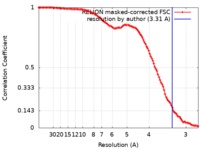
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.31 Å | |||||||||
 データ登録者 データ登録者 | Hartley AM / Lukoyanova N / Pinotsis N / Marechal A | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2019 ジャーナル: Nat Struct Mol Biol / 年: 2019タイトル: Structure of yeast cytochrome c oxidase in a supercomplex with cytochrome bc. 著者: Andrew M Hartley / Natalya Lukoyanova / Yunyi Zhang / Alfredo Cabrera-Orefice / Susanne Arnold / Brigitte Meunier / Nikos Pinotsis / Amandine Maréchal /    要旨: Cytochrome c oxidase (complex IV, CIV) is known in mammals to exist independently or in association with other respiratory proteins to form supercomplexes (SCs). In Saccharomyces cerevisiae, CIV is ...Cytochrome c oxidase (complex IV, CIV) is known in mammals to exist independently or in association with other respiratory proteins to form supercomplexes (SCs). In Saccharomyces cerevisiae, CIV is found solely in an SC with cytochrome bc (complex III, CIII). Here, we present the cryogenic electron microscopy (cryo-EM) structure of S. cerevisiae CIV in a IIIIV SC at 3.3 Å resolution. While overall similarity to mammalian homologs is high, we found notable differences in the supernumerary subunits Cox26 and Cox13; the latter exhibits a unique arrangement that precludes CIV dimerization as seen in bovine. A conformational shift in the matrix domain of Cox5A-involved in allosteric inhibition by ATP-may arise from its association with CIII. The CIII-CIV arrangement highlights a conserved interaction interface of CIII, albeit one occupied by complex I in mammalian respirasomes. We discuss our findings in the context of the potential impact of SC formation on CIV regulation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0262.map.gz emd_0262.map.gz | 106.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0262-v30.xml emd-0262-v30.xml emd-0262.xml emd-0262.xml | 30.6 KB 30.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0262_fsc.xml emd_0262_fsc.xml | 11 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0262.png emd_0262.png | 147.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0262 http://ftp.pdbj.org/pub/emdb/structures/EMD-0262 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0262 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0262 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_0262_validation.pdf.gz emd_0262_validation.pdf.gz | 335.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_0262_full_validation.pdf.gz emd_0262_full_validation.pdf.gz | 334.8 KB | 表示 | |
| XML形式データ |  emd_0262_validation.xml.gz emd_0262_validation.xml.gz | 11.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0262 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0262 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0262 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0262 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0262.map.gz / 形式: CCP4 / 大きさ: 113.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0262.map.gz / 形式: CCP4 / 大きさ: 113.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The sharpened map of the III2IV2 supercomplex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.3861 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : III2-IV2 mitochondrial respiratory supercomplex
+超分子 #1: III2-IV2 mitochondrial respiratory supercomplex
+超分子 #2: 10-subunit yeast cytochrome bc1
+超分子 #3: 12-subunit cytochrome c oxidase with isoform Cox5A
+分子 #1: Cytochrome b-c1 complex subunit 1, mitochondrial
+分子 #2: Cytochrome b-c1 complex subunit 2, mitochondrial
+分子 #3: Cytochrome b
+分子 #4: Cytochrome c1, heme protein, mitochondrial
+分子 #5: Cytochrome b-c1 complex subunit Rieske, mitochondrial
+分子 #6: Cytochrome b-c1 complex subunit 6
+分子 #7: Cytochrome b-c1 complex subunit 7
+分子 #8: Cytochrome b-c1 complex subunit 8
+分子 #9: Cytochrome b-c1 complex subunit 9
+分子 #10: Cytochrome b-c1 complex subunit 10
+分子 #11: Cox1
+分子 #12: Cox2
+分子 #13: Cox3
+分子 #14: Cox4
+分子 #15: Cox5A
+分子 #16: Cox6
+分子 #17: Cox7
+分子 #18: Cox8
+分子 #19: Cox9
+分子 #20: Cox12
+分子 #21: Cox13
+分子 #22: Cox26
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.2 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 4.0 kPa |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 92 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV 詳細: 3 uL of sample applied to negatively glow discharged grid, blot force -10; blotting time 8.5 sec. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - サンプリング間隔: 5.0 µm / 平均露光時間: 8.0 sec. / 平均電子線量: 1.645 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 倍率(公称値): 130000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー