+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9397 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
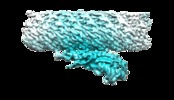
| タイトル | F-pilus/MS2 Maturation protein complex | |||||||||
 マップデータ マップデータ | Focused refinement of Escherichia coli F-pilus and Enterobacteria phage MS2 maturation protein. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | MS2 maturation protein / F-pilus / adsorption complex /  PROTEIN BINDING (タンパク質) PROTEIN BINDING (タンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral genome circularization /  : / virion attachment to host cell pilus / : / virion attachment to host cell pilus /  virion component / membrane => GO:0016020 / symbiont entry into host cell / virion component / membrane => GO:0016020 / symbiont entry into host cell /  RNA binding / extracellular region RNA binding / extracellular region類似検索 - 分子機能 | |||||||||
| 生物種 |   Escherichia coli (大腸菌) / Escherichia coli (大腸菌) /   Enterobacteria phage MS2 (ファージ) Enterobacteria phage MS2 (ファージ) | |||||||||
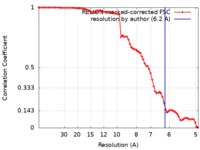
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 6.2 Å クライオ電子顕微鏡法 / 解像度: 6.2 Å | |||||||||
 データ登録者 データ登録者 | Meng R / Jiang M / Zhang J | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2019 ジャーナル: Nat Commun / 年: 2019タイトル: Structural basis for the adsorption of a single-stranded RNA bacteriophage. 著者: Ran Meng / Mengqiu Jiang / Zhicheng Cui / Jeng-Yih Chang / Kailu Yang / Joanita Jakana / Xinzhe Yu / Zhao Wang / Bo Hu / Junjie Zhang /  要旨: Single-stranded RNA bacteriophages (ssRNA phages) infect Gram-negative bacteria via a single maturation protein (Mat), which attaches to a retractile pilus of the host. Here we present structures of ...Single-stranded RNA bacteriophages (ssRNA phages) infect Gram-negative bacteria via a single maturation protein (Mat), which attaches to a retractile pilus of the host. Here we present structures of the ssRNA phage MS2 in complex with the Escherichia coli F-pilus, showing a network of hydrophobic and electrostatic interactions at the Mat-pilus interface. Moreover, binding of the pilus induces slight orientational variations of the Mat relative to the rest of the phage capsid, priming the Mat-connected genomic RNA (gRNA) for its release from the virions. The exposed tip of the attached Mat points opposite to the direction of the pilus retraction, which may facilitate the translocation of the gRNA from the capsid into the host cytosol. In addition, our structures determine the orientation of the assembled F-pilin subunits relative to the cell envelope, providing insights into the F-like type IV secretion systems. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9397.map.gz emd_9397.map.gz | 1.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9397-v30.xml emd-9397-v30.xml emd-9397.xml emd-9397.xml | 13.4 KB 13.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_9397_fsc.xml emd_9397_fsc.xml | 7.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_9397.png emd_9397.png | 237.3 KB | ||
| Filedesc metadata |  emd-9397.cif.gz emd-9397.cif.gz | 5.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9397 http://ftp.pdbj.org/pub/emdb/structures/EMD-9397 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9397 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9397 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9397.map.gz / 形式: CCP4 / 大きさ: 38.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9397.map.gz / 形式: CCP4 / 大きさ: 38.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focused refinement of Escherichia coli F-pilus and Enterobacteria phage MS2 maturation protein. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.46 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : F-pilus/MS2 maturation protein complex
| 全体 | 名称: F-pilus/MS2 maturation protein complex |
|---|---|
| 要素 |
|
-超分子 #1: F-pilus/MS2 maturation protein complex
| 超分子 | 名称: F-pilus/MS2 maturation protein complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 / 詳細: F-pilus/MS2 Maturation protein complex |
|---|
-超分子 #2: Enterobacteria phage MS2 maturation protein
| 超分子 | 名称: Enterobacteria phage MS2 maturation protein / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-超分子 #3: F-pilus
| 超分子 | 名称: F-pilus / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Enterobacteria phage MS2 (ファージ) Enterobacteria phage MS2 (ファージ) |
-分子 #1: Type IV conjugative transfer system pilin TraA
| 分子 | 名称: Type IV conjugative transfer system pilin TraA / タイプ: protein_or_peptide / ID: 1 / コピー数: 75 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 分子量 | 理論値: 6.831216 KDa |
| 配列 | 文字列: QDLMASGNTT VKATFGKDSS VVKWVVLAEV LVGAVMYMMT KNVKFLAGFA IISVFIAVGM AVVGL UniProtKB: Type IV conjugative transfer system pilin TraA |
-分子 #2: Maturation protein
| 分子 | 名称: Maturation protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Enterobacteria phage MS2 (ファージ) Enterobacteria phage MS2 (ファージ) |
| 分子量 | 理論値: 44.030934 KDa |
| 配列 | 文字列: MRAFSTLDRE NETFVPSVRV YADGETEDNS FSLKYRSNWT PGRFNSTGAK TKQWHYPSPY SRGALSVTSI DQGAYKRSGS SWGRPYEEK AGFGFSLDAR SCYSLFPVSQ NLTYIEVPQN VANRASTEVL QKVTQGNFNL GVALAEARST ASQLATQTIA L VKAYTAAR ...文字列: MRAFSTLDRE NETFVPSVRV YADGETEDNS FSLKYRSNWT PGRFNSTGAK TKQWHYPSPY SRGALSVTSI DQGAYKRSGS SWGRPYEEK AGFGFSLDAR SCYSLFPVSQ NLTYIEVPQN VANRASTEVL QKVTQGNFNL GVALAEARST ASQLATQTIA L VKAYTAAR RGNWRQALRY LALNEDRKFR SKHVAGRWLE LQFGWLPLMS DIQGAYEMLT KVHLQEFLPM RAVRQVGTNI KL DGRLSYP AANFQTTCNI SRRIVIWFYI NDARLAWLSS LGILNPLGIV WEKVPFSFVV DWLLPVGNML EGLTAPVGCS YMS GTVTDV ITGESIISVD APYGWTVERQ GTAKAQISAM HRGVQSVWPT TGAYVKSPFS MVHTLDALAL IRQRLSR UniProtKB: Maturation protein A |
-分子 #3: (2R)-2,3-dihydroxypropyl ethyl hydrogen (S)-phosphate
| 分子 | 名称: (2R)-2,3-dihydroxypropyl ethyl hydrogen (S)-phosphate タイプ: ligand / ID: 3 / コピー数: 70 / 式: KSV |
|---|---|
| 分子量 | 理論値: 200.127 Da |
| Chemical component information | 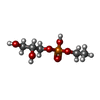 ChemComp-KSV: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 400 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 3200FSC |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 特殊光学系 | エネルギーフィルター - スリット幅: 30 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均露光時間: 8.0 sec. / 平均電子線量: 37.0 e/Å2 |
 ムービー
ムービー コントローラー
コントローラー