+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | cryo-EM structure of TRPM3 ion channel in the absence of PIP2 | |||||||||
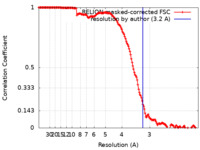
 マップデータ マップデータ | sharpened map with a B-factor of 13.2 | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報monoatomic cation transmembrane transport / monoatomic cation transport / monoatomic cation channel activity / protein tetramerization / calcium ion transmembrane transport /  calcium channel activity / calcium channel activity /  生体膜 生体膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.2 Å クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Zhao C / MacKinnon R | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Neuron / 年: 2023 ジャーナル: Neuron / 年: 2023タイトル: Structural and functional analyses of a GPCR-inhibited ion channel TRPM3. 著者: Chen Zhao / Roderick MacKinnon /  要旨: G-protein coupled receptors (GPCRs) govern the physiological response to stimuli by modulating the activity of downstream effectors, including ion channels. TRPM3 is an ion channel inhibited by GPCRs ...G-protein coupled receptors (GPCRs) govern the physiological response to stimuli by modulating the activity of downstream effectors, including ion channels. TRPM3 is an ion channel inhibited by GPCRs through direct interaction with G protein (Gβγ) released upon their activation. This GPCR-TRPM3 signaling pathway contributes to the analgesic effect of morphine. Here, we characterized Gβγ inhibition of TRPM3 using electrophysiology and single particle cryo-electron microscopy (cryo-EM). From electrophysiology, we obtained a half inhibition constant (IC50) of ∼240 nM. Using cryo-EM, we determined structures of mouse TRPM3 expressed in human cells with and without Gβγ and with and without PIP, a lipid required for TRPM3 activity, at resolutions of 2.7-4.7 Å. Gβγ-TRPM3 interfaces vary depending on PIP occupancy; however, in all cases, Gβγ appears loosely attached to TRPM3. The IC50 in electrophysiology experiments raises the possibility that additional unknown factors may stabilize the TRPM3-Gβγ complex. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27339.map.gz emd_27339.map.gz | 14.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27339-v30.xml emd-27339-v30.xml emd-27339.xml emd-27339.xml | 16.3 KB 16.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27339_fsc.xml emd_27339_fsc.xml | 15.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27339.png emd_27339.png | 173.4 KB | ||
| その他 |  emd_27339_half_map_1.map.gz emd_27339_half_map_1.map.gz emd_27339_half_map_2.map.gz emd_27339_half_map_2.map.gz | 254.4 MB 254.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27339 http://ftp.pdbj.org/pub/emdb/structures/EMD-27339 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27339 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27339 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8ddrMC  8ddqC  8ddsC  8ddtC  8dduC  8ddvC  8ddwC  8ddxC  8ed7C  8ed8C  8ed9C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27339.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27339.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map with a B-factor of 13.2 | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.046 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half map 1
| ファイル | emd_27339_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_27339_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : TRPM3
| 全体 | 名称: TRPM3 |
|---|---|
| 要素 |
|
-超分子 #1: TRPM3
| 超分子 | 名称: TRPM3 / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
-分子 #1: Transient receptor potential cation channel, subfamily M, member 3
| 分子 | 名称: Transient receptor potential cation channel, subfamily M, member 3 タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 分子量 | 理論値: 154.649328 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GKKWRDAGEL ERGCSDREDS AESRRRSRSA SRGRFAESWK RLSSKQGSTK RSGLPAQQTP AQKSWIERAF YKRECVHIIP STKDPHRCC CGRLIGQHVG LTPSISVLQN EKNESRLSRN DIQSEKWSIS KHTQLSPTDA FGTIEFQGGG HSNKAMYVRV S FDTKPDLL ...文字列: GKKWRDAGEL ERGCSDREDS AESRRRSRSA SRGRFAESWK RLSSKQGSTK RSGLPAQQTP AQKSWIERAF YKRECVHIIP STKDPHRCC CGRLIGQHVG LTPSISVLQN EKNESRLSRN DIQSEKWSIS KHTQLSPTDA FGTIEFQGGG HSNKAMYVRV S FDTKPDLL LHLMTKEWQL ELPKLLISVH GGLQNFELQP KLKQVFGKGL IKAAMTTGAW IFTGGVNTGV IRHVGDALKD HA SKSRGKI CTIGIAPWGI VENQEDLIGR DVVRPYQTMS NPMSKLTVLN SMHSHFILAD NGTTGKYGAE VKLRRQLEKH ISL QKINTR IGQGVPVVAL IVEGGPNVIS IVLEYLRDTP PVPVVVCDGS GRASDILAFG HKYSEEGGLI NESLRDQLLV TIQK TFTYT RTQAQHLFII LMECMKKKEL ITVFRMGSEG HQDIDLAILT ALLKGANASA PDQLSLALAW NRVDIARSQI FIYGQ QWPV GSLEQAMLDA LVLDRVDFVK LLIENGVSMH RFLTISRLEE LYNTRHGPSN TLYHLVRDVK KGNLPPDYRI SLIDIG LVI EYLMGGAYRC NYTRKRFRTL YHNLFGPKRP KALKLLGMED DIPLRRGRKT TKKREEEVDI DLDDPEINHF PFPFHEL MV WAVLMKRQKM ALFFWQHGEE AMAKALVACK LCKAMAHEAS ENDMVDDISQ ELNHNSRDFG QLAVELLDQS YKQDEQLA M KLLTYELKNW SNATCLQLAV AAKHRDFIAH TCSQMLLTDM WMGRLRMRKN SGLKVILGIL LPPSILSLEF KNKDDMPYM TQAQEIHLQE KEPEEPEKPT KEKDEEDMEL TAMLGRSNGE SSRKKDEEEV QSRHRLIPVG RKIYEFYNAP IVKFWFYTLA YIGYLMLFN YIVLVKMERW PSTQEWIVIS YIFTLGIEKM REILMSEPGK LLQKVKVWLQ EYWNVTDLIA ILLFSVGMIL R LQDQPFRS DGRVIYCVNI IYWYIRLLDI FGVNKYLGPY VMMIGKMMID MMYFVIIMLV VLMSFGVARQ AILFPNEEPS WK LAKNIFY MPYWMIYGEV FADQIDPPCG QNETREDGKT IQLPPCKTGA WIVPAIMACY LLVANILLVN LLIAVFNNTF FEV KSISNQ VWKFQRYQLI MTFHERPVLP PPLIIFSHMT MIFQHVCCRW RKHESDQDER DYGLKLFITD DELKKVHDFE EQCI EEYFR EKDDRFNSSN DERIRVTSER VENMSMRLEE VNEREHSMKA SLQTVDIRLA QLEDLIGRMA TALERLTGLE RAESN KIRS RTSSDCTDAA YIVRQSSFNS QEGNTFKLQE SIDPAGEETI SPTSPTLMPR MRSHSFYSV |
-分子 #2: Unidentified segment at the N-terminus of TRPM3
| 分子 | 名称: Unidentified segment at the N-terminus of TRPM3 / タイプ: protein_or_peptide / ID: 2 詳細: a segment at the N-terminus of TRPM3 whose sequence cannot be identified from the cryo-EM density コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 分子量 | 理論値: 1.464797 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK) |
-分子 #3: 1,2-DIACYL-GLYCEROL-3-SN-PHOSPHATE
| 分子 | 名称: 1,2-DIACYL-GLYCEROL-3-SN-PHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 4 / 式: 3PH |
|---|---|
| 分子量 | 理論値: 704.998 Da |
| Chemical component information |  ChemComp-3PH: |
-分子 #4: (3beta,14beta,17beta,25R)-3-[4-methoxy-3-(methoxymethyl)butoxy]sp...
| 分子 | 名称: (3beta,14beta,17beta,25R)-3-[4-methoxy-3-(methoxymethyl)butoxy]spirost-5-en タイプ: ligand / ID: 4 / コピー数: 4 / 式: 9Z9 |
|---|---|
| 分子量 | 理論値: 544.805 Da |
| Chemical component information |  ChemComp-9Z9: |
-分子 #5: SODIUM ION
| 分子 | 名称: SODIUM ION / タイプ: ligand / ID: 5 / コピー数: 2 |
|---|---|
| 分子量 | 理論値: 22.99 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K2 IS (4k x 4k) / 平均電子線量: 73.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z
Z Y
Y X
X