+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23048 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human Factor V at 3.3 Angstrom resolution | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報response to vitamin K /  Α顆粒 / Cargo concentration in the ER / Α顆粒 / Cargo concentration in the ER /  循環器 / COPII-mediated vesicle transport / COPII-coated ER to Golgi transport vesicle / Common Pathway of Fibrin Clot Formation / endoplasmic reticulum-Golgi intermediate compartment membrane / platelet alpha granule lumen / 循環器 / COPII-mediated vesicle transport / COPII-coated ER to Golgi transport vesicle / Common Pathway of Fibrin Clot Formation / endoplasmic reticulum-Golgi intermediate compartment membrane / platelet alpha granule lumen /  Post-translational protein phosphorylation ...response to vitamin K / Post-translational protein phosphorylation ...response to vitamin K /  Α顆粒 / Cargo concentration in the ER / Α顆粒 / Cargo concentration in the ER /  循環器 / COPII-mediated vesicle transport / COPII-coated ER to Golgi transport vesicle / Common Pathway of Fibrin Clot Formation / endoplasmic reticulum-Golgi intermediate compartment membrane / platelet alpha granule lumen / 循環器 / COPII-mediated vesicle transport / COPII-coated ER to Golgi transport vesicle / Common Pathway of Fibrin Clot Formation / endoplasmic reticulum-Golgi intermediate compartment membrane / platelet alpha granule lumen /  Post-translational protein phosphorylation / Regulation of Insulin-like Growth Factor (IGF) transport and uptake by Insulin-like Growth Factor Binding Proteins (IGFBPs) / Post-translational protein phosphorylation / Regulation of Insulin-like Growth Factor (IGF) transport and uptake by Insulin-like Growth Factor Binding Proteins (IGFBPs) /  extracellular vesicle / extracellular vesicle /  凝固・線溶系 / Platelet degranulation / copper ion binding / 凝固・線溶系 / Platelet degranulation / copper ion binding /  小胞体 / 小胞体 /  extracellular space / extracellular region / extracellular space / extracellular region /  生体膜 / 生体膜 /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) / Homo sapiens (ヒト) /   Human (ヒト) Human (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.3 Å クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Ruben EA / Di Cera E | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Blood / 年: 2021 ジャーナル: Blood / 年: 2021タイトル: Cryo-EM structures of human coagulation factors V and Va. 著者: Eliza A Ruben / Michael J Rau / James A J Fitzpatrick / Enrico Di Cera /  要旨: Coagulation factor V (fV) is the precursor of fVa, which, together with fXa, Ca2+, and phospholipids, defines the prothrombinase complex and activates prothrombin in the penultimate step of the ...Coagulation factor V (fV) is the precursor of fVa, which, together with fXa, Ca2+, and phospholipids, defines the prothrombinase complex and activates prothrombin in the penultimate step of the coagulation cascade. We solved the cryogenic electron microscopy (cryo-EM) structures of human fV and fVa at atomic (3.3 Å) and near-atomic (4.4 Å) resolution, respectively. The structure of fV reveals the entire A1-A2-B-A3-C1-C2 assembly, but with a surprisingly disordered B domain. The C1 and C2 domains provide a platform for interaction with phospholipid membranes and support the A1 and A3 domains, with the A2 domain sitting on top of them. The B domain is highly dynamic and visible only for short segments connecting to the A2 and A3 domains. The A2 domain reveals all sites of proteolytic processing by thrombin and activated protein C, a partially buried epitope for binding fXa, and fully exposed epitopes for binding activated protein C and prothrombin. Removal of the B domain and activation to fVa exposes the sites of cleavage by activated protein C at R306 and R506 and produces increased disorder in the A1-A2-A3-C1-C2 assembly, especially in the C-terminal acidic portion of the A2 domain that is responsible for prothrombin binding. Ordering of this region and full exposure of the fXa epitope emerge as necessary steps in the assembly of the prothrombin-prothrombinase complex. These structures offer molecular context for the function of fV and fVa and pioneer the analysis of coagulation factors by cryo-EM. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23048.map.gz emd_23048.map.gz | 93.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23048-v30.xml emd-23048-v30.xml emd-23048.xml emd-23048.xml | 14.8 KB 14.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23048.png emd_23048.png | 44.6 KB | ||
| マスクデータ |  emd_23048_msk_1.map emd_23048_msk_1.map | 98.9 MB |  マスクマップ マスクマップ | |
| その他 |  emd_23048_half_map_1.map.gz emd_23048_half_map_1.map.gz emd_23048_half_map_2.map.gz emd_23048_half_map_2.map.gz | 91.7 MB 91.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23048 http://ftp.pdbj.org/pub/emdb/structures/EMD-23048 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23048 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23048 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23048.map.gz / 形式: CCP4 / 大きさ: 98.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23048.map.gz / 形式: CCP4 / 大きさ: 98.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_23048_msk_1.map emd_23048_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
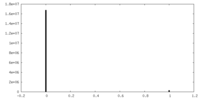
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_23048_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
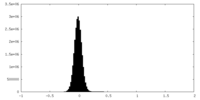
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_23048_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
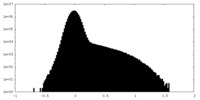
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : human Factor V
| 全体 | 名称: human Factor V |
|---|---|
| 要素 |
|
-超分子 #1: human Factor V
| 超分子 | 名称: human Factor V / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Coagulation factor V
| 分子 | 名称: Coagulation factor V / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human (ヒト) Human (ヒト) |
| 分子量 | 理論値: 248.973234 KDa |
| 配列 | 文字列: AQLRQFYVAA QGISWSYRPE PTNSSLNLSV TSFKKIVYRE YEPYFKKEKP QSTISGLLGP TLYAEVGDII KVHFKNKADK PLSIHPQGI RYSKLSEGAS YLDHTFPAEK MDDAVAPGRE YTYEWSISED SGPTHDDPPC LTHIYYSHEN LIEDFNSGLI G PLLICKKG ...文字列: AQLRQFYVAA QGISWSYRPE PTNSSLNLSV TSFKKIVYRE YEPYFKKEKP QSTISGLLGP TLYAEVGDII KVHFKNKADK PLSIHPQGI RYSKLSEGAS YLDHTFPAEK MDDAVAPGRE YTYEWSISED SGPTHDDPPC LTHIYYSHEN LIEDFNSGLI G PLLICKKG TLTEGGTQKT FDKQIVLLFA VFDESKSWSQ SSSLMYTVNG YVNGTMPDIT VCAHDHISWH LLGMSSGPEL FS IHFNGQV LEQNHHKVSA ITLVSATSTT ANMTVGPEGK WIISSLTPKH LQAGMQAYID IKNCPKKTRN LKKITREQRR HMK RWEYFI AAEEVIWDYA PVIPANMDKK YRSQHLDNFS NQIGKHYKKV MYTQYEDESF TKHTVNPNMK EDGILGPIIR AQVR DTLKI VFKNMASRPY SIYPHGVTFS PYEDEVNSSF TSGRNNTMIR AVQPGETYTY KWNILEFDEP TENDAQCLTR PYYSD VDIM RDIASGLIGL LLICKSRSLD RRGIQRAADI EQQAVFAVFD ENKSWYLEDN INKFCENPDE VKRDDPKFYE SNIMST ING YVPESITTLG FCFDDTVQWH FCSVGTQNEI LTIHFTGHSF IYGKRHEDTL TLFPMRGESV TVTMDNVGTW MLTSMNS SP RSKKLRLKFR DVKCIPDDDE DSYEIFEPPE STVMATRKMH DRLEPEDEES DADYDYQNRL AAALGIRSFR NSSLNQEE E EFNLTALALE NGTEFVSSNT DIIVGSNYSS PSNISKFTVN NLAEPQKAPS HQQATTAGSP LRHLIGKNSV LNSSTAEHS SPYSEDPIED PLQPDVTGIR LLSLGAGEFK SQEHAKHKGP KVERDQAAKH RFSWMKLLAH KVGRHLSQDT GSPSGMRPWE DLPSQDTGS PSRMRPWKDP PSDLLLLKQS NSSKILVGRW HLASEKGSYE IIQDTDEDTA VNNWLISPQN ASRAWGESTP L ANKPGKQS GHPKFPRVRH KSLQVRQDGG KSRLKKSQFL IKTRKKKKEK HTHHAPLSPR TFHPLRSEAY NTFSERRLKH SL VLHKSNE TSLPTDLNQT LPSMDFGWIA SLPDHNQNSS NDTGQASCPP GLYQTVPPEE HYQTFPIQDP DQMHSTSDPS HRS SSPELS EMLEYDRSHK SFPTDISQMS PSSEHEVWQT VISPDLSQVT LSPELSQTNL SPDLSHTTLS PELIQRNLSP ALGQ MPISP DLSHTTLSPD LSHTTLSLDL SQTNLSPELS QTNLSPALGQ MPLSPDLSHT TLSLDFSQTN LSPELSHMTL SPELS QTNL SPALGQMPIS PDLSHTTLSL DFSQTNLSPE LSQTNLSPAL GQMPLSPDPS HTTLSLDLSQ TNLSPELSQT NLSPDL SEM PLFADLSQIP LTPDLDQMTL SPDLGETDLS PNFGQMSLSP DLSQVTLSPD ISDTTLLPDL SQISPPPDLD QIFYPSE SS QSLLLQEFNE SFPYPDLGQM PSPSSPTLND TFLSKEFNPL VIVGLSKDGT DYIEIIPKEE VQSSEDDYAE IDYVPYDD P YKTDVRTNIN SSRDPDNIAA WYLRSNNGNR RNYYIAAEEI SWDYSEFVQR ETDIEDSDDI PEDTTYKKVV FRKYLDSTF TKRDPRGEYE EHLGILGPII RAEVDDVIQV RFKNLASRPY SLHAHGLSYE KSSEGKTYED DSPEWFKEDN AVQPNSSYTY VWHATERSG PESPGSACRA WAYYSAVNPE KDIHSGLIGP LLICQKGILH KDSNMPMDMR EFVLLFMTFD EKKSWYYEKK S RSSWRLTS SEMKKSHEFH AINGMIYSLP GLKMYEQEWV RLHLLNIGGS QDIHVVHFHG QTLLENGNKQ HQLGVWPLLP GS FKTLEMK ASKPGWWLLN TEVGENQRAG MQTPFLIMDR DCRMPMGLST GIISDSQIKA SEFLGYWEPR LARLNNGGSY NAW SVEKLA AEFASKPWIQ VDMQKEVIIT GIQTQGAKHY LKSCYTTEFY VAYSSNQINW QIFKGNSTRN VMYFNGNSDA STIK ENQFD PPIVARYIRI SPTRAYNRPT LRLELQGCEV NGCSTPLGME NGKIENKQIT ASSFKKSWWG DYWEPFRARL NAQGR VNAW QAKANNNKQW LEIDLLKIKK ITAIITQGCK SLSSEMYVKS YTIHYSEQGV EWKPYRLKSS MVDKIFEGNT NTKGHV KNF FNPPIISRFI RVIPKTWNQS IALRLELFGC DIY |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 詳細: 20 mM HEPES, 150 mM NaCl, 5 mM CaCl2 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 平均電子線量: 1.65 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
|---|---|
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 299182 |
-原子モデル構築 1
| 精密化 | プロトコル: OTHER |
|---|---|
| 得られたモデル |  PDB-7kve: |
 ムービー
ムービー コントローラー
コントローラー

























 Z
Z Y
Y X
X