+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of GroEL-nucleotide complex in ADP-like conformation plunged 50 ms after mixing with ATP | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード |  GroEL / GroEL /  GroES / GroES /  CHAPERONE (シャペロン) CHAPERONE (シャペロン) | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報GroEL-GroES complex /  chaperonin ATPase / chaperonin ATPase /  virion assembly / chaperone cofactor-dependent protein refolding / virion assembly / chaperone cofactor-dependent protein refolding /  isomerase activity / ATP-dependent protein folding chaperone / response to radiation / unfolded protein binding / isomerase activity / ATP-dependent protein folding chaperone / response to radiation / unfolded protein binding /  フォールディング / response to heat ...GroEL-GroES complex / フォールディング / response to heat ...GroEL-GroES complex /  chaperonin ATPase / chaperonin ATPase /  virion assembly / chaperone cofactor-dependent protein refolding / virion assembly / chaperone cofactor-dependent protein refolding /  isomerase activity / ATP-dependent protein folding chaperone / response to radiation / unfolded protein binding / isomerase activity / ATP-dependent protein folding chaperone / response to radiation / unfolded protein binding /  フォールディング / response to heat / protein refolding / magnesium ion binding / フォールディング / response to heat / protein refolding / magnesium ion binding /  ATP hydrolysis activity / ATP hydrolysis activity /  ATP binding / ATP binding /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  細胞質基質 細胞質基質類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Escherichia coli (大腸菌) Escherichia coli (大腸菌) | ||||||||||||
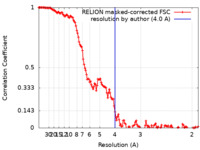
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.0 Å クライオ電子顕微鏡法 / 解像度: 4.0 Å | ||||||||||||
 データ登録者 データ登録者 | Dhurandhar M / Torino S / Efremov R | ||||||||||||
| 資金援助 | European Union,  ベルギー, 3件 ベルギー, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Methods / 年: 2023 ジャーナル: Nat Methods / 年: 2023タイトル: Time-resolved cryo-EM using a combination of droplet microfluidics with on-demand jetting. 著者: Stefania Torino / Mugdha Dhurandhar / Annelore Stroobants / Raf Claessens / Rouslan G Efremov /  要旨: Single-particle cryogenic electron microscopy (cryo-EM) allows reconstruction of high-resolution structures of proteins in different conformations. Protein function often involves transient ...Single-particle cryogenic electron microscopy (cryo-EM) allows reconstruction of high-resolution structures of proteins in different conformations. Protein function often involves transient functional conformations, which can be resolved using time-resolved cryo-EM (trEM). In trEM, reactions are arrested after a defined delay time by rapid vitrification of protein solution on the EM grid. Despite the increasing interest in trEM among the cryo-EM community, making trEM samples with a time resolution below 100 ms remains challenging. Here we report the design and the realization of a time-resolved cryo-plunger that combines a droplet-based microfluidic mixer with a laser-induced generator of microjets that allows rapid reaction initiation and plunge-freezing of cryo-EM grids. Using this approach, a time resolution of 5 ms was achieved and the protein density map was reconstructed to a resolution of 2.1 Å. trEM experiments on GroEL:GroES chaperonin complex resolved the kinetics of the complex formation and visualized putative short-lived conformations of GroEL-ATP complex. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16108.map.gz emd_16108.map.gz | 132.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16108-v30.xml emd-16108-v30.xml emd-16108.xml emd-16108.xml | 18.1 KB 18.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16108_fsc.xml emd_16108_fsc.xml | 13.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16108.png emd_16108.png | 94.5 KB | ||
| マスクデータ |  emd_16108_msk_1.map emd_16108_msk_1.map | 229.8 MB |  マスクマップ マスクマップ | |
| その他 |  emd_16108_half_map_1.map.gz emd_16108_half_map_1.map.gz emd_16108_half_map_2.map.gz emd_16108_half_map_2.map.gz | 182.5 MB 182.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16108 http://ftp.pdbj.org/pub/emdb/structures/EMD-16108 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16108 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16108 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8bleMC  8bk7C  8bk8C  8bk9C  8bkaC  8bkbC  8bkgC  8bkzC  8bl2C  8bl7C  8blcC  8bldC  8blfC  8blyC  8bm0C  8bm1C  8bmdC  8bmoC  8bmtC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16108.map.gz / 形式: CCP4 / 大きさ: 229.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16108.map.gz / 形式: CCP4 / 大きさ: 229.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.96 Å | ||||||||||||||||||||||||||||||||||||
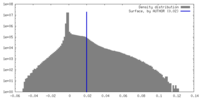
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_16108_msk_1.map emd_16108_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
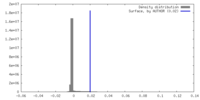
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_16108_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_16108_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Hetero 14mer assembled from 2 heptameric rings
| 全体 | 名称: Hetero 14mer assembled from 2 heptameric rings |
|---|---|
| 要素 |
|
-超分子 #1: Hetero 14mer assembled from 2 heptameric rings
| 超分子 | 名称: Hetero 14mer assembled from 2 heptameric rings / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 分子量 | 理論値: 800 KDa |
-分子 #1: Chaperonin GroEL
| 分子 | 名称: Chaperonin GroEL / タイプ: protein_or_peptide / ID: 1 / コピー数: 14 / 光学異性体: LEVO / EC番号:  chaperonin ATPase chaperonin ATPase |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) / 株: K12 Escherichia coli (大腸菌) / 株: K12 |
| 分子量 | 理論値: 57.391711 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MAAKDVKFGN DARVKMLRGV NVLADAVKVT LGPKGRNVVL DKSFGAPTIT KDGVSVAREI ELEDKFENMG AQMVKEVASK ANDAAGDGT TTATVLAQAI ITEGLKAVAA GMNPMDLKRG IDKAVTAAVE ELKALSVPCS DSKAIAQVGT ISANSDETVG K LIAEAMDK ...文字列: MAAKDVKFGN DARVKMLRGV NVLADAVKVT LGPKGRNVVL DKSFGAPTIT KDGVSVAREI ELEDKFENMG AQMVKEVASK ANDAAGDGT TTATVLAQAI ITEGLKAVAA GMNPMDLKRG IDKAVTAAVE ELKALSVPCS DSKAIAQVGT ISANSDETVG K LIAEAMDK VGKEGVITVE DGTGLQDELD VVEGMQFDRG YLSPYFINKP ETGAVELESP FILLADKKIS NIREMLPVLE AV AKAGKPL LIIAEDVEGE ALATLVVNTM RGIVKVAAVK APGFGDRRKA MLQDIATLTG GTVISEEIGM ELEKATLEDL GQA KRVVIN KDTTTIIDGV GEEAAIQGRV AQIRQQIEEA TSDYDREKLQ ERVAKLAGGV AVIKVGAATE VEMKEKKARV EDAL HATRA AVEEGVVAGG GVALIRVASK LADLRGQNED QNVGIKVALR AMEAPLRQIV LNCGEEPSVV ANTVKGGDGN YGYNA ATEE YGNMIDMGIL DPTKVTRSAL QYAASVAGLM ITTECMVTDL PKNDAADLGA AGGMGGMGGM GGMM UniProtKB: Chaperonin GroEL |
-分子 #2: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 14 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 8 mg/mL | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY | ||||||||||||||||
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 300 |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 0.5 µm Bright-field microscopy / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 試料ステージ | 試料ホルダーモデル: JEOL CRYOSPECPORTER / ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 63.6 e/Å2 |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-8ble: |
 ムービー
ムービー コントローラー
コントローラー

























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)