+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8xz3 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Mycobacterium smegmatis 50S ribosomal subunit with Erythromycin | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード | RIBOSOME / Antibiotic | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報large ribosomal subunit / transferase activity / 5S rRNA binding / ribosomal large subunit assembly / large ribosomal subunit rRNA binding / cytosolic large ribosomal subunit / cytoplasmic translation / tRNA binding / negative regulation of translation / rRNA binding ...large ribosomal subunit / transferase activity / 5S rRNA binding / ribosomal large subunit assembly / large ribosomal subunit rRNA binding / cytosolic large ribosomal subunit / cytoplasmic translation / tRNA binding / negative regulation of translation / rRNA binding / structural constituent of ribosome / ribosome / translation / ribonucleoprotein complex / mRNA binding / metal ion binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) | |||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Srinivasan, K. / Banerjee, A. / Sengupta, J. | |||||||||
| 資金援助 |  インド, 2件 インド, 2件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2024 ジャーナル: Structure / 年: 2024タイトル: Cryo-EM structures reveal the molecular mechanism of HflX-mediated erythromycin resistance in mycobacteria. 著者: Krishnamoorthi Srinivasan / Aneek Banerjee / Jayati Sengupta /  要旨: Mycobacterial HflX confers resistance against macrolide antibiotics. However, the exact molecular mechanism is poorly understood. To gain further insights, we determined the cryo-EM structures of M. ...Mycobacterial HflX confers resistance against macrolide antibiotics. However, the exact molecular mechanism is poorly understood. To gain further insights, we determined the cryo-EM structures of M. smegmatis (Msm) HflX-50S subunit and 50S subunit-erythromycin (ERY) complexes at a global resolution of approximately 3 Å. A conserved nucleotide A2286 at the gate of nascent peptide exit tunnel (NPET) adopts a swayed conformation in HflX-50S complex and interacts with a loop within the linker helical (LH) domain of MsmHflX that contains an additional 9 residues insertion. Interestingly, the swaying of this nucleotide, which is usually found in the non-swayed conformation, is induced by erythromycin binding. Furthermore, we observed that erythromycin decreases HflX's ribosome-dependent GTP hydrolysis, resulting in its enhanced binding and anti-association activity on the 50S subunit. Our findings reveal how mycobacterial HflX senses the presence of macrolides at the peptide tunnel entrance and confers antibiotic resistance in mycobacteria. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8xz3.cif.gz 8xz3.cif.gz | 2.1 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8xz3.ent.gz pdb8xz3.ent.gz | 1.6 MB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8xz3.json.gz 8xz3.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8xz3_validation.pdf.gz 8xz3_validation.pdf.gz | 1.7 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8xz3_full_validation.pdf.gz 8xz3_full_validation.pdf.gz | 1.9 MB | 表示 | |
| XML形式データ |  8xz3_validation.xml.gz 8xz3_validation.xml.gz | 166.5 KB | 表示 | |
| CIF形式データ |  8xz3_validation.cif.gz 8xz3_validation.cif.gz | 282 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/xz/8xz3 https://data.pdbj.org/pub/pdb/validation_reports/xz/8xz3 ftp://data.pdbj.org/pub/pdb/validation_reports/xz/8xz3 ftp://data.pdbj.org/pub/pdb/validation_reports/xz/8xz3 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  38788MC  8kabC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-50S ribosomal protein ... , 5種, 5分子 3HLQf
| #1: タンパク質・ペプチド | 分子量: 2710.179 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア)参照: UniProt: A0QTP4 |
|---|---|
| #9: タンパク質 | 分子量: 15949.253 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア)参照: UniProt: A0R7F6 |
| #13: タンパク質 | 分子量: 13400.625 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア)参照: UniProt: A0QSF9 |
| #18: タンパク質 | 分子量: 12892.806 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア)参照: UniProt: A0QV42 |
| #33: タンパク質・ペプチド | 分子量: 4341.277 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア)参照: UniProt: A0QSL4 |
-RNA鎖 , 2種, 2分子 AB
| #2: RNA鎖 | 分子量: 1011834.750 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア)参照: GenBank: 118168627 |
|---|---|
| #3: RNA鎖 | 分子量: 38061.816 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア)参照: GenBank: 118168627 |
+Large ribosomal subunit protein ... , 27種, 27分子 CDEFGIJKMNOPRSTUVWXYZabcdeg
-非ポリマー , 1種, 1分子 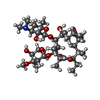
| #35: 化合物 | ChemComp-ERY / |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 |
| |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 実験値: NO | |||||||||||||||||||||
| 由来(天然) |
| |||||||||||||||||||||
| 由来(組換発現) |
| |||||||||||||||||||||
| 緩衝液 | pH: 7.5 | |||||||||||||||||||||
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES | |||||||||||||||||||||
| 急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3300 nm / 最小 デフォーカス(公称値): 1800 nm |
| 撮影 | 電子線照射量: 28.75 e/Å2 フィルム・検出器のモデル: FEI FALCON III (4k x 4k) |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||
| 3次元再構成 | 解像度: 3.6 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 43558 / 対称性のタイプ: POINT |
 ムービー
ムービー コントローラー
コントローラー




 PDBj
PDBj































