| 登録情報 | データベース: PDB / ID: 8x35
|
|---|
| タイトル | Neryl diphosphate synthase from Solanum lycopersicum complexed with DMSAPP, IPP, and magnesium ion (form A) |
|---|
 要素 要素 | Neryl diphosphate synthase 1 |
|---|
 キーワード キーワード | TRANSFERASE / cis-prenyltransferase |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
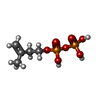
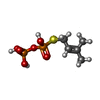
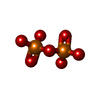
dimethylallylcistransferase / dimethylallylcistransferase activity / plastid membrane organization / polyprenol biosynthetic process / prenyltransferase activity / chloroplast stroma / response to cold / chloroplast / magnesium ion binding類似検索 - 分子機能 Di-trans-poly-cis-decaprenylcistransferase-like, conserved site / Undecaprenyl pyrophosphate synthase family signature. / Decaprenyl diphosphate synthase-like / Putative undecaprenyl diphosphate synthase / Decaprenyl diphosphate synthase-like superfamily類似検索 - ドメイン・相同性 DIPHOSPHATE / DIMETHYLALLYL S-THIOLODIPHOSPHATE / 3-METHYLBUT-3-ENYL TRIHYDROGEN DIPHOSPHATE / DI(HYDROXYETHYL)ETHER / Dimethylallylcistransferase CPT1, chloroplastic類似検索 - 構成要素 |
|---|
| 生物種 |   Solanum lycopersicum (トマト) Solanum lycopersicum (トマト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.92 Å 分子置換 / 解像度: 1.92 Å |
|---|
 データ登録者 データ登録者 | Imaizumi, R. / Matsuura, H. / Yanai, T. / Takeshita, K. / Misawa, S. / Yamaguchi, H. / Sakai, N. / Miyagi-Inoue, Y. / Suenaga-Hiromori, M. / Kataoka, K. ...Imaizumi, R. / Matsuura, H. / Yanai, T. / Takeshita, K. / Misawa, S. / Yamaguchi, H. / Sakai, N. / Miyagi-Inoue, Y. / Suenaga-Hiromori, M. / Kataoka, K. / Nakayama, T. / Yamamoto, M. / Takahashi, S. / Yamashita, S. |
|---|
| 資金援助 |  日本, 6件 日本, 6件 | 組織 | 認可番号 | 国 |
|---|
| Japan Society for the Promotion of Science (JSPS) | 23H05470 |  日本 日本 | | Japan Agency for Medical Research and Development (AMED) | JP20am0101070 |  日本 日本 | | Japan Society for the Promotion of Science (JSPS) | 22K19143 |  日本 日本 | | Japan Society for the Promotion of Science (JSPS) | 22KJ1466 |  日本 日本 | | Japan Society for the Promotion of Science (JSPS) | 20H02909 |  日本 日本 | | Japan Society for the Promotion of Science (JSPS) | 21H02115 |  日本 日本 |
|
|---|
 引用 引用 |  ジャーナル: Chembiochem / 年: 2024 ジャーナル: Chembiochem / 年: 2024
タイトル: Structural-Functional Correlations between Unique N-terminal Region and C-terminal Conserved Motif in Short-chain cis-Prenyltransferase from Tomato.
著者: Imaizumi, R. / Matsuura, H. / Yanai, T. / Takeshita, K. / Misawa, S. / Yamaguchi, H. / Sakai, N. / Miyagi-Inoue, Y. / Suenaga-Hiromori, M. / Waki, T. / Kataoka, K. / Nakayama, T. / Yamamoto, ...著者: Imaizumi, R. / Matsuura, H. / Yanai, T. / Takeshita, K. / Misawa, S. / Yamaguchi, H. / Sakai, N. / Miyagi-Inoue, Y. / Suenaga-Hiromori, M. / Waki, T. / Kataoka, K. / Nakayama, T. / Yamamoto, M. / Takahashi, S. / Yamashita, S. |
|---|
| 履歴 | | 登録 | 2023年11月12日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2024年2月7日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2024年4月17日 | Group: Database references / カテゴリ: citation
Item: _citation.journal_volume / _citation.page_first / _citation.page_last |
|---|
| 改定 1.2 | 2024年4月24日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.page_first / _citation.page_last / _citation_author.identifier_ORCID |
|---|
| 改定 1.3 | 2024年10月23日 | Group: Structure summary
カテゴリ: pdbx_entry_details / pdbx_modification_feature
Item: _pdbx_entry_details.has_protein_modification |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.92 Å
分子置換 / 解像度: 1.92 Å  データ登録者
データ登録者 日本, 6件
日本, 6件  引用
引用 ジャーナル: Chembiochem / 年: 2024
ジャーナル: Chembiochem / 年: 2024 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 8x35.cif.gz
8x35.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb8x35.ent.gz
pdb8x35.ent.gz PDB形式
PDB形式 8x35.json.gz
8x35.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 8x35_validation.pdf.gz
8x35_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 8x35_full_validation.pdf.gz
8x35_full_validation.pdf.gz 8x35_validation.xml.gz
8x35_validation.xml.gz 8x35_validation.cif.gz
8x35_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/x3/8x35
https://data.pdbj.org/pub/pdb/validation_reports/x3/8x35 ftp://data.pdbj.org/pub/pdb/validation_reports/x3/8x35
ftp://data.pdbj.org/pub/pdb/validation_reports/x3/8x35


 F&H 検索
F&H 検索 リンク
リンク 集合体
集合体
 要素
要素











 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SPring-8
SPring-8  / ビームライン: BL32XU / 波長: 1 Å
/ ビームライン: BL32XU / 波長: 1 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj






