+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8s7v | |||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
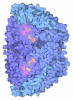
| タイトル | Methyl-coenzyme M reductase activation complex binding to the A2 component | |||||||||||||||||||||||||||||||||||||||||||||||||||
 要素 要素 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||
 キーワード キーワード | OXIDOREDUCTASE / Methyl-coenzyme M reductase / activation complex / ATPase / Iron-sulfur clusters | |||||||||||||||||||||||||||||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報organic phosphonate catabolic process / coenzyme-B sulfoethylthiotransferase / coenzyme-B sulfoethylthiotransferase activity / methanogenesis / ATP hydrolysis activity / ATP binding / metal ion binding / cytoplasm 類似検索 - 分子機能 | |||||||||||||||||||||||||||||||||||||||||||||||||||
| 生物種 |  Methanococcus maripaludis (古細菌) Methanococcus maripaludis (古細菌) | |||||||||||||||||||||||||||||||||||||||||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.56 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||
 データ登録者 データ登録者 | Ramirez-Amador, F. / Paul, S. / Kumar, A. / Schuller, J.M. | |||||||||||||||||||||||||||||||||||||||||||||||||||
| 資金援助 | European Union, 1件
| |||||||||||||||||||||||||||||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2025 ジャーナル: Nature / 年: 2025タイトル: Structure of the ATP-driven methyl-coenzyme M reductase activation complex. 著者: Fidel Ramírez-Amador / Sophia Paul / Anuj Kumar / Christian Lorent / Sebastian Keller / Stefan Bohn / Thinh Nguyen / Stefano Lometto / Dennis Vlegels / Jörg Kahnt / Darja Deobald / Frank ...著者: Fidel Ramírez-Amador / Sophia Paul / Anuj Kumar / Christian Lorent / Sebastian Keller / Stefan Bohn / Thinh Nguyen / Stefano Lometto / Dennis Vlegels / Jörg Kahnt / Darja Deobald / Frank Abendroth / Olalla Vázquez / Georg Hochberg / Silvan Scheller / Sven T Stripp / Jan Michael Schuller /   要旨: Methyl-coenzyme M reductase (MCR) is the enzyme responsible for nearly all biologically generated methane. Its active site comprises coenzyme F, a porphyrin-based cofactor with a central nickel ion ...Methyl-coenzyme M reductase (MCR) is the enzyme responsible for nearly all biologically generated methane. Its active site comprises coenzyme F, a porphyrin-based cofactor with a central nickel ion that is active exclusively in the Ni(I) state. How methanogenic archaea perform the reductive activation of F represents a major gap in our understanding of one of the most ancient bioenergetic systems in nature. Here we purified and characterized the MCR activation complex from Methanococcus maripaludis. McrC, a small subunit encoded in the mcr operon, co-purifies with the methanogenic marker proteins Mmp7, Mmp17, Mmp3 and the A2 component. We demonstrated that this complex can activate MCR in vitro in a strictly ATP-dependent manner, enabling the formation of methane. In addition, we determined the cryo-electron microscopy structure of the MCR activation complex exhibiting different functional states with local resolutions reaching 1.8-2.1 Å. Our data revealed three complex iron-sulfur clusters that formed an electron transfer pathway towards F. Topology and electron paramagnetic resonance spectroscopy analyses indicate that these clusters are similar to the [8Fe-9S-C] cluster, a maturation intermediate of the catalytic cofactor in nitrogenase. Altogether, our findings offer insights into the activation mechanism of MCR and prospects on the early evolution of nitrogenase. | |||||||||||||||||||||||||||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8s7v.cif.gz 8s7v.cif.gz | 811.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8s7v.ent.gz pdb8s7v.ent.gz | 654.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8s7v.json.gz 8s7v.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8s7v_validation.pdf.gz 8s7v_validation.pdf.gz | 2.3 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8s7v_full_validation.pdf.gz 8s7v_full_validation.pdf.gz | 2.4 MB | 表示 | |
| XML形式データ |  8s7v_validation.xml.gz 8s7v_validation.xml.gz | 118.8 KB | 表示 | |
| CIF形式データ |  8s7v_validation.cif.gz 8s7v_validation.cif.gz | 183.2 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/s7/8s7v https://data.pdbj.org/pub/pdb/validation_reports/s7/8s7v ftp://data.pdbj.org/pub/pdb/validation_reports/s7/8s7v ftp://data.pdbj.org/pub/pdb/validation_reports/s7/8s7v | HTTPS FTP |
-関連構造データ
| 関連構造データ |  19787MC  8s7xC  9h1lC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-Methyl-coenzyme M reductase subunit ... , 3種, 6分子 ABEDCF
| #1: タンパク質 | 分子量: 29665.488 Da / 分子数: 2 / 由来タイプ: 天然 / 詳細: Methyl-coenzyme M reductase subunit gamma / 由来: (天然)  Methanococcus maripaludis (古細菌) Methanococcus maripaludis (古細菌)参照: UniProt: A0A2L1CBG2, coenzyme-B sulfoethylthiotransferase #2: タンパク質 | 分子量: 46701.203 Da / 分子数: 2 / 由来タイプ: 天然 / 詳細: Methyl-coenzyme M reductase subunit beta / 由来: (天然)  Methanococcus maripaludis (古細菌) Methanococcus maripaludis (古細菌)参照: UniProt: A0A2L1CBB3, coenzyme-B sulfoethylthiotransferase #3: タンパク質 | 分子量: 61230.703 Da / 分子数: 2 / 由来タイプ: 天然 / 詳細: Methyl-coenzyme M reductase subunit alpha / 由来: (天然)  Methanococcus maripaludis (古細菌) Methanococcus maripaludis (古細菌)参照: UniProt: A0A2L1CBB0, coenzyme-B sulfoethylthiotransferase |
|---|
-Methanogenesis marker protein ... , 2種, 2分子 GH
| #4: タンパク質 | 分子量: 21119.539 Da / 分子数: 1 / 由来タイプ: 天然 / 詳細: Methanogenesis marker protein 17 / 由来: (天然)  Methanococcus maripaludis (古細菌) / 参照: UniProt: G0H411 Methanococcus maripaludis (古細菌) / 参照: UniProt: G0H411 |
|---|---|
| #5: タンパク質 | 分子量: 35024.746 Da / 分子数: 1 / 由来タイプ: 天然 / 詳細: Methanogenesis marker protein 7 / 由来: (天然)  Methanococcus maripaludis (古細菌) / 参照: UniProt: G0H350 Methanococcus maripaludis (古細菌) / 参照: UniProt: G0H350 |
-タンパク質 , 4種, 4分子 IKJL
| #6: タンパク質 | 分子量: 24906.715 Da / 分子数: 1 / 由来タイプ: 組換発現 / 詳細: Methyl-coenzyme M reductase operon protein C 由来: (組換発現)  Methanococcus maripaludis (古細菌) Methanococcus maripaludis (古細菌)遺伝子: GYY_08635 / 発現宿主:  Methanococcus maripaludis (古細菌) / 株 (発現宿主): JJ -upt / 参照: UniProt: G0H3B1 Methanococcus maripaludis (古細菌) / 株 (発現宿主): JJ -upt / 参照: UniProt: G0H3B1 |
|---|---|
| #7: タンパク質 | 分子量: 59572.562 Da / 分子数: 1 / 由来タイプ: 天然 / 詳細: Methyl-coenzyme M reductase system, component A2 / 由来: (天然)  Methanococcus maripaludis (古細菌) / 参照: UniProt: A0A2L1C9A1 Methanococcus maripaludis (古細菌) / 参照: UniProt: A0A2L1C9A1 |
| #8: タンパク質 | 分子量: 56520.801 Da / 分子数: 1 / 由来タイプ: 天然 / 詳細: Methanogenesis marker protein 3 / 由来: (天然)  Methanococcus maripaludis (古細菌) / 参照: UniProt: A9A8E0 Methanococcus maripaludis (古細菌) / 参照: UniProt: A9A8E0 |
| #9: タンパク質 | 分子量: 10640.849 Da / 分子数: 1 / 由来タイプ: 天然 / 詳細: DUF2098 domain-containing protein / 由来: (天然)  Methanococcus maripaludis (古細菌) / 参照: UniProt: A0A2L1CAX0 Methanococcus maripaludis (古細菌) / 参照: UniProt: A0A2L1CAX0 |
-非ポリマー , 8種, 13分子 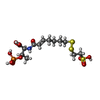

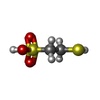










| #10: 化合物 | ChemComp-SHT / | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| #11: 化合物 | ChemComp-TP7 / | ||||||||
| #12: 化合物 | ChemComp-COM / | ||||||||
| #13: 化合物 | | #14: 化合物 | #15: 化合物 | ChemComp-ZN / | #16: 化合物 | #17: 化合物 | |
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|---|
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: 3D ARRAY / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Methyl-coenzyme M reductase activation complex binding to A2 component タイプ: COMPLEX / Entity ID: #1, #3, #2, #4-#6, #8, #7, #9 / 由来: NATURAL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 値: 0.48 MDa / 実験値: YES | |||||||||||||||
| 由来(天然) | 生物種:  Methanococcus maripaludis (古細菌) Methanococcus maripaludis (古細菌) | |||||||||||||||
| 緩衝液 | pH: 7.6 | |||||||||||||||
| 緩衝液成分 |
| |||||||||||||||
| 試料 | 濃度: 1.25 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES | |||||||||||||||
| 試料支持 | 詳細: 15 mA / グリッドの材料: COPPER / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: Quantifoil R1.2/1.3 | |||||||||||||||
| 急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE-PROPANE / 湿度: 100 % / 凍結前の試料温度: 277 K 詳細: Vitrification setup inside a Coy Lab's Vinyl Anaerobic Chamber to preserve the protein sample under strict anaerobic conditions (95% O2 / 5% H2). |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: TFS KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 165000 X / 最大 デフォーカス(公称値): 2000 nm / 最小 デフォーカス(公称値): 500 nm / Cs: 2.7 mm |
| 撮影 | 電子線照射量: 60 e/Å2 フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 実像数: 17548 詳細: 7781 micrographs with a 20 deg pretilt to overcome preferred orientation problems |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 1557902 詳細: 1023667 from non tilted and 534235 from pretilted datasets | ||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 2.56 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 484016 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: AB INITIO MODEL / 空間: REAL 詳細: Rigid body fitting in Chimera and Coot. Further real-space refinement in Phenix. | ||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | Source name: AlphaFold / タイプ: in silico model |
 ムービー
ムービー コントローラー
コントローラー





 PDBj
PDBj