+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8s63 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Low pH (5.5) as-isolated MSOX movie series dataset 3 of the copper nitrite reductase (NirK) from Bradyrhizobium japonicum USDA110 [1.71 MGy] | |||||||||
 要素 要素 | Copper-containing nitrite reductase | |||||||||
 キーワード キーワード | OXIDOREDUCTASE / electron transfer / Nitrite binding / nitrite reductase | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報denitrification pathway / nitrite reductase (NO-forming) / nitrite reductase (NO-forming) activity / nitrate assimilation / periplasmic space / copper ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Bradyrhizobium sp. (根粒菌) Bradyrhizobium sp. (根粒菌) | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.17 Å 分子置換 / 解像度: 1.17 Å | |||||||||
 データ登録者 データ登録者 | Rose, S.L. / Ferroni, F.M. / Horrell, S. / Brondino, C.D. / Eady, R.R. / Jaho, S. / Hough, M.A. / Owen, A.L. / Antonyuk, S.V. / Hasnain, S.S. | |||||||||
| 資金援助 |  アルゼンチン, アルゼンチン,  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 2024 ジャーナル: J.Mol.Biol. / 年: 2024タイトル: Spectroscopically Validated pH-dependent MSOX Movies Provide Detailed Mechanism of Copper Nitrite Reductases. 著者: Rose, S.L. / Ferroni, F.M. / Horrell, S. / Brondino, C.D. / Eady, R.R. / Jaho, S. / Hough, M.A. / Owen, R.L. / Antonyuk, S.V. / Hasnain, S.S. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8s63.cif.gz 8s63.cif.gz | 364.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8s63.ent.gz pdb8s63.ent.gz | 241.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8s63.json.gz 8s63.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8s63_validation.pdf.gz 8s63_validation.pdf.gz | 1.7 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8s63_full_validation.pdf.gz 8s63_full_validation.pdf.gz | 1.7 MB | 表示 | |
| XML形式データ |  8s63_validation.xml.gz 8s63_validation.xml.gz | 20 KB | 表示 | |
| CIF形式データ |  8s63_validation.cif.gz 8s63_validation.cif.gz | 31.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/s6/8s63 https://data.pdbj.org/pub/pdb/validation_reports/s6/8s63 ftp://data.pdbj.org/pub/pdb/validation_reports/s6/8s63 ftp://data.pdbj.org/pub/pdb/validation_reports/s6/8s63 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8r8sC  8rflC  8rfoC  8rfpC  8rfqC  8rfrC  8rfsC  8rftC  8rfuC  8rfvC  8rfwC  8rfxC  8rfyC  8rg8C  8rg9C  8rgbC  8rgcC  8rgdC  8ru9C  8rurC  8ryjC  8ryrC  8ryuC  8ryvC  8s0wC  8s2qC  8s5xC  8s5yC  8s64C  8s68C  8s69C  9evmC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 1分子 A
| #1: タンパク質 | 分子量: 39758.395 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Bradyrhizobium sp. (根粒菌) / 株: ORS375 / 遺伝子: nirK / プラスミド: p22BK / 詳細 (発現宿主): pET-22b(+):blr7089 / 発現宿主: Bradyrhizobium sp. (根粒菌) / 株: ORS375 / 遺伝子: nirK / プラスミド: p22BK / 詳細 (発現宿主): pET-22b(+):blr7089 / 発現宿主:  |
|---|
-糖 , 2種, 2分子 
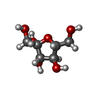

| #4: 糖 | ChemComp-GLC / |
|---|---|
| #5: 糖 | ChemComp-FRU / |
-非ポリマー , 3種, 511分子 




| #2: 化合物 | | #3: 化合物 | ChemComp-SO4 / #6: 水 | ChemComp-HOH / | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.52 Å3/Da / 溶媒含有率: 51.17 % |
|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 5.5 / 詳細: 50 mM Hepes pH 5.5, 1.8 M Ammonium Sulphate |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  Diamond Diamond  / ビームライン: I24 / 波長: 0.855 Å / ビームライン: I24 / 波長: 0.855 Å |
| 検出器 | タイプ: DECTRIS PILATUS 6M-F / 検出器: PIXEL / 日付: 2022年7月18日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.855 Å / 相対比: 1 |
| 反射 | 解像度: 1.17→52.48 Å / Num. obs: 132306 / % possible obs: 99.1 % / 冗長度: 4.3 % / CC1/2: 0.998 / Rmerge(I) obs: 0.057 / Rpim(I) all: 0.04 / Rrim(I) all: 0.071 / Net I/σ(I): 9.2 |
| 反射 シェル | 解像度: 1.17→1.19 Å / 冗長度: 3.8 % / Rmerge(I) obs: 1.698 / Num. unique obs: 6482 / CC1/2: 0.327 / Rpim(I) all: 1.191 / Rrim(I) all: 2.092 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 / 解像度: 1.17→52.049 Å / Cor.coef. Fo:Fc: 0.989 / Cor.coef. Fo:Fc free: 0.984 / SU B: 1.276 / SU ML: 0.023 / 交差検証法: FREE R-VALUE / ESU R: 0.026 / ESU R Free: 0.027 分子置換 / 解像度: 1.17→52.049 Å / Cor.coef. Fo:Fc: 0.989 / Cor.coef. Fo:Fc free: 0.984 / SU B: 1.276 / SU ML: 0.023 / 交差検証法: FREE R-VALUE / ESU R: 0.026 / ESU R Free: 0.027 詳細: Hydrogens have been used if present in the input file
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK BULK SOLVENT | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 18.505 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.17→52.049 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | Refine-ID: X-RAY DIFFRACTION / Total num. of bins used: 20
|
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj






