[English] 日本語
 Yorodumi
Yorodumi- PDB-8ryv: MSOX movie series dataset 10 (11.5 MGy) for nitrite bound BrJNiR ... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 8ryv | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | MSOX movie series dataset 10 (11.5 MGy) for nitrite bound BrJNiR (Cu containing nitrite reductase (NirK) from Bradyrhizobium japonicum USDA110 at pH 8. | |||||||||
 Components Components | Copper-containing nitrite reductase | |||||||||
 Keywords Keywords | OXIDOREDUCTASE / electron transfer / Nitrite binding / nitrite reductase | |||||||||
| Function / homology |  Function and homology information Function and homology informationdenitrification pathway / nitrite reductase (NO-forming) / nitrite reductase (NO-forming) activity / nitrate assimilation / periplasmic space / copper ion binding Similarity search - Function | |||||||||
| Biological species |  Bradyrhizobium sp. (bacteria) Bradyrhizobium sp. (bacteria) | |||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.45 Å MOLECULAR REPLACEMENT / Resolution: 1.45 Å | |||||||||
 Authors Authors | Rose, S.L. / Ferroni, F.M. / Horrell, S. / Brondino, C.D. / Eady, R.R. / Jaho, S. / Hough, M.A. / Owen, A.L. / Antonyuk, S.V. / Hasnain, S.S. | |||||||||
| Funding support |  Argentina, Argentina,  United Kingdom, 2items United Kingdom, 2items
| |||||||||
 Citation Citation |  Journal: J.Mol.Biol. / Year: 2024 Journal: J.Mol.Biol. / Year: 2024Title: Spectroscopically Validated pH-dependent MSOX Movies Provide Detailed Mechanism of Copper Nitrite Reductases. Authors: Rose, S.L. / Ferroni, F.M. / Horrell, S. / Brondino, C.D. / Eady, R.R. / Jaho, S. / Hough, M.A. / Owen, R.L. / Antonyuk, S.V. / Hasnain, S.S. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  8ryv.cif.gz 8ryv.cif.gz | 164.4 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb8ryv.ent.gz pdb8ryv.ent.gz | 125.1 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  8ryv.json.gz 8ryv.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/ry/8ryv https://data.pdbj.org/pub/pdb/validation_reports/ry/8ryv ftp://data.pdbj.org/pub/pdb/validation_reports/ry/8ryv ftp://data.pdbj.org/pub/pdb/validation_reports/ry/8ryv | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  8r8sC  8rflC  8rfoC  8rfpC  8rfqC  8rfrC  8rfsC  8rftC  8rfuC  8rfvC  8rfwC  8rfxC  8rfyC  8rg8C  8rg9C  8rgbC  8rgcC  8rgdC  8ru9C  8rurC  8ryjC  8ryrC  8ryuC  8s0wC  8s2qC  8s5xC  8s5yC  8s63C  8s64C  8s68C  8s69C  9evmC C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| Unit cell |
| ||||||||
| Components on special symmetry positions |
|
- Components
Components
-Protein / Sugars , 2 types, 2 molecules A
| #1: Protein | Mass: 39758.395 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Bradyrhizobium sp. (bacteria) / Strain: ORS375 / Gene: nirK / Plasmid: p22BK / Details (production host): pET-22b(+):blr7089 / Production host: Bradyrhizobium sp. (bacteria) / Strain: ORS375 / Gene: nirK / Plasmid: p22BK / Details (production host): pET-22b(+):blr7089 / Production host:  |
|---|---|
| #2: Polysaccharide | beta-D-fructofuranose-(2-1)-alpha-D-glucopyranose |
-Non-polymers , 5 types, 428 molecules 
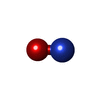







| #3: Chemical | | #4: Chemical | ChemComp-NO / | #5: Chemical | ChemComp-NO2 / | #6: Chemical | ChemComp-SO4 / #7: Water | ChemComp-HOH / | |
|---|
-Details
| Has ligand of interest | Y |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.52 Å3/Da / Density % sol: 51.17 % |
|---|---|
| Crystal grow | Temperature: 293 K / Method: vapor diffusion, hanging drop / pH: 8 / Details: 100 mM Tris pH 7.3, 1.8 M Ammonium Sulphate |
-Data collection
| Diffraction | Mean temperature: 100 K / Serial crystal experiment: N |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  Diamond Diamond  / Beamline: I24 / Wavelength: 0.77491 Å / Beamline: I24 / Wavelength: 0.77491 Å |
| Detector | Type: DECTRIS PILATUS 6M-F / Detector: PIXEL / Date: Feb 15, 2023 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.77491 Å / Relative weight: 1 |
| Reflection | Resolution: 1.45→45.204 Å / Num. obs: 70743 / % possible obs: 100 % / Redundancy: 5.5 % / CC1/2: 0.997 / Rmerge(I) obs: 0.082 / Rpim(I) all: 0.058 / Rrim(I) all: 0.101 / Net I/σ(I): 9.4 |
| Reflection shell | Resolution: 1.45→1.47 Å / Redundancy: 4.3 % / Rmerge(I) obs: 0.999 / Num. unique obs: 2237 / CC1/2: 0.392 / Rpim(I) all: 0.785 / Rrim(I) all: 1.281 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT / Resolution: 1.45→45.204 Å / Cor.coef. Fo:Fc: 0.982 / Cor.coef. Fo:Fc free: 0.974 / SU B: 1.134 / SU ML: 0.041 / Cross valid method: FREE R-VALUE / ESU R: 0.051 / ESU R Free: 0.053 MOLECULAR REPLACEMENT / Resolution: 1.45→45.204 Å / Cor.coef. Fo:Fc: 0.982 / Cor.coef. Fo:Fc free: 0.974 / SU B: 1.134 / SU ML: 0.041 / Cross valid method: FREE R-VALUE / ESU R: 0.051 / ESU R Free: 0.053 Details: Hydrogens have been used if present in the input file
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Ion probe radii: 0.8 Å / Shrinkage radii: 0.8 Å / VDW probe radii: 1.2 Å / Solvent model: MASK BULK SOLVENT | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 20.598 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 1.45→45.204 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Refine-ID: X-RAY DIFFRACTION / Total num. of bins used: 20
|
 Movie
Movie Controller
Controller


 PDBj
PDBj






