| 登録情報 | データベース: PDB / ID: 8fvf
|
|---|
| タイトル | Bromodomain of EP300 liganded with CCS-1477 |
|---|
 要素 要素 | Histone acetyltransferase p300 |
|---|
 キーワード キーワード | TRANSCRIPTION/INHIBITOR / bromodomain / small molecule ligand / complex / TRANSCRIPTION / TRANSCRIPTION-INHIBITOR complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
behavioral defense response / negative regulation of protein oligomerization / peptidyl-lysine propionylation / histone lactyltransferase (CoA-dependent) activity / peptidyl-lysine crotonylation / peptidyl-lysine butyrylation / histone butyryltransferase activity / swimming / histone H3K122 acetyltransferase activity / peptide butyryltransferase activity ...behavioral defense response / negative regulation of protein oligomerization / peptidyl-lysine propionylation / histone lactyltransferase (CoA-dependent) activity / peptidyl-lysine crotonylation / peptidyl-lysine butyrylation / histone butyryltransferase activity / swimming / histone H3K122 acetyltransferase activity / peptide butyryltransferase activity / regulation of tubulin deacetylation / histone H2B acetyltransferase activity / internal protein amino acid acetylation / peptide 2-hydroxyisobutyryltransferase activity / histone crotonyltransferase activity / protein propionyltransferase activity / NOTCH2 intracellular domain regulates transcription / thigmotaxis / L-lysine N-acetyltransferase activity, acting on acetyl phosphate as donor / positive regulation of TORC2 signaling / internal peptidyl-lysine acetylation / histone H4 acetyltransferase activity / cellular response to L-leucine / histone H3 acetyltransferase activity / NFE2L2 regulating ER-stress associated genes / acetylation-dependent protein binding / Activation of the TFAP2 (AP-2) family of transcription factors / NFE2L2 regulating inflammation associated genes / NGF-stimulated transcription / histone H3K18 acetyltransferase activity / N-terminal peptidyl-lysine acetylation / histone H3K27 acetyltransferase activity / LRR FLII-interacting protein 1 (LRRFIP1) activates type I IFN production / NFE2L2 regulates pentose phosphate pathway genes / STAT3 nuclear events downstream of ALK signaling / NFE2L2 regulating MDR associated enzymes / Polo-like kinase mediated events / host-mediated activation of viral transcription / TGFBR3 expression / regulation of androgen receptor signaling pathway / regulation of mitochondrion organization / Regulation of gene expression in late stage (branching morphogenesis) pancreatic bud precursor cells / Regulation of FOXO transcriptional activity by acetylation / RUNX3 regulates NOTCH signaling / NOTCH4 Intracellular Domain Regulates Transcription / Nuclear events mediated by NFE2L2 / Regulation of NFE2L2 gene expression / face morphogenesis / Regulation of gene expression by Hypoxia-inducible Factor / platelet formation / regulation of glycolytic process / NOTCH3 Intracellular Domain Regulates Transcription / TRAF6 mediated IRF7 activation / NFE2L2 regulating tumorigenic genes / NFE2L2 regulating anti-oxidant/detoxification enzymes / megakaryocyte development / protein-lysine-acetyltransferase activity / nuclear androgen receptor binding / protein acetylation / acyltransferase activity / STAT family protein binding / Formation of paraxial mesoderm / positive regulation of transforming growth factor beta receptor signaling pathway / acetyltransferase activity / FOXO-mediated transcription of cell death genes / fat cell differentiation / stimulatory C-type lectin receptor signaling pathway / RUNX1 interacts with co-factors whose precise effect on RUNX1 targets is not known / Zygotic genome activation (ZGA) / PI5P Regulates TP53 Acetylation / histone acetyltransferase complex / RUNX3 regulates p14-ARF / intrinsic apoptotic signaling pathway in response to DNA damage by p53 class mediator / canonical NF-kappaB signal transduction / negative regulation of gluconeogenesis / NF-kappaB binding / pre-mRNA intronic binding / Attenuation phase / somitogenesis / positive regulation of T-helper 17 cell lineage commitment / positive regulation of DNA-binding transcription factor activity / histone acetyltransferase activity / cellular response to nutrient levels / negative regulation of protein-containing complex assembly / skeletal muscle tissue development / histone acetyltransferase / regulation of cellular response to heat / NR1H3 & NR1H2 regulate gene expression linked to cholesterol transport and efflux / SARS-CoV-1 targets host intracellular signalling and regulatory pathways / Regulation of TP53 Activity through Acetylation / : / positive regulation of TORC1 signaling / 転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの / Transcriptional and post-translational regulation of MITF-M expression and activity / CD209 (DC-SIGN) signaling / transcription initiation-coupled chromatin remodeling / lung development / negative regulation of autophagy / SUMOylation of transcription cofactors / TP53 Regulates Transcription of Genes Involved in G2 Cell Cycle Arrest類似検索 - 分子機能 Nuclear receptor coactivator, CREB-bp-like, interlocking / Nuclear receptor coactivator, CREB-bp-like, interlocking domain superfamily / Creb binding / Zinc finger, TAZ-type / TAZ domain superfamily / TAZ zinc finger / Zinc finger TAZ-type profile. / TAZ zinc finger, present in p300 and CBP / : / Histone acetyltransferase p300-like, PHD domain ...Nuclear receptor coactivator, CREB-bp-like, interlocking / Nuclear receptor coactivator, CREB-bp-like, interlocking domain superfamily / Creb binding / Zinc finger, TAZ-type / TAZ domain superfamily / TAZ zinc finger / Zinc finger TAZ-type profile. / TAZ zinc finger, present in p300 and CBP / : / Histone acetyltransferase p300-like, PHD domain / Coactivator CBP, KIX domain / CREB-binding protein/p300, atypical RING domain / CBP/p300-type histone acetyltransferase domain / CBP/p300, atypical RING domain superfamily / KIX domain / CREB-binding protein/p300, atypical RING domain / KIX domain profile. / CBP/p300-type histone acetyltransferase (HAT) domain profile. / Histone acetyltransferase Rtt109/CBP / Histone acetylation protein / Histone acetylation protein / Coactivator CBP, KIX domain superfamily / Zinc finger ZZ-type signature. / Zinc-binding domain, present in Dystrophin, CREB-binding protein. / Zinc finger, ZZ type / Zinc finger, ZZ-type / Zinc finger, ZZ-type superfamily / Zinc finger ZZ-type profile. / Nuclear receptor coactivator, interlocking / Bromodomain, conserved site / Bromodomain signature. / Bromodomain / bromo domain / Bromodomain / Bromodomain (BrD) profile. / Bromodomain-like superfamily / Zinc finger, RING/FYVE/PHD-type類似検索 - ドメイン・相同性 Chem-JHL / NICKEL (II) ION / Histone acetyltransferase p300類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.1 Å 分子置換 / 解像度: 2.1 Å |
|---|
 データ登録者 データ登録者 | Schonbrunn, E. / Bikowitz, M. |
|---|
| 資金援助 | 1件 |
|---|
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024
タイトル: Group 3 medulloblastoma transcriptional networks collapse under domain specific EP300/CBP inhibition.
著者: Shendy, N.A.M. / Bikowitz, M. / Sigua, L.H. / Zhang, Y. / Mercier, A. / Khashana, Y. / Nance, S. / Liu, Q. / Delahunty, I.M. / Robinson, S. / Goel, V. / Rees, M.G. / Ronan, M.A. / Wang, T. / ...著者: Shendy, N.A.M. / Bikowitz, M. / Sigua, L.H. / Zhang, Y. / Mercier, A. / Khashana, Y. / Nance, S. / Liu, Q. / Delahunty, I.M. / Robinson, S. / Goel, V. / Rees, M.G. / Ronan, M.A. / Wang, T. / Kocak, M. / Roth, J.A. / Wang, Y. / Freeman, B.B. / Orr, B.A. / Abraham, B.J. / Roussel, M.F. / Schonbrunn, E. / Qi, J. / Durbin, A.D. |
|---|
| 履歴 | | 登録 | 2023年1月18日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2024年2月7日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2024年6月5日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.1 Å
分子置換 / 解像度: 2.1 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Nat Commun / 年: 2024
ジャーナル: Nat Commun / 年: 2024 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 8fvf.cif.gz
8fvf.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb8fvf.ent.gz
pdb8fvf.ent.gz PDB形式
PDB形式 8fvf.json.gz
8fvf.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/fv/8fvf
https://data.pdbj.org/pub/pdb/validation_reports/fv/8fvf ftp://data.pdbj.org/pub/pdb/validation_reports/fv/8fvf
ftp://data.pdbj.org/pub/pdb/validation_reports/fv/8fvf リンク
リンク 集合体
集合体


 要素
要素 Homo sapiens (ヒト) / 遺伝子: EP300, P300 / プラスミド: pNIC28-Bsa4 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: EP300, P300 / プラスミド: pNIC28-Bsa4 / 発現宿主: 
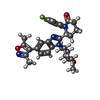








 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 22-BM / 波長: 1 Å
/ ビームライン: 22-BM / 波長: 1 Å 解析
解析 分子置換 / 解像度: 2.1→19.74 Å / SU ML: 0.21 / 交差検証法: THROUGHOUT / σ(F): 1.36 / 位相誤差: 21.96 / 立体化学のターゲット値: ML
分子置換 / 解像度: 2.1→19.74 Å / SU ML: 0.21 / 交差検証法: THROUGHOUT / σ(F): 1.36 / 位相誤差: 21.96 / 立体化学のターゲット値: ML ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj

























