| 登録情報 | データベース: PDB / ID: 8e3v
|
|---|
| タイトル | Cobalt-reconstituted nitrogenase MoFeP mutant S188A from Azotobacter vinelandii after IDS oxidation |
|---|
 要素 要素 | (Nitrogenase molybdenum-iron protein ...) x 2 |
|---|
 キーワード キーワード | OXIDOREDUCTASE / MoFeP / MoFe-protein |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
molybdenum-iron nitrogenase complex / nitrogenase / nitrogenase activity / nitrogen fixation / iron-sulfur cluster binding / ATP binding / metal ion binding類似検索 - 分子機能 Nitrogenase molybdenum-iron protein beta chain, N-terminal / Domain of unknown function (DUF3364) / Nitrogenase molybdenum-iron protein alpha chain / Nitrogenase molybdenum-iron protein beta chain / Nitrogenase component 1, alpha chain / Nitrogenase component 1, conserved site / Nitrogenases component 1 alpha and beta subunits signature 2. / Nitrogenases component 1 alpha and beta subunits signature 1. / Nitrogenase/oxidoreductase, component 1 / : / Nitrogenase component 1 type Oxidoreductase類似検索 - ドメイン・相同性 FE(8)-S(7) CLUSTER / : / 3-HYDROXY-3-CARBOXY-ADIPIC ACID / Chem-ICS / Nitrogenase molybdenum-iron protein beta chain / Nitrogenase molybdenum-iron protein alpha chain類似検索 - 構成要素 |
|---|
| 生物種 |  Azotobacter vinelandii DJ (窒素固定) Azotobacter vinelandii DJ (窒素固定) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2 Å 分子置換 / 解像度: 2 Å |
|---|
 データ登録者 データ登録者 | Rutledge, H.L. / Tezcan, F.A. |
|---|
| 資金援助 |  米国, 2件 米国, 2件 | 組織 | 認可番号 | 国 |
|---|
| National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) | GM099813 |  米国 米国 | | National Aeronautic Space Administration (NASA, United States) | 80NSSC18M0093 |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: J.Am.Chem.Soc. / 年: 2022 ジャーナル: J.Am.Chem.Soc. / 年: 2022
タイトル: Role of Serine Coordination in the Structural and Functional Protection of the Nitrogenase P-Cluster.
著者: Rutledge, H.L. / Field, M.J. / Rittle, J. / Green, M.T. / Tezcan, F.A. |
|---|
| 履歴 | | 登録 | 2022年8月17日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2022年12月14日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2022年12月21日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.journal_volume / _citation.page_first ..._citation.journal_volume / _citation.page_first / _citation.page_last / _citation_author.identifier_ORCID |
|---|
| 改定 1.2 | 2023年10月25日 | Group: Data collection / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond / pdbx_initial_refinement_model |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Azotobacter vinelandii DJ (窒素固定)
Azotobacter vinelandii DJ (窒素固定) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2 Å
分子置換 / 解像度: 2 Å  データ登録者
データ登録者 米国, 2件
米国, 2件  引用
引用 ジャーナル: J.Am.Chem.Soc. / 年: 2022
ジャーナル: J.Am.Chem.Soc. / 年: 2022 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 8e3v.cif.gz
8e3v.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb8e3v.ent.gz
pdb8e3v.ent.gz PDB形式
PDB形式 8e3v.json.gz
8e3v.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 8e3v_validation.pdf.gz
8e3v_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 8e3v_full_validation.pdf.gz
8e3v_full_validation.pdf.gz 8e3v_validation.xml.gz
8e3v_validation.xml.gz 8e3v_validation.cif.gz
8e3v_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/e3/8e3v
https://data.pdbj.org/pub/pdb/validation_reports/e3/8e3v ftp://data.pdbj.org/pub/pdb/validation_reports/e3/8e3v
ftp://data.pdbj.org/pub/pdb/validation_reports/e3/8e3v


 F&H 検索
F&H 検索 リンク
リンク 集合体
集合体
 要素
要素 Azotobacter vinelandii DJ (窒素固定)
Azotobacter vinelandii DJ (窒素固定) Azotobacter vinelandii DJ (窒素固定) / 株 (発現宿主): DJ / ATCC BAA-1303 / 参照: UniProt: P07328, nitrogenase
Azotobacter vinelandii DJ (窒素固定) / 株 (発現宿主): DJ / ATCC BAA-1303 / 参照: UniProt: P07328, nitrogenase Azotobacter vinelandii DJ (窒素固定)
Azotobacter vinelandii DJ (窒素固定) Azotobacter vinelandii DJ (窒素固定) / 株 (発現宿主): DJ / ATCC BAA-1303 / 参照: UniProt: C1DGZ8, nitrogenase
Azotobacter vinelandii DJ (窒素固定) / 株 (発現宿主): DJ / ATCC BAA-1303 / 参照: UniProt: C1DGZ8, nitrogenase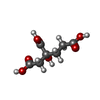
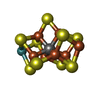

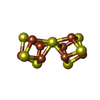

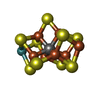


 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SSRL
SSRL  / ビームライン: BL9-2 / 波長: 1.60388, 1.61223
/ ビームライン: BL9-2 / 波長: 1.60388, 1.61223 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj




