| 登録情報 | データベース: PDB / ID: 8dea
|
|---|
| タイトル | Scaffold Hopping via Ring Opening Enables Identification of Acyclic Compounds as New Complement Factor D Inhibitors |
|---|
 要素 要素 | Complement factor D |
|---|
 キーワード キーワード | HYDROLASE/HYDROLASE INHIBITOR / serine protease inhibitor complex / HYDROLASE / HYDROLASE-HYDROLASE INHIBITOR complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
complement factor D / Alternative complement activation / complement activation / complement activation, alternative pathway / serine-type peptidase activity / platelet alpha granule lumen / protein maturation / response to bacterium / Platelet degranulation / secretory granule lumen ...complement factor D / Alternative complement activation / complement activation / complement activation, alternative pathway / serine-type peptidase activity / platelet alpha granule lumen / protein maturation / response to bacterium / Platelet degranulation / secretory granule lumen / ficolin-1-rich granule lumen / serine-type endopeptidase activity / Neutrophil degranulation / proteolysis / extracellular space / extracellular exosome / extracellular region類似検索 - 分子機能 Serine proteases, trypsin family, histidine active site / Serine proteases, trypsin family, serine active site / Serine proteases, trypsin family, histidine active site. / Peptidase S1A, chymotrypsin family / Serine proteases, trypsin family, serine active site. / Serine proteases, trypsin domain profile. / Trypsin-like serine protease / Serine proteases, trypsin domain / Trypsin / Peptidase S1, PA clan, chymotrypsin-like fold / Peptidase S1, PA clan類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 2.214 Å 分子置換 / 解像度: 2.214 Å |
|---|
 データ登録者 データ登録者 | Raman, K. / Babu, Y.S. |
|---|
| 資金援助 |  米国, 1件 米国, 1件 | 組織 | 認可番号 | 国 |
|---|
| Other private | |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: Bioorg.Med.Chem. / 年: 2022 ジャーナル: Bioorg.Med.Chem. / 年: 2022
タイトル: Scaffold hopping via ring opening enables identification of acyclic compounds as new complement Factor D inhibitors.
著者: Zhang, W. / Wu, M. / Vadlakonda, S. / Juarez, L. / Cheng, X. / Muppa, S. / Chintareddy, V. / Vogeti, L. / Kellogg-Yelder, D. / Williams, J. / Polach, K. / Chen, X. / Raman, K. / Babu, Y.S. / Kotian, P. |
|---|
| 履歴 | | 登録 | 2022年6月20日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2022年11月2日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2023年10月18日 | Group: Data collection / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond / pdbx_initial_refinement_model |
|---|
| 改定 1.2 | 2024年10月23日 | Group: Structure summary
カテゴリ: pdbx_entry_details / pdbx_modification_feature
Item: _pdbx_entry_details.has_protein_modification |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  分子置換 / 解像度: 2.214 Å
分子置換 / 解像度: 2.214 Å  データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: Bioorg.Med.Chem. / 年: 2022
ジャーナル: Bioorg.Med.Chem. / 年: 2022 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 8dea.cif.gz
8dea.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb8dea.ent.gz
pdb8dea.ent.gz PDB形式
PDB形式 8dea.json.gz
8dea.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 8dea_validation.pdf.gz
8dea_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 8dea_full_validation.pdf.gz
8dea_full_validation.pdf.gz 8dea_validation.xml.gz
8dea_validation.xml.gz 8dea_validation.cif.gz
8dea_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/de/8dea
https://data.pdbj.org/pub/pdb/validation_reports/de/8dea ftp://data.pdbj.org/pub/pdb/validation_reports/de/8dea
ftp://data.pdbj.org/pub/pdb/validation_reports/de/8dea リンク
リンク 集合体
集合体


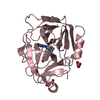

 要素
要素 Homo sapiens (ヒト) / 遺伝子: CFD, DF, PFD / Cell (発現宿主): Embryonic kidney / 細胞株 (発現宿主): Hek293 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: CFD, DF, PFD / Cell (発現宿主): Embryonic kidney / 細胞株 (発現宿主): Hek293 / 発現宿主:  Homo sapiens (ヒト) / 参照: UniProt: P00746, complement factor D
Homo sapiens (ヒト) / 参照: UniProt: P00746, complement factor D X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 回転陽極 / タイプ: RIGAKU MICROMAX-007 / 波長: 1.5418 Å
回転陽極 / タイプ: RIGAKU MICROMAX-007 / 波長: 1.5418 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー







 PDBj
PDBj