| 登録情報 | データベース: PDB / ID: 8bss
|
|---|
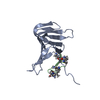
| タイトル | Solution Structure of thanatin-like derivative 5 in complex with E. coli LptA mutant Q62L |
|---|
 要素 要素 | - Lipopolysaccharide export system protein LptA
- Thanatin-like derivative
|
|---|
 キーワード キーワード | ANTIBIOTIC |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
glycolipid transfer activity / transporter complex / lipopolysaccharide transport / Gram-negative-bacterium-type cell outer membrane assembly / defense response to fungus / lipopolysaccharide binding / cell outer membrane / outer membrane-bounded periplasmic space / killing of cells of another organism / periplasmic space ...glycolipid transfer activity / transporter complex / lipopolysaccharide transport / Gram-negative-bacterium-type cell outer membrane assembly / defense response to fungus / lipopolysaccharide binding / cell outer membrane / outer membrane-bounded periplasmic space / killing of cells of another organism / periplasmic space / defense response to bacterium / innate immune response / extracellular region / identical protein binding類似検索 - 分子機能 : / Lipopolysaccharide export system protein LptA / Organic solvent tolerance-like, N-terminal / LptA/(LptD N-terminal domain) LPS transport protein類似検索 - ドメイン・相同性 Lipopolysaccharide export system protein LptA / Thanatin類似検索 - 構成要素 |
|---|
| 生物種 |   Escherichia coli K-12 (大腸菌) Escherichia coli K-12 (大腸菌)
 Podisus maculiventris (昆虫) Podisus maculiventris (昆虫) |
|---|
| 手法 | 溶液NMR / molecular dynamics |
|---|
 データ登録者 データ登録者 | Oi, K.K. / Jurt, S. / Moehle, K. / Zerbe, O. |
|---|
| 資金援助 | European Union, 1件 | 組織 | 認可番号 | 国 |
|---|
| Innosuisse | 33285.1 IP-LS | European Union |
|
|---|
 引用 引用 |  ジャーナル: Sci Adv / 年: 2023 ジャーナル: Sci Adv / 年: 2023
タイトル: Peptidomimetic antibiotics disrupt the lipopolysaccharide transport bridge of drug-resistant Enterobacteriaceae.
著者: Schuster, M. / Brabet, E. / Oi, K.K. / Desjonqueres, N. / Moehle, K. / Le Poupon, K. / Hell, S. / Gable, S. / Rithie, V. / Dillinger, S. / Zbinden, P. / Luther, A. / Li, C. / Stiegeler, S. / ...著者: Schuster, M. / Brabet, E. / Oi, K.K. / Desjonqueres, N. / Moehle, K. / Le Poupon, K. / Hell, S. / Gable, S. / Rithie, V. / Dillinger, S. / Zbinden, P. / Luther, A. / Li, C. / Stiegeler, S. / D'Arco, C. / Locher, H. / Remus, T. / DiMaio, S. / Motta, P. / Wach, A. / Jung, F. / Upert, G. / Obrecht, D. / Benghezal, M. / Zerbe, O. |
|---|
| 履歴 | | 登録 | 2022年11月26日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2023年6月7日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 2.0 | 2023年6月14日 | Group: Atomic model / Database references ...Atomic model / Database references / Derived calculations / Source and taxonomy / Structure summary
カテゴリ: atom_site / entity ...atom_site / entity / pdbx_entity_src_syn / pdbx_struct_mod_residue / struct_ref / struct_ref_seq / struct_ref_seq_dif
Item: _entity.pdbx_description / _pdbx_entity_src_syn.organism_common_name ..._entity.pdbx_description / _pdbx_entity_src_syn.organism_common_name / _struct_ref.db_code / _struct_ref.db_name / _struct_ref.pdbx_align_begin / _struct_ref.pdbx_db_accession / _struct_ref.pdbx_seq_one_letter_code / _struct_ref_seq.db_align_beg / _struct_ref_seq.db_align_end / _struct_ref_seq.pdbx_db_accession |
|---|
| 改定 3.0 | 2023年11月15日 | Group: Atomic model / Data collection / Derived calculations
カテゴリ: atom_site / chem_comp_atom ...atom_site / chem_comp_atom / chem_comp_bond / struct_conn
Item: _atom_site.auth_atom_id / _atom_site.label_atom_id / _struct_conn.pdbx_leaving_atom_flag |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 Podisus maculiventris (昆虫)
Podisus maculiventris (昆虫) データ登録者
データ登録者 引用
引用 ジャーナル: Sci Adv / 年: 2023
ジャーナル: Sci Adv / 年: 2023 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 8bss.cif.gz
8bss.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb8bss.ent.gz
pdb8bss.ent.gz PDB形式
PDB形式 8bss.json.gz
8bss.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 8bss_validation.pdf.gz
8bss_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 8bss_full_validation.pdf.gz
8bss_full_validation.pdf.gz 8bss_validation.xml.gz
8bss_validation.xml.gz 8bss_validation.cif.gz
8bss_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/bs/8bss
https://data.pdbj.org/pub/pdb/validation_reports/bs/8bss ftp://data.pdbj.org/pub/pdb/validation_reports/bs/8bss
ftp://data.pdbj.org/pub/pdb/validation_reports/bs/8bss

 F&H 検索
F&H 検索 リンク
リンク 集合体
集合体
 要素
要素

 Podisus maculiventris (昆虫) / 参照: UniProt: P55788
Podisus maculiventris (昆虫) / 参照: UniProt: P55788 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj HSQC
HSQC