| 登録情報 | データベース: PDB / ID: 8ah7
|
|---|
| タイトル | Crystal Structure of the third PDZ domain of PSD-95 protein in the space group P212121 at pH 4.0 |
|---|
 要素 要素 | cDNA FLJ50577, highly similar to Discs large homolog 4 |
|---|
 キーワード キーワード | PROTEIN BINDING / PDZ domain |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
neuromuscular junction / kinase binding / chemical synaptic transmission / neuron projection類似検索 - 分子機能 PDZ-associated domain of NMDA receptors / PDZ-associated domain of NMDA receptors / Disks large 1-like / : / Guanylate kinase, conserved site / Guanylate kinase-like signature. / Guanylate kinase-like domain profile. / Guanylate kinase-like domain / Guanylate kinase/L-type calcium channel beta subunit / Guanylate kinase ...PDZ-associated domain of NMDA receptors / PDZ-associated domain of NMDA receptors / Disks large 1-like / : / Guanylate kinase, conserved site / Guanylate kinase-like signature. / Guanylate kinase-like domain profile. / Guanylate kinase-like domain / Guanylate kinase/L-type calcium channel beta subunit / Guanylate kinase / Guanylate kinase homologues. / PDZ domain / SH3 domain / PDZ domain profile. / Domain present in PSD-95, Dlg, and ZO-1/2. / PDZ domain / PDZ superfamily / Src homology 3 domains / SH3-like domain superfamily / Src homology 3 (SH3) domain profile. / SH3 domain / P-loop containing nucleoside triphosphate hydrolase類似検索 - ドメイン・相同性 cDNA FLJ50577, highly similar to Discs large homolog 4類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.25 Å 分子置換 / 解像度: 1.25 Å |
|---|
| Model details | pH 4.0 P3112 polymorph |
|---|
 データ登録者 データ登録者 | Camara-Artigas, A. / Salinas-Garcia, M.C. |
|---|
| 資金援助 |  スペイン, 1件 スペイン, 1件 | 組織 | 認可番号 | 国 |
|---|
| Other government | UAL-FEDER-UAL18-BIO-B005-B |  スペイン スペイン |
|
|---|
 引用 引用 |  ジャーナル: Crystals / 年: 2023 ジャーナル: Crystals / 年: 2023
タイトル: pH-Driven Polymorphic Behaviour of the Third PDZ Domain of PSD95: The Role of Electrostatic Interactions
著者: Salinas-Garcia, M.C. / Plaza-Garrido, M. / Gavira, J.A. / Murciano-Calles, J. / Andujar-Sanchez, M. / Ortiz-Salmeron, E. / Martinez, J.C. / Camara-Artigas, A. |
|---|
| 履歴 | | 登録 | 2022年7月20日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2023年3月8日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2024年2月7日 | Group: Data collection / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond / pdbx_initial_refinement_model |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.25 Å
分子置換 / 解像度: 1.25 Å  データ登録者
データ登録者 スペイン, 1件
スペイン, 1件  引用
引用 ジャーナル: Crystals / 年: 2023
ジャーナル: Crystals / 年: 2023 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 8ah7.cif.gz
8ah7.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb8ah7.ent.gz
pdb8ah7.ent.gz PDB形式
PDB形式 8ah7.json.gz
8ah7.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/ah/8ah7
https://data.pdbj.org/pub/pdb/validation_reports/ah/8ah7 ftp://data.pdbj.org/pub/pdb/validation_reports/ah/8ah7
ftp://data.pdbj.org/pub/pdb/validation_reports/ah/8ah7 リンク
リンク 集合体
集合体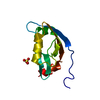
 要素
要素 Homo sapiens (ヒト) / プラスミド: pET15 / 発現宿主:
Homo sapiens (ヒト) / プラスミド: pET15 / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ESRF
ESRF  / ビームライン: ID30B / 波長: 0.7749 Å
/ ビームライン: ID30B / 波長: 0.7749 Å 分子置換
分子置換 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー







 PDBj
PDBj


