+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7z9z | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Mutant L39Y of recombinant bovine beta-lactoglobulin in complex with pramocaine | ||||||
 要素 要素 | Beta-lactoglobulin | ||||||
 キーワード キーワード | TRANSPORT PROTEIN / lactoglobulin / ligand / mutation / pramocaine | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報retinol binding / long-chain fatty acid binding / extracellular region / identical protein binding 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 2.5 Å 分子置換 / 解像度: 2.5 Å | ||||||
 データ登録者 データ登録者 | Loch, J.I. / Kurpiewska, K. / Bonarek, P. | ||||||
| 資金援助 | 1件
| ||||||
 引用 引用 |  ジャーナル: J.Mol.Recognit. / 年: 2023 ジャーナル: J.Mol.Recognit. / 年: 2023タイトル: beta-Lactoglobulin variants as potential carriers of pramoxine: Comprehensive structural and biophysical studies. 著者: Bonarek, P. / Mularczyk, D. / Loch, J.I. / Kurpiewska, K. / Dziedzicka-Wasylewska, M. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7z9z.cif.gz 7z9z.cif.gz | 49.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7z9z.ent.gz pdb7z9z.ent.gz | 表示 |  PDB形式 PDB形式 | |
| PDBx/mmJSON形式 |  7z9z.json.gz 7z9z.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7z9z_validation.pdf.gz 7z9z_validation.pdf.gz | 611.3 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7z9z_full_validation.pdf.gz 7z9z_full_validation.pdf.gz | 613.2 KB | 表示 | |
| XML形式データ |  7z9z_validation.xml.gz 7z9z_validation.xml.gz | 8.8 KB | 表示 | |
| CIF形式データ |  7z9z_validation.cif.gz 7z9z_validation.cif.gz | 11.1 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/z9/7z9z https://data.pdbj.org/pub/pdb/validation_reports/z9/7z9z ftp://data.pdbj.org/pub/pdb/validation_reports/z9/7z9z ftp://data.pdbj.org/pub/pdb/validation_reports/z9/7z9z | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 単位格子 |
| ||||||||
| Components on special symmetry positions |
|
- 要素
要素
| #1: タンパク質 | 分子量: 18325.111 Da / 分子数: 1 / 変異: L1A, I2S, L39Y / 由来タイプ: 組換発現 / 由来: (組換発現)   |
|---|---|
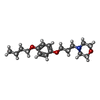
| #2: 化合物 | ChemComp-PX9 / |
| #3: 化合物 | ChemComp-SO4 / |
| #4: 化合物 | ChemComp-CL / |
| #5: 水 | ChemComp-HOH / |
| 研究の焦点であるリガンドがあるか | Y |
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.48 Å3/Da / 溶媒含有率: 50.38 % |
|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 8.5 / 詳細: 2.4 M ammonium sulfate in 0.5 M Tris-HCl, pH 8.5 |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来: SEALED TUBE / タイプ: Agilent SuperNova / 波長: 1.54056 Å |
| 検出器 | タイプ: AGILENT ATLAS CCD / 検出器: CCD / 日付: 2018年10月19日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.54056 Å / 相対比: 1 |
| 反射 | 解像度: 2.5→14.76 Å / Num. obs: 6648 / % possible obs: 98.9 % / 冗長度: 4.7 % / CC1/2: 0.995 / Rmerge(I) obs: 0.111 / Rrim(I) all: 0.124 / Net I/σ(I): 8.1 |
| 反射 シェル | 解像度: 2.5→2.61 Å / Rmerge(I) obs: 0.64 / Mean I/σ(I) obs: 1.7 / Num. unique obs: 796 / CC1/2: 0.723 / Rrim(I) all: 0.751 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 1BSY 解像度: 2.5→14.76 Å / Cor.coef. Fo:Fc: 0.941 / Cor.coef. Fo:Fc free: 0.891 / SU B: 12.339 / SU ML: 0.256 / 交差検証法: FREE R-VALUE / ESU R: 0.85 / ESU R Free: 0.349
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK BULK SOLVENT | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 38.846 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.5→14.76 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | Refine-ID: X-RAY DIFFRACTION / Total num. of bins used: 20
|
 ムービー
ムービー コントローラー
コントローラー







 PDBj
PDBj