| 登録情報 | データベース: PDB / ID: 7wku
|
|---|
| タイトル | Structure of PDCoV Mpro in complex with an inhibitor |
|---|
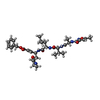
 要素 要素 | - N-[(5-METHYLISOXAZOL-3-YL)CARBONYL]ALANYL-L-VALYL-N~1~-((1R,2Z)-4-(BENZYLOXY)-4-OXO-1-{[(3R)-2-OXOPYRROLIDIN-3-YL]METHYL}BUT-2-ENYL)-L-LEUCINAMIDE
- Peptidase C30
|
|---|
 キーワード キーワード | VIRAL PROTEIN / virus / viral protease / drug design / inhibitor / broad-spectrum antivirals |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
RNA exonuclease activity / endoplasmic reticulum-Golgi intermediate compartment / host cell membrane / DNA helicase activity / methyltransferase activity / host cell endoplasmic reticulum-Golgi intermediate compartment / omega peptidase activity / methylation / endonuclease activity / membrane => GO:0016020 ...RNA exonuclease activity / endoplasmic reticulum-Golgi intermediate compartment / host cell membrane / DNA helicase activity / methyltransferase activity / host cell endoplasmic reticulum-Golgi intermediate compartment / omega peptidase activity / methylation / endonuclease activity / membrane => GO:0016020 / RNA helicase activity / host cell perinuclear region of cytoplasm / viral protein processing / induction by virus of host autophagy / viral RNA genome replication / cysteine-type endopeptidase activity / RNA-dependent RNA polymerase activity / DNA-templated transcription / RNA binding / zinc ion binding / ATP binding類似検索 - 分子機能 Non-structural protein 5, deltacoronavirus / Nonstructural protein 14, deltacoronavirus / Nonstructural protein 15, middle domain, deltacoronavirus / Nonstructural protein 15, N-terminal, deltacoronavirus / RNA-dependent RNA polymerase, deltacoronavirus / Non-structural protein 6, deltacoronavirus / Non-structural protein 2, IBV-like / Viral (Superfamily 1) RNA helicase / NSP15, NendoU domain, coronavirus / Nonstructural protein 13, 1B domain, coronavirus ...Non-structural protein 5, deltacoronavirus / Nonstructural protein 14, deltacoronavirus / Nonstructural protein 15, middle domain, deltacoronavirus / Nonstructural protein 15, N-terminal, deltacoronavirus / RNA-dependent RNA polymerase, deltacoronavirus / Non-structural protein 6, deltacoronavirus / Non-structural protein 2, IBV-like / Viral (Superfamily 1) RNA helicase / NSP15, NendoU domain, coronavirus / Nonstructural protein 13, 1B domain, coronavirus / Non-structural protein NSP15, N-terminal domain superfamily, coronavirus / Non-structural protein NSP15, middle domain superfamily / Coronavirus replicase NSP15, N-terminal oligomerization / Nonstructural protein 15, middle domain, coronavirus / Coronavirus replicase NSP15, middle domain / Coronavirus replicase NSP15, N-terminal oligomerisation / Non-structural protein NSP16, coronavirus-like / Non-structural protein 14, coronavirus / RNA polymerase, N-terminal, coronavirus / Coronavirus 2'-O-methyltransferase / Coronavirus proofreading exoribonuclease / Coronavirus RNA-dependent RNA polymerase, N-terminal / Nidovirus RdRp-associated nucleotidyl transferase (NiRAN) domain / Nonstructural protein 13, zinc-binding domain, coronavirus-like / Coronaviridae zinc-binding (CV ZBD) domain profile. / Nidovirus RdRp-associated nucleotidyl transferase (NiRAN) domain profile. / Endoribonuclease EndoU-like / NendoU domain, nidovirus / Coronavirus replicase NSP15, uridylate-specific endoribonuclease / (+) RNA virus helicase core domain / (+)RNA virus helicase core domain profile. / Coronavirus Nsp4 C-terminal (Nsp4C) domain profile. / Papain-like protease, thumb domain superfamily, coronavirus / Peptidase C30, coronavirus / Peptidase C16, coronavirus / Non-structural protein NSP9, coronavirus / Non-structural protein NSP8, coronavirus / RNA synthesis protein NSP10, coronavirus / Non-structural protein NSP4, C-terminal, coronavirus / RNA synthesis protein NSP10 superfamily, coronavirus / Non-structural protein NSP9 superfamily, coronavirus / Non-structural protein NSP7 superfamily, coronavirus / Non-structural protein NSP8 superfamily, coronavirus / Non-structural protein NSP4, C-terminal superfamily, coronavirus / Peptidase C30, domain 3, coronavirus / Non-structural protein 6, coronavirus / Coronavirus replicase NSP3, C-terminal / Non-structural protein NSP4, N-terminal, coronavirus / Coronavirus endopeptidase C30 / Coronavirus papain-like peptidase / Coronavirus replicase NSP8 / Coronavirus RNA synthesis protein NSP10 / Coronavirus replicase NSP4, C-terminal / Coronavirus replicase NSP6 / Coronavirus replicase NSP4, N-terminal / Coronavirus replicase NSP3, C-terminal / Coronavirus main protease (M-pro) domain profile. / Coronavirus replicase NSP9 / Non-structural protein 3, X-domain-like / Macro domain / Appr-1"-p processing enzyme / Macro domain / Macro domain profile. / Macro domain-like / RNA-directed RNA polymerase, C-terminal domain / Viral RNA-dependent RNA polymerase / RNA-directed RNA polymerase, catalytic domain / RdRp of positive ssRNA viruses catalytic domain profile. / S-adenosyl-L-methionine-dependent methyltransferase superfamily / Peptidase S1, PA clan, chymotrypsin-like fold / Peptidase S1, PA clan / DNA/RNA polymerase superfamily / P-loop containing nucleoside triphosphate hydrolase類似検索 - ドメイン・相同性 N-[(5-METHYLISOXAZOL-3-YL)CARBONYL]ALANYL-L-VALYL-N~1~-((1R,2Z)-4-(BENZYLOXY)-4-OXO-1-{[(3R)-2-OXOPYRROLIDIN-3-YL]METHYL}BUT-2-ENYL)-L-LEUCINAMIDE / ORF1ab polyprotein類似検索 - 構成要素 |
|---|
| 生物種 |  Porcine deltacoronavirus (ウイルス) Porcine deltacoronavirus (ウイルス)
synthetic construct (人工物) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.6 Å 分子置換 / 解像度: 2.6 Å |
|---|
 データ登録者 データ登録者 | Wang, F.H. / Yang, H.T. |
|---|
| 資金援助 |  中国, 1件 中国, 1件 | 組織 | 認可番号 | 国 |
|---|
| National Natural Science Foundation of China (NSFC) | 81772204 |  中国 中国 |
|
|---|
 引用 引用 |  ジャーナル: Viruses / 年: 2022 ジャーナル: Viruses / 年: 2022
タイトル: The Structure of the Porcine Deltacoronavirus Main Protease Reveals a Conserved Target for the Design of Antivirals.
著者: Wang, F. / Chen, C. / Wang, Z. / Han, X. / Shi, P. / Zhou, K. / Liu, X. / Xiao, Y. / Cai, Y. / Huang, J. / Zhang, L. / Yang, H. |
|---|
| 履歴 | | 登録 | 2022年1月11日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2022年5月25日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 2.0 | 2023年11月15日 | Group: Atomic model / Data collection / Derived calculations
カテゴリ: atom_site / chem_comp_atom ...atom_site / chem_comp_atom / chem_comp_bond / pdbx_validate_rmsd_bond / pdbx_validate_torsion / struct_conn
Item: _atom_site.auth_atom_id / _atom_site.label_atom_id ..._atom_site.auth_atom_id / _atom_site.label_atom_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr2_label_atom_id |
|---|
| 改定 2.1 | 2023年11月29日 | Group: Refinement description / カテゴリ: pdbx_initial_refinement_model |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Porcine deltacoronavirus (ウイルス)
Porcine deltacoronavirus (ウイルス) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.6 Å
分子置換 / 解像度: 2.6 Å  データ登録者
データ登録者 中国, 1件
中国, 1件  引用
引用 ジャーナル: Viruses / 年: 2022
ジャーナル: Viruses / 年: 2022 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 7wku.cif.gz
7wku.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb7wku.ent.gz
pdb7wku.ent.gz PDB形式
PDB形式 7wku.json.gz
7wku.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 7wku_validation.pdf.gz
7wku_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 7wku_full_validation.pdf.gz
7wku_full_validation.pdf.gz 7wku_validation.xml.gz
7wku_validation.xml.gz 7wku_validation.cif.gz
7wku_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/wk/7wku
https://data.pdbj.org/pub/pdb/validation_reports/wk/7wku ftp://data.pdbj.org/pub/pdb/validation_reports/wk/7wku
ftp://data.pdbj.org/pub/pdb/validation_reports/wk/7wku
 F&H 検索
F&H 検索 リンク
リンク 集合体
集合体
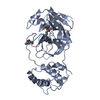





 要素
要素 Porcine deltacoronavirus (ウイルス)
Porcine deltacoronavirus (ウイルス)
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SSRF
SSRF  / ビームライン: BL19U1 / 波長: 0.97923 Å
/ ビームライン: BL19U1 / 波長: 0.97923 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj