+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7uq3 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | JmjC domain-containing protein 5 (JMJD5) in complex with Mn and (S)-2-(1-hydroxy-2,5-dioxopyrrolidin-3-yl)acetic acid | ||||||
 要素 要素 | Bifunctional peptidase and arginyl-hydroxylase JMJD5 | ||||||
 キーワード キーワード | OXIDOREDUCTASE / NON-HEME / IRON / 2-OXOGLUTARATE / DIOXYGENASE / JMJC / JMJC DOMAIN / Lysine-specific demethylase 8 / JmjC domain-containing protein 5 / Arginyl C-3 Hydroxylase / JMJD5 / KDM8 / OXYGENASE / HYPOXIA / DNA-BINDING / METAL-BINDING / TRANSLATION / DSBH / FACIAL TRIAD / CYTOPLASM / JMJC HYDROXYLASE / JMJC DEMETHYLASE / KDMS / POST-TRANSLATIONAL MODIFICATIONS / PTM / BETA-HYDROXYLATION / HYDROXYLATION / ARGININE HYDROXYLATION / RCC1 domain-containing protein 1 / RCCD1 / Regulator of chromosome condensation / 40S ribosomal protein S6 / RPS6 / RIBOSOME BIOGENESIS / TRANSCRIPTION / EPIGENETIC REGULATION / SIGNALING / DEVELOPMENT / CELL STRUCTURE / TRANSCRIPTION ACTIVATOR/INHIBITOR / PHOSPHORYLATION / CANCER / POLYMORPHISM | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報[protein]-arginine 3-hydroxylase / peptidyl-arginine 3-dioxygenase activity / histone H3K36 demethylase activity / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素 / Protein hydroxylation / aminopeptidase activity / regulation of signal transduction by p53 class mediator / circadian regulation of gene expression / protein destabilization / G2/M transition of mitotic cell cycle ...[protein]-arginine 3-hydroxylase / peptidyl-arginine 3-dioxygenase activity / histone H3K36 demethylase activity / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素 / Protein hydroxylation / aminopeptidase activity / regulation of signal transduction by p53 class mediator / circadian regulation of gene expression / protein destabilization / G2/M transition of mitotic cell cycle / p53 binding / chromosome / fibroblast proliferation / endopeptidase activity / in utero embryonic development / negative regulation of DNA-templated transcription / chromatin binding / positive regulation of DNA-templated transcription / proteolysis / nucleoplasm / metal ion binding / nucleus / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.49 Å 分子置換 / 解像度: 1.49 Å | ||||||
 データ登録者 データ登録者 | Chowdhury, R. / Islam, M.S. / Schofield, C.J. | ||||||
| 資金援助 | 1件
| ||||||
 引用 引用 |  ジャーナル: Sci Rep / 年: 2022 ジャーナル: Sci Rep / 年: 2022タイトル: Structural analysis of the 2-oxoglutarate binding site of the circadian rhythm linked oxygenase JMJD5. 著者: Islam, M.S. / Markoulides, M. / Chowdhury, R. / Schofield, C.J. #1:  ジャーナル: Nat Commun / 年: 2018 ジャーナル: Nat Commun / 年: 2018タイトル: JMJD5 is a human arginyl C-3 hydroxylase. 著者: Wilkins, S.E. / Islam, M.S. / Gannon, J.M. / Markolovic, S. / Hopkinson, R.J. / Ge, W. / Schofield, C.J. / Chowdhury, R. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7uq3.cif.gz 7uq3.cif.gz | 159.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7uq3.ent.gz pdb7uq3.ent.gz | 124.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7uq3.json.gz 7uq3.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7uq3_validation.pdf.gz 7uq3_validation.pdf.gz | 772.3 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7uq3_full_validation.pdf.gz 7uq3_full_validation.pdf.gz | 772 KB | 表示 | |
| XML形式データ |  7uq3_validation.xml.gz 7uq3_validation.xml.gz | 13.8 KB | 表示 | |
| CIF形式データ |  7uq3_validation.cif.gz 7uq3_validation.cif.gz | 20.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/uq/7uq3 https://data.pdbj.org/pub/pdb/validation_reports/uq/7uq3 ftp://data.pdbj.org/pub/pdb/validation_reports/uq/7uq3 ftp://data.pdbj.org/pub/pdb/validation_reports/uq/7uq3 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 29733.547 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: KDM8, JMJD5 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: KDM8, JMJD5 / 発現宿主:  参照: UniProt: Q8N371, [protein]-arginine 3-hydroxylase, 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素 | ||||
|---|---|---|---|---|---|
| #2: 化合物 | ChemComp-MN / | ||||
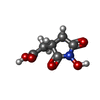
| #3: 化合物 | ChemComp-O2U / [( | ||||
| #4: 化合物 | | #5: 水 | ChemComp-HOH / | 研究の焦点であるリガンドがあるか | Y | |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.11 Å3/Da / 溶媒含有率: 41.58 % |
|---|---|
| 結晶化 | 温度: 298 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 6.5 詳細: Sample: 22 mg/mL JMJD5, 1.5 mM MnCl2, 5 mM compound (IS-52); Reservoir: 0.1 M Bis-Tris pH 6.5, 15.0 % PEG3350, 0.002 M MnCl2; Cryo-protection: 25% (v/v) glycerol; Method: 300 nL sitting drops ...詳細: Sample: 22 mg/mL JMJD5, 1.5 mM MnCl2, 5 mM compound (IS-52); Reservoir: 0.1 M Bis-Tris pH 6.5, 15.0 % PEG3350, 0.002 M MnCl2; Cryo-protection: 25% (v/v) glycerol; Method: 300 nL sitting drops (sample:well, 2:1 ratio) |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  Diamond Diamond  / ビームライン: I02 / 波長: 0.98 Å / ビームライン: I02 / 波長: 0.98 Å | ||||||||||||||||||||||||
| 検出器 | タイプ: DECTRIS PILATUS 6M / 検出器: PIXEL / 日付: 2015年5月5日 / 詳細: MIRRORS | ||||||||||||||||||||||||
| 放射 | モノクロメーター: SI 111 / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | ||||||||||||||||||||||||
| 放射波長 | 波長: 0.98 Å / 相対比: 1 | ||||||||||||||||||||||||
| 反射 | 解像度: 1.49→78.23 Å / Num. obs: 41815 / % possible obs: 100 % / 冗長度: 7 % / Biso Wilson estimate: 14 Å2 / CC1/2: 0.996 / Rpim(I) all: 0.04 / Rrim(I) all: 0.107 / Rsym value: 0.099 / Net I/σ(I): 11 | ||||||||||||||||||||||||
| 反射 シェル | Diffraction-ID: 1
|
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 4GJZ 解像度: 1.49→49.853 Å / SU ML: 0.12 / 交差検証法: THROUGHOUT / σ(F): 1.35 / 位相誤差: 14.13 / 立体化学のターゲット値: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 70.15 Å2 / Biso mean: 21.1625 Å2 / Biso min: 6.89 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 1.49→49.853 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | Refine-ID: X-RAY DIFFRACTION / Rfactor Rfree error: 0 / % reflection obs: 100 %
|
 ムービー
ムービー コントローラー
コントローラー







 PDBj
PDBj