+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7pil | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the Rhodobacter sphaeroides RC-LH1-PufXY monomer complex at 2.5 A | ||||||||||||
 要素 要素 |
| ||||||||||||
 キーワード キーワード | PHOTOSYNTHESIS / Photosynthetic bacteria / light harvesting complex / Cryo-EM / RC-LH1 / RC-LH1-PufX / RC-LH1-PufX-Y | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報organelle inner membrane / plasma membrane-derived chromatophore membrane / plasma membrane light-harvesting complex / bacteriochlorophyll binding / photosynthesis, light reaction / electron transporter, transferring electrons within the cyclic electron transport pathway of photosynthesis activity / photosynthetic electron transport in photosystem II / photosynthesis / membrane / metal ion binding / plasma membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Rhodobacter sphaeroides (バクテリア) Rhodobacter sphaeroides (バクテリア) | ||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.5 Å | ||||||||||||
 データ登録者 データ登録者 | Qian, P. / Hunter, C.N. | ||||||||||||
| 資金援助 |  英国, 3件 英国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Biochem J / 年: 2021 ジャーナル: Biochem J / 年: 2021タイトル: Cryo-EM structure of the monomeric Rhodobacter sphaeroides RC-LH1 core complex at 2.5 Å. 著者: Pu Qian / David J K Swainsbury / Tristan I Croll / Jack H Salisbury / Elizabeth C Martin / Philip J Jackson / Andrew Hitchcock / Pablo Castro-Hartmann / Kasim Sader / C Neil Hunter /   要旨: Reaction centre light-harvesting 1 (RC-LH1) complexes are the essential components of bacterial photosynthesis. The membrane-intrinsic LH1 complex absorbs light and the energy migrates to an enclosed ...Reaction centre light-harvesting 1 (RC-LH1) complexes are the essential components of bacterial photosynthesis. The membrane-intrinsic LH1 complex absorbs light and the energy migrates to an enclosed RC where a succession of electron and proton transfers conserves the energy as a quinol, which is exported to the cytochrome bc1 complex. In some RC-LH1 variants quinols can diffuse through small pores in a fully circular, 16-subunit LH1 ring, while in others missing LH1 subunits create a gap for quinol export. We used cryogenic electron microscopy to obtain a 2.5 Å resolution structure of one such RC-LH1, a monomeric complex from Rhodobacter sphaeroides. The structure shows that the RC is partly enclosed by a 14-subunit LH1 ring in which each αβ heterodimer binds two bacteriochlorophylls and, unusually for currently reported complexes, two carotenoids rather than one. Although the extra carotenoids confer an advantage in terms of photoprotection and light harvesting, they could impede passage of quinones through small, transient pores in the LH1 ring, necessitating a mechanism to create a dedicated quinone channel. The structure shows that two transmembrane proteins play a part in stabilising an open ring structure; one of these components, the PufX polypeptide, is augmented by a hitherto undescribed protein subunit we designate as protein-Y, which lies against the transmembrane regions of the thirteenth and fourteenth LH1α polypeptides. Protein-Y prevents LH1 subunits 11-14 adjacent to the RC QB site from bending inwards towards the RC and, with PufX preventing complete encirclement of the RC, this pair of polypeptides ensures unhindered quinone diffusion. #1:  ジャーナル: Acta Crystallogr., Sect. D: Biol. Crystallogr. ジャーナル: Acta Crystallogr., Sect. D: Biol. Crystallogr.年: 2018 タイトル: Real-space refinement in PHENIX for cryo-EM and crystallography 著者: qian, P. / Hunter, C.N. #2:  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Cryo-EM structure of the Rhodobacter sphaeroides RC-LH1-PufXY monomer complex at 2.5 A 著者: Qian, P. / Hunter, C.N. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7pil.cif.gz 7pil.cif.gz | 552 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7pil.ent.gz pdb7pil.ent.gz | 表示 |  PDB形式 PDB形式 | |
| PDBx/mmJSON形式 |  7pil.json.gz 7pil.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7pil_validation.pdf.gz 7pil_validation.pdf.gz | 3.6 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7pil_full_validation.pdf.gz 7pil_full_validation.pdf.gz | 3.7 MB | 表示 | |
| XML形式データ |  7pil_validation.xml.gz 7pil_validation.xml.gz | 86.1 KB | 表示 | |
| CIF形式データ |  7pil_validation.cif.gz 7pil_validation.cif.gz | 121.4 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/pi/7pil https://data.pdbj.org/pub/pdb/validation_reports/pi/7pil ftp://data.pdbj.org/pub/pdb/validation_reports/pi/7pil ftp://data.pdbj.org/pub/pdb/validation_reports/pi/7pil | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-Light-harvesting protein B-875 ... , 2種, 28分子 AAABACADAEAFAGAHAIAJAKALAMANBABBBCBDBEBFBGBHBIBJBKBLBMBN
| #1: タンパク質 | 分子量: 6516.847 Da / 分子数: 14 / 由来タイプ: 天然 由来: (天然)  Rhodobacter sphaeroides (strain ATCC 17023 / DSM 158 / JCM 6121 / NBRC 12203 / NCIMB 8253 / ATH 2.4.1.) (バクテリア) Rhodobacter sphaeroides (strain ATCC 17023 / DSM 158 / JCM 6121 / NBRC 12203 / NCIMB 8253 / ATH 2.4.1.) (バクテリア)株: ATCC 17023 / DSM 158 / JCM 6121 / NBRC 12203 / NCIMB 8253 / ATH 2.4.1. 参照: UniProt: Q3J1A4 #2: タンパク質・ペプチド | 分子量: 4943.656 Da / 分子数: 14 / 由来タイプ: 天然 由来: (天然)  Rhodobacter sphaeroides (strain ATCC 17023 / DSM 158 / JCM 6121 / NBRC 12203 / NCIMB 8253 / ATH 2.4.1.) (バクテリア) Rhodobacter sphaeroides (strain ATCC 17023 / DSM 158 / JCM 6121 / NBRC 12203 / NCIMB 8253 / ATH 2.4.1.) (バクテリア)株: ATCC 17023 / DSM 158 / JCM 6121 / NBRC 12203 / NCIMB 8253 / ATH 2.4.1. 参照: UniProt: Q3J1A3 |
|---|
-Reaction center protein ... , 3種, 3分子 HLM
| #3: タンパク質 | 分子量: 26544.471 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Rhodobacter sphaeroides (strain ATCC 17023 / DSM 158 / JCM 6121 / NBRC 12203 / NCIMB 8253 / ATH 2.4.1.) (バクテリア) Rhodobacter sphaeroides (strain ATCC 17023 / DSM 158 / JCM 6121 / NBRC 12203 / NCIMB 8253 / ATH 2.4.1.) (バクテリア)株: ATCC 17023 / DSM 158 / JCM 6121 / NBRC 12203 / NCIMB 8253 / ATH 2.4.1. 参照: UniProt: Q3J170 |
|---|---|
| #4: タンパク質 | 分子量: 31346.389 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Rhodobacter sphaeroides (strain ATCC 17023 / DSM 158 / JCM 6121 / NBRC 12203 / NCIMB 8253 / ATH 2.4.1.) (バクテリア) Rhodobacter sphaeroides (strain ATCC 17023 / DSM 158 / JCM 6121 / NBRC 12203 / NCIMB 8253 / ATH 2.4.1.) (バクテリア)株: ATCC 17023 / DSM 158 / JCM 6121 / NBRC 12203 / NCIMB 8253 / ATH 2.4.1. 参照: UniProt: Q3J1A5 |
| #5: タンパク質 | 分子量: 34398.543 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Rhodobacter sphaeroides (strain ATCC 17023 / 2.4.1 / NCIB 8253 / DSM 158) (バクテリア) Rhodobacter sphaeroides (strain ATCC 17023 / 2.4.1 / NCIB 8253 / DSM 158) (バクテリア)株: ATCC 17023 / 2.4.1 / NCIB 8253 / DSM 158 / 参照: UniProt: Q3J1A6 |
-タンパク質・ペプチド / タンパク質 / 糖 , 3種, 6分子 UUX

| #10: 糖 | ChemComp-LMT / #6: タンパク質・ペプチド | | 分子量: 5126.067 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Rhodobacter sphaeroides (strain ATCC 17023 / DSM 158 / JCM 6121 / NBRC 12203 / NCIMB 8253 / ATH 2.4.1.) (バクテリア) Rhodobacter sphaeroides (strain ATCC 17023 / DSM 158 / JCM 6121 / NBRC 12203 / NCIMB 8253 / ATH 2.4.1.) (バクテリア)株: ATCC 17023 / DSM 158 / JCM 6121 / NBRC 12203 / NCIMB 8253 / ATH 2.4.1. 参照: UniProt: U5NME9 #7: タンパク質 | | 分子量: 5980.121 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Rhodobacter sphaeroides (strain ATCC 17023 / DSM 158 / JCM 6121 / NBRC 12203 / NCIMB 8253 / ATH 2.4.1.) (バクテリア) Rhodobacter sphaeroides (strain ATCC 17023 / DSM 158 / JCM 6121 / NBRC 12203 / NCIMB 8253 / ATH 2.4.1.) (バクテリア)株: ATCC 17023 / DSM 158 / JCM 6121 / NBRC 12203 / NCIMB 8253 / ATH 2.4.1. 参照: UniProt: P13402 |
|---|
-非ポリマー , 9種, 73分子 




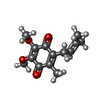











| #8: 化合物 | ChemComp-BCL / #9: 化合物 | ChemComp-SPO / #11: 化合物 | ChemComp-3PE / #12: 化合物 | #13: 化合物 | #14: 化合物 | ChemComp-UQ1 / | #15: 化合物 | ChemComp-CD4 / ( | #16: 化合物 | ChemComp-FE / | #17: 水 | ChemComp-HOH / | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Light harvesting complex / タイプ: COMPLEX / Entity ID: #1-#7 / 由来: NATURAL |
|---|---|
| 由来(天然) | 生物種:  Cereibacter sphaeroides 2.4.1 (バクテリア) Cereibacter sphaeroides 2.4.1 (バクテリア) |
| 緩衝液 | pH: 7.8 / 詳細: 20 mM HEPES, pH 7.8 , 0.03% beta-DDM |
| 緩衝液成分 | 濃度: 20 mMol / 式: HEPES |
| 試料 | 濃度: 4 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES / 詳細: in 0.03% beta-DDM detergent |
| 試料支持 | グリッドの材料: COPPER / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: Quantifoil R1.2/1.3 |
| 急速凍結 | 装置: FEI VITROBOT MARK III / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 277 K / 詳細: QF R1.2/1.3 grid coated graphene oxide |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 120000 X / 最大 デフォーカス(公称値): 2200 nm / 最小 デフォーカス(公称値): 800 nm / Cs: 2.7 mm / C2レンズ絞り径: 50 µm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 平均露光時間: 12.21 sec. / 電子線照射量: 44.94 e/Å2 フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 撮影したグリッド数: 1 / 実像数: 3180 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 1057624 | ||||||||||||||||||||||||
| 対称性 | 点対称性: C1 (非対称) | ||||||||||||||||||||||||
| 3次元再構成 | 解像度: 2.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 250613 / アルゴリズム: BACK PROJECTION / クラス平均像の数: 1 / 対称性のタイプ: POINT |
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj











