+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7o60 | ||||||
|---|---|---|---|---|---|---|---|
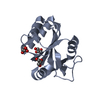
| タイトル | Crystal structure of human myelin protein P2 at room temperature from joint X-ray and neutron refinement. | ||||||
 要素 要素 | Myelin P2 protein | ||||||
 キーワード キーワード | LIPID BINDING PROTEIN / myelin / fatty acid binding protein / neuropathy / beta barrel | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報membrane organization / cholesterol binding / fatty acid transport / fatty acid binding / myelin sheath / extracellular exosome / nucleus / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / 中性子回折 / NUCLEAR REACTOR / X線回折 / 中性子回折 / NUCLEAR REACTOR /  分子置換 / 解像度: 2 Å 分子置換 / 解像度: 2 Å | ||||||
 データ登録者 データ登録者 | Laulumaa, S. / Blakeley, M.P. / Kursula, P. | ||||||
 引用 引用 |  ジャーナル: Febs J. / 年: 2021 ジャーナル: Febs J. / 年: 2021タイトル: Human myelin protein P2: from crystallography to time-lapse membrane imaging and neuropathy-associated variants. 著者: Uusitalo, M. / Klenow, M.B. / Laulumaa, S. / Blakeley, M.P. / Simonsen, A.C. / Ruskamo, S. / Kursula, P. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7o60.cif.gz 7o60.cif.gz | 93.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7o60.ent.gz pdb7o60.ent.gz | 59.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7o60.json.gz 7o60.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7o60_validation.pdf.gz 7o60_validation.pdf.gz | 314.3 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7o60_full_validation.pdf.gz 7o60_full_validation.pdf.gz | 316.4 KB | 表示 | |
| XML形式データ |  7o60_validation.xml.gz 7o60_validation.xml.gz | 5.6 KB | 表示 | |
| CIF形式データ |  7o60_validation.cif.gz 7o60_validation.cif.gz | 8.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/o6/7o60 https://data.pdbj.org/pub/pdb/validation_reports/o6/7o60 ftp://data.pdbj.org/pub/pdb/validation_reports/o6/7o60 ftp://data.pdbj.org/pub/pdb/validation_reports/o6/7o60 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 14991.470 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: PMP2 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: PMP2 / 発現宿主:  |
|---|---|
| #2: 化合物 | ChemComp-CIT / |
| #3: 化合物 | ChemComp-PLM / |
| #4: 化合物 | ChemComp-DOD / |
| 研究の焦点であるリガンドがあるか | N |
| Has protein modification | Y |
-実験情報
-実験
| 実験 |
|
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.82 Å3/Da / 溶媒含有率: 56.42 % |
|---|---|
| 結晶化 | 温度: 281 K / 手法: 蒸気拡散法, ハンギングドロップ法 / 詳細: 28% PEG 6000, 0.1 M citrate, pD 4.75 |
-データ収集
| 回折 |
| |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 |
| |||||||||||||||||||||||||||
| 検出器 |
| |||||||||||||||||||||||||||
| 放射 |
| |||||||||||||||||||||||||||
| 放射波長 |
| |||||||||||||||||||||||||||
| 反射 | Biso Wilson estimate: 27.66 Å2 / Entry-ID: 7O60
| |||||||||||||||||||||||||||
| 反射 シェル |
|
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | SU ML: 0.167 / 交差検証法: FREE R-VALUE / 構造決定の手法:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2→14.53 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
|
 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj















