+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7o2k | ||||||
|---|---|---|---|---|---|---|---|
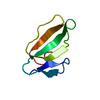
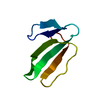
| タイトル | NMR solution structue of cytotoxin 2 from Naja Kaouthia | ||||||
 要素 要素 | Cytotoxin 2 | ||||||
 キーワード キーワード | TOXIN / S-type cytotoxin / cobra venom / three-finger protein / membrane-active polypeptide | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報other organism cell membrane / toxin activity / killing of cells of another organism / extracellular region / membrane 類似検索 - 分子機能 | ||||||
| 生物種 |  Naja kaouthia (コブラ) Naja kaouthia (コブラ) | ||||||
| 手法 | 溶液NMR / torsion angle dynamics | ||||||
 データ登録者 データ登録者 | Dubinnyi, M.A. / Dubovskii, P.V. | ||||||
 引用 引用 |  ジャーナル: Biochem.Biophys.Res.Commun. / 年: 2021 ジャーナル: Biochem.Biophys.Res.Commun. / 年: 2021タイトル: The omega-loop of cobra cytotoxins tolerates multiple amino acid substitutions. 著者: Dubinnyi, M.A. / Dubovskii, P.V. / Starkov, V.G. / Utkin, Y.N. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7o2k.cif.gz 7o2k.cif.gz | 343.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7o2k.ent.gz pdb7o2k.ent.gz | 282.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7o2k.json.gz 7o2k.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7o2k_validation.pdf.gz 7o2k_validation.pdf.gz | 443.1 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7o2k_full_validation.pdf.gz 7o2k_full_validation.pdf.gz | 571.3 KB | 表示 | |
| XML形式データ |  7o2k_validation.xml.gz 7o2k_validation.xml.gz | 23.2 KB | 表示 | |
| CIF形式データ |  7o2k_validation.cif.gz 7o2k_validation.cif.gz | 38 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/o2/7o2k https://data.pdbj.org/pub/pdb/validation_reports/o2/7o2k ftp://data.pdbj.org/pub/pdb/validation_reports/o2/7o2k ftp://data.pdbj.org/pub/pdb/validation_reports/o2/7o2k | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 6756.275 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Naja kaouthia (コブラ) / 参照: UniProt: P01445 Naja kaouthia (コブラ) / 参照: UniProt: P01445 |
|---|---|
| #2: 水 | ChemComp-HOH / |
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 | タイプ: solution / 内容: 2 mM cytotoxin 2, 3 mM Cytotoxin 3, 90% H2O/10% D2O 詳細: 3 mM No Cytotoxin 3 from Naja kaouthia, 2 mM No Cytotoxin 2 from Naja kaouthia, 90% H2O/10% D2O Label: The mixture of CT3 and CT2 Naja kaouthia / 溶媒系: 90% H2O/10% D2O | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料 |
| ||||||||||||
| 試料状態 | イオン強度: 0 mM / Ionic strength err: 0 / Label: Cytotoxins 2 and 3 / pH: 6 / PH err: 0.1 / 圧: 1 atm / Pressure err: 0.05 / 温度: 303 K / Temperature err: 1 |
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: torsion angle dynamics / ソフトェア番号: 1 詳細: 582 NOE constraints, 109 angular constraints, 27 H-bonds, 4 SS-bonds | ||||||||||||||||||||
| 代表構造 | 選択基準: fewest violations | ||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: target function / 計算したコンフォーマーの数: 200 / 登録したコンフォーマーの数: 20 |
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj


 HSQC
HSQC