| 登録情報 | データベース: PDB / ID: 7o25
|
|---|
| タイトル | Complex-B bound [FeFe]-hydrogenase maturase HydE from T. maritima (reaction triggered in the crystal) |
|---|
 要素 要素 | [FeFe] hydrogenase maturase subunit HydE |
|---|
 キーワード キーワード | METAL BINDING PROTEIN / Radical SAM protein / Hydrogenase maturase / metalloprotein |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
water-soluble vitamin biosynthetic process / sulfur compound biosynthetic process / 酸化還元酵素; 含硫化合物に対し酸化酵素として働く / 2 iron, 2 sulfur cluster binding / transferase activity / 4 iron, 4 sulfur cluster binding / oxidoreductase activity / metal ion binding類似検索 - 分子機能 Biotin and Thiamin Synthesis associated domain / [FeFe]-hydrogenase maturation HydE, radical SAM / HydE/PylB-like / Biotin and thiamin synthesis-associated domain / Biotin and Thiamin Synthesis associated domain / Elp3/MiaB/NifB / Elongator protein 3, MiaB family, Radical SAM / Radical SAM superfamily / Radical SAM core domain profile. / Radical SAM / Aldolase-type TIM barrel類似検索 - ドメイン・相同性 S-adenosyl-L-cysteine / CARBON MONOXIDE / CYANIDE ION / CYSTEINE / : / METHIONINE / PHOSPHATE ION / PYRUVIC ACID / S-ADENOSYLMETHIONINE / IRON/SULFUR CLUSTER / [FeFe] hydrogenase maturase subunit HydE類似検索 - 構成要素 |
|---|
| 生物種 |   Thermotoga maritima (バクテリア) Thermotoga maritima (バクテリア) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.34 Å 分子置換 / 解像度: 1.34 Å |
|---|
 データ登録者 データ登録者 | Rohac, R. / Martin, L. / Liu, L. / Basu, D. / Tao, L. / Britt, R.D. / Rauchfuss, T. / Nicolet, Y. |
|---|
| 資金援助 |  フランス, フランス,  米国, 3件 米国, 3件 | 組織 | 認可番号 | 国 |
|---|
| Agence Nationale de la Recherche (ANR) | ANR-15-IDEX-02 |  フランス フランス | | National Institutes of Health/National Institute of Environmental Health Sciences (NIH/NIEHS) | 1R35GM126961 |  米国 米国 | | National Institutes of Health/National Institute of Environmental Health Sciences (NIH/NIEHS) | GM-61153 |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: J.Am.Chem.Soc. / 年: 2021 ジャーナル: J.Am.Chem.Soc. / 年: 2021
タイトル: Crystal Structure of the [FeFe]-Hydrogenase Maturase HydE Bound to Complex-B.
著者: Rohac, R. / Martin, L. / Liu, L. / Basu, D. / Tao, L. / Britt, R.D. / Rauchfuss, T.B. / Nicolet, Y. |
|---|
| 履歴 | | 登録 | 2021年3月30日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2021年5月26日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2021年8月18日 | Group: Advisory / Database references
カテゴリ: citation / citation_author ...citation / citation_author / database_2 / pdbx_unobs_or_zero_occ_atoms
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_ASTM / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year / _citation_author.identifier_ORCID / _citation_author.name / _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
| 改定 2.0 | 2023年11月15日 | Group: Atomic model / Data collection
カテゴリ: atom_site / atom_site_anisotrop ...atom_site / atom_site_anisotrop / chem_comp_atom / chem_comp_bond
Item: _atom_site.auth_atom_id / _atom_site.label_atom_id ..._atom_site.auth_atom_id / _atom_site.label_atom_id / _atom_site_anisotrop.pdbx_auth_atom_id / _atom_site_anisotrop.pdbx_label_atom_id |
|---|
| 改定 2.1 | 2024年1月31日 | Group: Refinement description / カテゴリ: pdbx_initial_refinement_model |
|---|
| 改定 2.2 | 2025年10月1日 | Group: Derived calculations / Structure summary
カテゴリ: pdbx_entry_details / struct_conn / struct_conn_type
Item: _pdbx_entry_details.has_protein_modification |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 Thermotoga maritima (バクテリア)
Thermotoga maritima (バクテリア) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.34 Å
分子置換 / 解像度: 1.34 Å  データ登録者
データ登録者 フランス,
フランス,  米国, 3件
米国, 3件  引用
引用 ジャーナル: J.Am.Chem.Soc. / 年: 2021
ジャーナル: J.Am.Chem.Soc. / 年: 2021 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 7o25.cif.gz
7o25.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb7o25.ent.gz
pdb7o25.ent.gz PDB形式
PDB形式 7o25.json.gz
7o25.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 7o25_validation.pdf.gz
7o25_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 7o25_full_validation.pdf.gz
7o25_full_validation.pdf.gz 7o25_validation.xml.gz
7o25_validation.xml.gz 7o25_validation.cif.gz
7o25_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/o2/7o25
https://data.pdbj.org/pub/pdb/validation_reports/o2/7o25 ftp://data.pdbj.org/pub/pdb/validation_reports/o2/7o25
ftp://data.pdbj.org/pub/pdb/validation_reports/o2/7o25 リンク
リンク 集合体
集合体
 要素
要素
 Thermotoga maritima (バクテリア)
Thermotoga maritima (バクテリア)






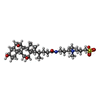




















 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ESRF
ESRF  / ビームライン: ID23-1 / 波長: 0.9805 Å
/ ビームライン: ID23-1 / 波長: 0.9805 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー



















 PDBj
PDBj































