| 登録情報 | データベース: PDB / ID: 7n21
|
|---|
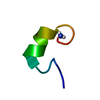
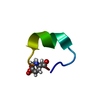
| タイトル | NMR structure of AnIB-OH |
|---|
 要素 要素 | Alpha-conotoxin AnIB |
|---|
 キーワード キーワード | TOXIN / alpha-conotoxin / CC-C-C framework / alpha4/7 pattern / sulfotyrosine |
|---|
| 機能・相同性 | Conotoxin, alpha-type, conserved site / Alpha-conotoxin family signature. / host cell postsynaptic membrane / acetylcholine receptor inhibitor activity / ion channel regulator activity / toxin activity / extracellular region / Alpha-conotoxin AnIB 機能・相同性情報 機能・相同性情報 |
|---|
| 生物種 |  Conus anemone (アネモネイモ) Conus anemone (アネモネイモ) |
|---|
| 手法 | 溶液NMR / simulated annealing / na |
|---|
| Model details | AnIB, tyrosine sulfated, C-terminal acid |
|---|
 データ登録者 データ登録者 | Conibear, A.C. / Rosengren, K.J. / Lee, H.S. |
|---|
| 資金援助 |  オーストラリア, 2件 オーストラリア, 2件 | 組織 | 認可番号 | 国 |
|---|
| The University Grants Committee, Research Grants Council (RGC) | 616535 |  オーストラリア オーストラリア | | The University Grants Committee, Research Grants Council (RGC) | 613982 |  オーストラリア オーストラリア |
|
|---|
 引用 引用 |  ジャーナル: Rsc Med Chem / 年: 2021 ジャーナル: Rsc Med Chem / 年: 2021
タイトル: Posttranslational modifications of alpha-conotoxins: sulfotyrosine and C-terminal amidation stabilise structures and increase acetylcholine receptor binding.
著者: Ho, T.N.T. / Lee, H.S. / Swaminathan, S. / Goodwin, L. / Rai, N. / Ushay, B. / Lewis, R.J. / Rosengren, K.J. / Conibear, A.C. |
|---|
| 履歴 | | 登録 | 2021年5月28日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2021年11月17日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2023年6月14日 | Group: Other / カテゴリ: pdbx_database_status / Item: _pdbx_database_status.status_code_nmr_data |
|---|
| 改定 1.2 | 2023年11月15日 | Group: Data collection / カテゴリ: chem_comp_atom / chem_comp_bond |
|---|
| 改定 1.3 | 2024年11月6日 | Group: Database references / Structure summary
カテゴリ: database_2 / pdbx_entry_details / pdbx_modification_feature
Item: _database_2.pdbx_DOI / _pdbx_entry_details.has_protein_modification |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Conus anemone (アネモネイモ)
Conus anemone (アネモネイモ) データ登録者
データ登録者 オーストラリア, 2件
オーストラリア, 2件  引用
引用 ジャーナル: Rsc Med Chem / 年: 2021
ジャーナル: Rsc Med Chem / 年: 2021 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 7n21.cif.gz
7n21.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb7n21.ent.gz
pdb7n21.ent.gz PDB形式
PDB形式 7n21.json.gz
7n21.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 7n21_validation.pdf.gz
7n21_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 7n21_full_validation.pdf.gz
7n21_full_validation.pdf.gz 7n21_validation.xml.gz
7n21_validation.xml.gz 7n21_validation.cif.gz
7n21_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/n2/7n21
https://data.pdbj.org/pub/pdb/validation_reports/n2/7n21 ftp://data.pdbj.org/pub/pdb/validation_reports/n2/7n21
ftp://data.pdbj.org/pub/pdb/validation_reports/n2/7n21





 リンク
リンク 集合体
集合体
 要素
要素 Conus anemone (アネモネイモ) / 参照: UniProt: P0C1V7
Conus anemone (アネモネイモ) / 参照: UniProt: P0C1V7 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj HSQC
HSQC