+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7jqr | ||||||
|---|---|---|---|---|---|---|---|
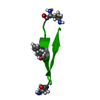
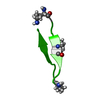
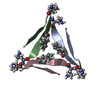
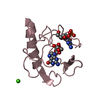
| タイトル | Abeta 16-36 beta-hairpin mimic with E22G Arctic mutation | ||||||
 要素 要素 | Abeta 16-36 beta-hairpin mimic VAL-ORN-LYS-LEU-VAL-MEA-PHE-ALA-GLY-ORN-ALA-ILE-ILE-GLY-LEU-MET | ||||||
 キーワード キーワード |  DE NOVO PROTEIN (De novo) / DE NOVO PROTEIN (De novo) /  Alzheimer's disease (アルツハイマー病) / Alzheimer's disease (アルツハイマー病) /  amyloid (アミロイド) / amyloid (アミロイド) /  Abeta (アミロイドβ) / Abeta (アミロイドβ) /  oligomer (オリゴマー) / familial mutant oligomer (オリゴマー) / familial mutant | ||||||
| 機能・相同性 | IODIDE ION 機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  単波長異常分散 / 解像度: 2.07 Å 単波長異常分散 / 解像度: 2.07 Å | ||||||
 データ登録者 データ登録者 | Kreutzer, A.G. / McKnelly, K.J. / Nowick, J.S. | ||||||
| 資金援助 |  米国, 1件 米国, 1件
| ||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 2022 ジャーナル: Biochemistry / 年: 2022タイトル: Effects of Familial Alzheimer's Disease Mutations on the Assembly of a beta-Hairpin Peptide Derived from A beta 16-36 . 著者: McKnelly, K.J. / Kreutzer, A.G. / Howitz, W.J. / Haduong, K. / Yoo, S. / Hart, C. / Nowick, J.S. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7jqr.cif.gz 7jqr.cif.gz | 18.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7jqr.ent.gz pdb7jqr.ent.gz | 13.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7jqr.json.gz 7jqr.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/jq/7jqr https://data.pdbj.org/pub/pdb/validation_reports/jq/7jqr ftp://data.pdbj.org/pub/pdb/validation_reports/jq/7jqr ftp://data.pdbj.org/pub/pdb/validation_reports/jq/7jqr | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | x 6
| ||||||||||||
| 単位格子 |
| ||||||||||||
| Components on special symmetry positions |
|
- 要素
要素
| #1: タンパク質・ペプチド | 分子量: 1722.209 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成)   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||
|---|---|---|---|---|---|
| #2: 化合物 |  ヨウ化物 ヨウ化物#3: 水 | ChemComp-HOH / |  水 水研究の焦点であるリガンドがあるか | N | |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.55 Å3/Da / 溶媒含有率: 51.71 % |
|---|---|
結晶化 | 温度: 297.15 K / 手法: 蒸気拡散法, ハンギングドロップ法 / 詳細: calcium chloride, sodium acetate, isopropanol |
-データ収集
| 回折 | 平均測定温度: 123.15 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: RIGAKU MICROMAX-007 HF / 波長: 1.54 Å 回転陽極 / タイプ: RIGAKU MICROMAX-007 HF / 波長: 1.54 Å |
| 検出器 | タイプ: RIGAKU SATURN 944+ / 検出器: CCD / 日付: 2017年10月13日 |
| 放射 | モノクロメーター: Cu anode / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1.54 Å / 相対比: 1 : 1.54 Å / 相対比: 1 |
| 反射 | 解像度: 2.07→19.28 Å / Num. obs: 2070 / % possible obs: 99.37 % / 冗長度: 90.7 % / CC1/2: 1 / Rmerge(I) obs: 0.01235 / Rrim(I) all: 0.01747 / Net I/σ(I): 65.87 |
| 反射 シェル | 解像度: 2.075→2.149 Å / Num. unique obs: 126 / CC1/2: 0.999 / % possible all: 100 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  単波長異常分散 / 解像度: 2.07→19.28 Å / SU ML: 0.04 / 交差検証法: THROUGHOUT / σ(F): 1.34 / 位相誤差: 19.92 / 立体化学のターゲット値: ML 単波長異常分散 / 解像度: 2.07→19.28 Å / SU ML: 0.04 / 交差検証法: THROUGHOUT / σ(F): 1.34 / 位相誤差: 19.92 / 立体化学のターゲット値: ML
| ||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | ||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 89.73 Å2 / Biso mean: 40.8307 Å2 / Biso min: 20.94 Å2 | ||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 2.07→19.28 Å
| ||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.075→2.149 Å / Rfactor Rfree error: 0 / Total num. of bins used: 1
| ||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Origin x: 13.6285 Å / Origin y: 13.1734 Å / Origin z: 27.118 Å
| ||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ | Selection details: chain 'A' and (resid 1 through 16 ) |
 ムービー
ムービー コントローラー
コントローラー








 PDBj
PDBj




