+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7e5e | ||||||
|---|---|---|---|---|---|---|---|
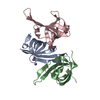
| タイトル | Crystal structure of GDP-bound GNAS in complex with the cyclic peptide inhibitor GD20 | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | SIGNALING PROTEIN / G protein / GNAS / adenylyl cyclase / inactive state / inhibitor / mRNA display / RaPID / cyclic peptide / GD20 | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報PKA activation in glucagon signalling / developmental growth / hair follicle placode formation / D1 dopamine receptor binding / intracellular transport / vascular endothelial cell response to laminar fluid shear stress / renal water homeostasis / activation of adenylate cyclase activity / Hedgehog 'off' state / adenylate cyclase-activating adrenergic receptor signaling pathway ...PKA activation in glucagon signalling / developmental growth / hair follicle placode formation / D1 dopamine receptor binding / intracellular transport / vascular endothelial cell response to laminar fluid shear stress / renal water homeostasis / activation of adenylate cyclase activity / Hedgehog 'off' state / adenylate cyclase-activating adrenergic receptor signaling pathway / cellular response to glucagon stimulus / regulation of insulin secretion / adenylate cyclase activator activity / trans-Golgi network membrane / negative regulation of inflammatory response to antigenic stimulus / bone development / G-protein beta/gamma-subunit complex binding / platelet aggregation / cognition / adenylate cyclase-activating G protein-coupled receptor signaling pathway / Prostacyclin signalling through prostacyclin receptor / Glucagon signaling in metabolic regulation / Glucagon-type ligand receptors / Vasopressin regulates renal water homeostasis via Aquaporins / Glucagon-like Peptide-1 (GLP1) regulates insulin secretion / G alpha (z) signalling events / cellular response to catecholamine stimulus / ADORA2B mediated anti-inflammatory cytokines production / sensory perception of smell / adenylate cyclase-activating dopamine receptor signaling pathway / GPER1 signaling / cellular response to prostaglandin E stimulus / heterotrimeric G-protein complex / positive regulation of cold-induced thermogenesis / G protein activity / High laminar flow shear stress activates signaling by PIEZO1 and PECAM1:CDH5:KDR in endothelial cells / G alpha (i) signalling events / G alpha (s) signalling events / 加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 / G protein-coupled receptor signaling pathway / GTPase activity / GTP binding / extracellular exosome / metal ion binding / membrane / plasma membrane / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト)synthetic construct (人工物) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.95 Å 分子置換 / 解像度: 1.95 Å | ||||||
 データ登録者 データ登録者 | Hu, Q. / Dai, S. / Shokat, K.M. | ||||||
| 資金援助 |  米国, 1件 米国, 1件
| ||||||
 引用 引用 |  ジャーナル: Cell / 年: 2022 ジャーナル: Cell / 年: 2022タイトル: State-selective modulation of heterotrimeric G alpha s signaling with macrocyclic peptides. 著者: Dai, S.A. / Hu, Q. / Gao, R. / Blythe, E.E. / Touhara, K.K. / Peacock, H. / Zhang, Z. / von Zastrow, M. / Suga, H. / Shokat, K.M. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7e5e.cif.gz 7e5e.cif.gz | 346.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7e5e.ent.gz pdb7e5e.ent.gz | 238.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7e5e.json.gz 7e5e.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7e5e_validation.pdf.gz 7e5e_validation.pdf.gz | 3.3 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7e5e_full_validation.pdf.gz 7e5e_full_validation.pdf.gz | 3.3 MB | 表示 | |
| XML形式データ |  7e5e_validation.xml.gz 7e5e_validation.xml.gz | 52.6 KB | 表示 | |
| CIF形式データ |  7e5e_validation.cif.gz 7e5e_validation.cif.gz | 71.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/e5/7e5e https://data.pdbj.org/pub/pdb/validation_reports/e5/7e5e ftp://data.pdbj.org/pub/pdb/validation_reports/e5/7e5e ftp://data.pdbj.org/pub/pdb/validation_reports/e5/7e5e | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||
| 2 | 
| ||||||||||||
| 3 | 
| ||||||||||||
| 4 | 
| ||||||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 40512.871 Da / 分子数: 4 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: GNAS, GNAS1, GSP / 発現宿主: Homo sapiens (ヒト) / 遺伝子: GNAS, GNAS1, GSP / 発現宿主:  #2: タンパク質・ペプチド | 分子量: 1854.182 Da / 分子数: 4 / 由来タイプ: 合成 / 由来: (合成) synthetic construct (人工物) #3: 化合物 | ChemComp-GDP / #4: 化合物 | ChemComp-CL / #5: 水 | ChemComp-HOH / | 研究の焦点であるリガンドがあるか | Y | Has protein modification | Y | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.18 Å3/Da / 溶媒含有率: 43.62 % |
|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 8.2 / 詳細: 0.1M Tris 8.2, 26% PEG 4000, 0.8M LiCl |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ALS ALS  / ビームライン: 8.2.1 / 波長: 0.999965 Å / ビームライン: 8.2.1 / 波長: 0.999965 Å |
| 検出器 | タイプ: ADSC QUANTUM 315r / 検出器: CCD / 日付: 2018年12月14日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.999965 Å / 相対比: 1 |
| 反射 | 解像度: 1.95→50 Å / Num. obs: 95974 / % possible obs: 97 % / 冗長度: 3.4 % / Biso Wilson estimate: 19.71 Å2 / CC1/2: 0.991 / Rmerge(I) obs: 0.081 / Rpim(I) all: 0.059 / Net I/σ(I): 11.4 |
| 反射 シェル | 解像度: 1.95→1.98 Å / Rmerge(I) obs: 1.202 / Mean I/σ(I) obs: 0.815 / Num. unique obs: 4489 / CC1/2: 0.422 / Rpim(I) all: 0.762 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 6AU6 解像度: 1.95→48.54 Å / SU ML: 0.2546 / 交差検証法: FREE R-VALUE / σ(F): 1.96 / 位相誤差: 29.8601 立体化学のターゲット値: GeoStd + Monomer Library + CDL v1.2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 29.01 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.95→48.54 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
|
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj















