+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7dwq | |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Photosystem I from a chlorophyll d-containing cyanobacterium Acaryochloris marina | |||||||||||||||||||||||||||
 要素 要素 |
| |||||||||||||||||||||||||||
 キーワード キーワード | PHOTOSYNTHESIS / Photosystem I / reaction center / cryo-EM / cyanobacteria | |||||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報photosystem I reaction center / photosystem I / photosynthetic electron transport in photosystem I / photosystem I / chlorophyll binding / plasma membrane-derived thylakoid membrane / photosynthesis / endomembrane system / 4 iron, 4 sulfur cluster binding / electron transfer activity ...photosystem I reaction center / photosystem I / photosynthetic electron transport in photosystem I / photosystem I / chlorophyll binding / plasma membrane-derived thylakoid membrane / photosynthesis / endomembrane system / 4 iron, 4 sulfur cluster binding / electron transfer activity / oxidoreductase activity / magnesium ion binding / metal ion binding 類似検索 - 分子機能 | |||||||||||||||||||||||||||
| 生物種 |  Acaryochloris marina MBIC11017 (バクテリア) Acaryochloris marina MBIC11017 (バクテリア) Acaryochloris marinan MBIC11017 (バクテリア) Acaryochloris marinan MBIC11017 (バクテリア) Acaryochloris marina MBIC 11017 (バクテリア) Acaryochloris marina MBIC 11017 (バクテリア) | |||||||||||||||||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||||||||||||||||||||
 データ登録者 データ登録者 | Chen, J.H. / Zhang, X. / Shen, J.R. | |||||||||||||||||||||||||||
 引用 引用 |  ジャーナル: J Integr Plant Biol / 年: 2021 ジャーナル: J Integr Plant Biol / 年: 2021タイトル: A unique photosystem I reaction center from a chlorophyll d-containing cyanobacterium Acaryochloris marina. 著者: Caihuang Xu / Qingjun Zhu / Jing-Hua Chen / Liangliang Shen / Xiaohan Yi / Zihui Huang / Wenda Wang / Min Chen / Tingyun Kuang / Jian-Ren Shen / Xing Zhang / Guangye Han /    要旨: Photosystem I (PSI) is a large protein supercomplex that catalyzes the light-dependent oxidation of plastocyanin (or cytochrome c ) and the reduction of ferredoxin. This catalytic reaction is ...Photosystem I (PSI) is a large protein supercomplex that catalyzes the light-dependent oxidation of plastocyanin (or cytochrome c ) and the reduction of ferredoxin. This catalytic reaction is realized by a transmembrane electron transfer chain consisting of primary electron donor (a special chlorophyll (Chl) pair) and electron acceptors A , A , and three Fe S clusters, F , F , and F . Here we report the PSI structure from a Chl d-dominated cyanobacterium Acaryochloris marina at 3.3 Å resolution obtained by single-particle cryo-electron microscopy. The A. marina PSI exists as a trimer with three identical monomers. Surprisingly, the structure reveals a unique composition of electron transfer chain in which the primary electron acceptor A is composed of two pheophytin a rather than Chl a found in any other well-known PSI structures. A novel subunit Psa27 is observed in the A. marina PSI structure. In addition, 77 Chls, 13 α-carotenes, two phylloquinones, three Fe-S clusters, two phosphatidyl glycerols, and one monogalactosyl-diglyceride were identified in each PSI monomer. Our results provide a structural basis for deciphering the mechanism of photosynthesis in a PSI complex with Chl d as the dominating pigments and absorbing far-red light. | |||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7dwq.cif.gz 7dwq.cif.gz | 461.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7dwq.ent.gz pdb7dwq.ent.gz | 386.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7dwq.json.gz 7dwq.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7dwq_validation.pdf.gz 7dwq_validation.pdf.gz | 6.2 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7dwq_full_validation.pdf.gz 7dwq_full_validation.pdf.gz | 6.6 MB | 表示 | |
| XML形式データ |  7dwq_validation.xml.gz 7dwq_validation.xml.gz | 106.1 KB | 表示 | |
| CIF形式データ |  7dwq_validation.cif.gz 7dwq_validation.cif.gz | 139.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/dw/7dwq https://data.pdbj.org/pub/pdb/validation_reports/dw/7dwq ftp://data.pdbj.org/pub/pdb/validation_reports/dw/7dwq ftp://data.pdbj.org/pub/pdb/validation_reports/dw/7dwq | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-Photosystem I protein ... , 4種, 4分子 DFLW
| #1: タンパク質 | 分子量: 15114.146 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Acaryochloris marina MBIC11017 (バクテリア) Acaryochloris marina MBIC11017 (バクテリア)株: MBIC 11017 / 参照: UniProt: B0C8F1 |
|---|---|
| #6: タンパク質 | 分子量: 17844.621 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Acaryochloris marina MBIC11017 (バクテリア) Acaryochloris marina MBIC11017 (バクテリア)株: MBIC 11017 / 参照: UniProt: B0C7S7 |
| #8: タンパク質 | 分子量: 15349.588 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Acaryochloris marina MBIC 11017 (バクテリア) Acaryochloris marina MBIC 11017 (バクテリア)株: MBIC 11017 / 参照: UniProt: B0C7S4 |
| #10: タンパク質・ペプチド | 分子量: 3648.337 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Acaryochloris marina MBIC11017 (バクテリア) Acaryochloris marina MBIC11017 (バクテリア)株: MBIC 11017 |
-Photosystem I P740 chlorophyll a apoprotein ... , 2種, 2分子 AB
| #2: タンパク質 | 分子量: 83470.539 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Acaryochloris marina MBIC11017 (バクテリア) Acaryochloris marina MBIC11017 (バクテリア)株: MBIC 11017 / 参照: UniProt: B0C474, photosystem I |
|---|---|
| #3: タンパク質 | 分子量: 82210.555 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Acaryochloris marina MBIC11017 (バクテリア) Acaryochloris marina MBIC11017 (バクテリア)株: MBIC 11017 / 参照: UniProt: B0C475, photosystem I |
-タンパク質 , 1種, 1分子 C
| #4: タンパク質 | 分子量: 8825.207 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Acaryochloris marinan MBIC11017 (バクテリア) Acaryochloris marinan MBIC11017 (バクテリア)株: MBIC 11017 / 参照: UniProt: B0CB42, photosystem I |
|---|
-Photosystem I reaction center subunit ... , 3種, 3分子 EJM
| #5: タンパク質 | 分子量: 9560.822 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Acaryochloris marina MBIC11017 (バクテリア) Acaryochloris marina MBIC11017 (バクテリア)株: MBIC 11017 / 参照: UniProt: B0C5D5 |
|---|---|
| #7: タンパク質 | 分子量: 5900.939 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Acaryochloris marina MBIC 11017 (バクテリア) Acaryochloris marina MBIC 11017 (バクテリア)株: MBIC 11017 / 参照: UniProt: B0C7S6 |
| #9: タンパク質・ペプチド | 分子量: 3190.899 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Acaryochloris marina MBIC11017 (バクテリア) Acaryochloris marina MBIC11017 (バクテリア)株: MBIC 11017 |
-非ポリマー , 8種, 100分子 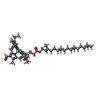



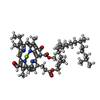

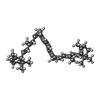








| #11: 化合物 | ChemComp-CL7 / #12: 化合物 | #13: 化合物 | #14: 化合物 | #15: 化合物 | ChemComp-G9R / | #16: 化合物 | #17: 化合物 | ChemComp-8CT / ( #18: 化合物 | ChemComp-LMG / | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|---|
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Photosystem I from a chlorophyll d-containing cyanobacterium Acaryochloris marina タイプ: COMPLEX / Entity ID: #1-#10 / 由来: NATURAL |
|---|---|
| 由来(天然) | 生物種:  Acaryochloris marina MBIC11017 (バクテリア) Acaryochloris marina MBIC11017 (バクテリア) |
| 緩衝液 | pH: 6.5 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 試料支持 | グリッドの材料: GOLD / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: Quantifoil R2/1 |
| 急速凍結 | 凍結剤: ETHANE / 湿度: 100 % |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 22500 X / 倍率(補正後): 38244 X / 最大 デフォーカス(公称値): 3000 nm / 最小 デフォーカス(公称値): 2500 nm / Calibrated defocus min: 2500 nm / 最大 デフォーカス(補正後): 3000 nm / Cs: 2.7 mm / C2レンズ絞り径: 70 µm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 平均露光時間: 10 sec. / 電子線照射量: 47 e/Å2 / 検出モード: SUPER-RESOLUTION フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) |
| 画像スキャン | 横: 3710 / 縦: 3838 / 動画フレーム数/画像: 4 / 利用したフレーム数/画像: 1-40 |
- 解析
解析
| ソフトウェア | 名称: PHENIX / バージョン: 1.19.1_4122: / 分類: 精密化 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||
| CTF補正 | タイプ: NONE | ||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 1523356 / 詳細: raw particles | ||||||||||||||||||||||||
| 対称性 | 点対称性: C3 (3回回転対称) | ||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 240880 / クラス平均像の数: 1 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj
















