登録情報 データベース : PDB / ID : 7bcbタイトル Crystal structure of the HTH DNA binding protein ArdK from R388 plasmid bound to IR3 DNA DNA (5'-D(*TP*AP*AP*TP*GP*TP*CP*AP*AP*AP*TP*AP*TP*TP*GP*AP*CP*A)-3')DNA (5'-D(*TP*GP*TP*CP*AP*AP*TP*AP*TP*TP*TP*GP*AP*CP*AP*TP*TP*A)-3')KORA domain-containing protein キーワード / / / 機能・相同性 ドメイン・相同性 構成要素
/ / / / / / / / / 生物種 Escherichia coli K-12 (大腸菌)手法 / / / 解像度 : 2.8 Å データ登録者 Fernandez-Lopez, R. / Boer, D.R. / Moncalian, G. 資金援助 組織 認可番号 国 Spanish Ministry of Economy and Competitiveness BFU2014-55534-C2-2-P Spanish Ministry of Science, Innovation, and Universities PGC2018-093885-B-I00
ジャーナル : Nucleic Acids Res. / 年 : 2022タイトル : Structural basis of direct and inverted DNA sequence repeat recognition by helix-turn-helix transcription factors.著者 : Fernandez-Lopez, R. / Ruiz, R. / Del Campo, I. / Gonzalez-Montes, L. / Boer, D.R. / de la Cruz, F. / Moncalian, G. 履歴 登録 2020年12月19日 登録サイト / 処理サイト 改定 1.0 2022年1月12日 Provider / タイプ 改定 2.0 2022年7月20日 Group Advisory / Atomic model ... Advisory / Atomic model / Data collection / Database references / Derived calculations / Other / Refinement description / Structure summary カテゴリ atom_site / cell ... atom_site / cell / ndb_struct_na_base_pair / ndb_struct_na_base_pair_step / pdbx_contact_author / pdbx_validate_close_contact / pdbx_validate_rmsd_angle / pdbx_validate_torsion / refine / refine_hist / refine_ls_restr / refine_ls_shell / reflns / software / struct_conf / struct_conn / struct_ref_seq_dif / symmetry Item _atom_site.B_iso_or_equiv / _atom_site.Cartn_x ... _atom_site.B_iso_or_equiv / _atom_site.Cartn_x / _atom_site.Cartn_y / _atom_site.Cartn_z / _cell.volume / _ndb_struct_na_base_pair.buckle / _ndb_struct_na_base_pair.hbond_type_28 / _ndb_struct_na_base_pair.opening / _ndb_struct_na_base_pair.propeller / _ndb_struct_na_base_pair.shear / _ndb_struct_na_base_pair.stagger / _ndb_struct_na_base_pair.stretch / _ndb_struct_na_base_pair_step.helical_rise / _ndb_struct_na_base_pair_step.helical_twist / _ndb_struct_na_base_pair_step.inclination / _ndb_struct_na_base_pair_step.rise / _ndb_struct_na_base_pair_step.roll / _ndb_struct_na_base_pair_step.shift / _ndb_struct_na_base_pair_step.slide / _ndb_struct_na_base_pair_step.tilt / _ndb_struct_na_base_pair_step.tip / _ndb_struct_na_base_pair_step.twist / _ndb_struct_na_base_pair_step.x_displacement / _ndb_struct_na_base_pair_step.y_displacement / _refine.B_iso_max / _refine.B_iso_mean / _refine.B_iso_min / _refine.ls_R_factor_R_free / _refine.ls_R_factor_R_work / _refine.ls_R_factor_obs / _refine.ls_d_res_low / _refine.ls_number_reflns_R_free / _refine.ls_number_reflns_R_work / _refine.ls_number_reflns_obs / _refine.ls_percent_reflns_R_free / _refine.ls_percent_reflns_obs / _refine.overall_SU_ML / _refine.pdbx_ls_cross_valid_method / _refine.pdbx_overall_phase_error / _refine.pdbx_solvent_vdw_probe_radii / _refine.pdbx_stereochemistry_target_values / _refine_hist.cycle_id / _refine_hist.d_res_low / _refine_hist.pdbx_number_residues_total / _refine_ls_shell.R_factor_R_free / _refine_ls_shell.R_factor_R_free_error / _refine_ls_shell.R_factor_R_work / _refine_ls_shell.d_res_high / _refine_ls_shell.d_res_low / _refine_ls_shell.number_reflns_R_free / _refine_ls_shell.number_reflns_R_work / _refine_ls_shell.number_reflns_all / _refine_ls_shell.pdbx_total_number_of_bins_used / _refine_ls_shell.percent_reflns_obs / _reflns.B_iso_Wilson_estimate / _software.version / _struct_conf.beg_auth_asym_id / _struct_conf.beg_auth_comp_id / _struct_conf.beg_auth_seq_id / _struct_conf.beg_label_asym_id / _struct_conf.beg_label_comp_id / _struct_conf.beg_label_seq_id / _struct_conf.end_auth_asym_id / _struct_conf.end_auth_comp_id / _struct_conf.end_auth_seq_id / _struct_conf.end_label_asym_id / _struct_conf.end_label_comp_id / _struct_conf.end_label_seq_id / _struct_conf.pdbx_PDB_helix_class / _struct_conf.pdbx_PDB_helix_length / _struct_ref_seq_dif.details / _symmetry.space_group_name_Hall 解説 / Provider / タイプ 改定 2.1 2022年11月23日 Group / カテゴリ / citation_authorItem _citation.country / _citation.journal_abbrev ... _citation.country / _citation.journal_abbrev / _citation.journal_id_ASTM / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year 改定 2.2 2022年12月14日 Group / カテゴリ / citation_authorItem _citation.journal_volume / _citation.page_first ... _citation.journal_volume / _citation.page_first / _citation.page_last / _citation_author.identifier_ORCID 改定 2.3 2024年1月31日 Group / Refinement descriptionカテゴリ / chem_comp_bond / pdbx_initial_refinement_model
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.8 Å
分子置換 / 解像度: 2.8 Å  データ登録者
データ登録者 スペイン, 2件
スペイン, 2件  引用
引用 ジャーナル: Nucleic Acids Res. / 年: 2022
ジャーナル: Nucleic Acids Res. / 年: 2022 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 7bcb.cif.gz
7bcb.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb7bcb.ent.gz
pdb7bcb.ent.gz PDB形式
PDB形式 7bcb.json.gz
7bcb.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 7bcb_validation.pdf.gz
7bcb_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 7bcb_full_validation.pdf.gz
7bcb_full_validation.pdf.gz 7bcb_validation.xml.gz
7bcb_validation.xml.gz 7bcb_validation.cif.gz
7bcb_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/bc/7bcb
https://data.pdbj.org/pub/pdb/validation_reports/bc/7bcb ftp://data.pdbj.org/pub/pdb/validation_reports/bc/7bcb
ftp://data.pdbj.org/pub/pdb/validation_reports/bc/7bcb リンク
リンク 集合体
集合体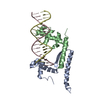
 要素
要素



 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ALBA
ALBA  / ビームライン: XALOC / 波長: 0.9792 Å
/ ビームライン: XALOC / 波長: 0.9792 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj






































