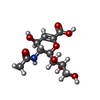
| 登録情報 | データベース: PDB / ID: 7a54
|
|---|
| タイトル | Two copies of the catalytic domain of NanA sialidase from Streptococcus pneumoniae juxtaposed in the P212121 space group, in complex with DANA |
|---|
 要素 要素 | Sialidase A |
|---|
 キーワード キーワード | STRUCTURAL PROTEIN / Sialidase / Catalytic domain / DANA |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
ganglioside catabolic process / oligosaccharide catabolic process / exo-alpha-sialidase / exo-alpha-sialidase activity / intracellular membrane-bounded organelle / extracellular region / membrane / cytoplasm類似検索 - 分子機能 : / Glycoside hydrolase, family 33, N-terminal / Trans-sialidase, domain 3 / Sialidase, N-terminal domain / BNR repeat-like domain / Laminin G domain / Laminin G domain / Sialidase family / Sialidase / YSIRK type signal peptide ...: / Glycoside hydrolase, family 33, N-terminal / Trans-sialidase, domain 3 / Sialidase, N-terminal domain / BNR repeat-like domain / Laminin G domain / Laminin G domain / Sialidase family / Sialidase / YSIRK type signal peptide / YSIRK Gram-positive signal peptide / Gram-positive cocci surface proteins LPxTG motif profile. / LPXTG cell wall anchor domain / Sialidase superfamily / Concanavalin A-like lectin/glucanase domain superfamily類似検索 - ドメイン・相同性 2-DEOXY-2,3-DEHYDRO-N-ACETYL-NEURAMINIC ACID / Sialidase A類似検索 - 構成要素 |
|---|
| 生物種 |   Streptococcus pneumoniae (肺炎レンサ球菌) Streptococcus pneumoniae (肺炎レンサ球菌) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.7 Å 分子置換 / 解像度: 2.7 Å |
|---|
 データ登録者 データ登録者 | Bridot, C. / Bouckaert, J. |
|---|
| 資金援助 |  フランス, 1件 フランス, 1件 | 組織 | 認可番号 | 国 |
|---|
| French National Research Agency | ANR-17-CE07-0028-03 |  フランス フランス |
|
|---|
 引用 引用 |  ジャーナル: Chemistry / 年: 2021タイトル ジャーナル: Chemistry / 年: 2021タイトル: Polyvalent Transition-State Analogues of Sialyl Substrates Strongly Inhibit Bacterial Sialidases*. 著者: Assailly, C. / Bridot, C. / Saumonneau, A. / Lottin, P. / Roubinet, B. / Krammer, E.M. / Francois, F. / Vena, F. / Landemarre, L. / Alvarez Dorta, D. / Deniaud, D. / Grandjean, C. / Tellier, ...著者: Assailly, C. / Bridot, C. / Saumonneau, A. / Lottin, P. / Roubinet, B. / Krammer, E.M. / Francois, F. / Vena, F. / Landemarre, L. / Alvarez Dorta, D. / Deniaud, D. / Grandjean, C. / Tellier, C. / Pascual, S. / Montembault, V. / Fontaine, L. / Daligault, F. / Bouckaert, J. / Gouin, S.G.#1: ジャーナル: Chemistry / 年: 2019タイトル: Multivalent Thiosialosides and Their Synergistic Interaction with Pathogenic Sialidases. 著者: Brissonnet, Y. / Assailly, C. / Saumonneau, A. / Bouckaert, J. / Maillasson, M. / Petitot, C. / Roubinet, B. / Didak, B. / Landemarre, L. / Bridot, C. / Blossey, R. / Deniaud, D. / Yan, X. / ...著者: Brissonnet, Y. / Assailly, C. / Saumonneau, A. / Bouckaert, J. / Maillasson, M. / Petitot, C. / Roubinet, B. / Didak, B. / Landemarre, L. / Bridot, C. / Blossey, R. / Deniaud, D. / Yan, X. / Bernard, J. / Tellier, C. / Grandjean, C. / Daligault, F. / Gouin, S.G. |
|---|
| 履歴 | | 登録 | 2020年8月20日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2020年11月18日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2021年3月3日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.journal_volume / _citation.page_first ..._citation.journal_volume / _citation.page_first / _citation.page_last / _citation.title / _citation.year / _citation_author.name |
|---|
| 改定 1.2 | 2024年1月31日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.7 Å
分子置換 / 解像度: 2.7 Å  データ登録者
データ登録者 フランス, 1件
フランス, 1件  引用
引用 ジャーナル: Chemistry / 年: 2021
ジャーナル: Chemistry / 年: 2021 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 7a54.cif.gz
7a54.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb7a54.ent.gz
pdb7a54.ent.gz PDB形式
PDB形式 7a54.json.gz
7a54.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 7a54_validation.pdf.gz
7a54_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 7a54_full_validation.pdf.gz
7a54_full_validation.pdf.gz 7a54_validation.xml.gz
7a54_validation.xml.gz 7a54_validation.cif.gz
7a54_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/a5/7a54
https://data.pdbj.org/pub/pdb/validation_reports/a5/7a54 ftp://data.pdbj.org/pub/pdb/validation_reports/a5/7a54
ftp://data.pdbj.org/pub/pdb/validation_reports/a5/7a54 リンク
リンク 集合体
集合体


 要素
要素

 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SOLEIL
SOLEIL  / ビームライン: PROXIMA 1 / 波長: 0.987 Å
/ ビームライン: PROXIMA 1 / 波長: 0.987 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj