| ソフトウェア | | 名称 | バージョン | 分類 | NB |
|---|
| PHENIX | 1.12_2829| 精密化 | | | XDS | | データ削減 | | | XSCALE | | データスケーリング | | | PHASER | | 位相決定 | | | PDB_EXTRACT | 3.25 | データ抽出 | | |
|
|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換
開始モデル: Modeler
解像度: 2.124→47.691 Å / SU ML: 0.26 / 交差検証法: THROUGHOUT / σ(F): 1.35 / 位相誤差: 24.02 / 立体化学のターゲット値: ML
| Rfactor | 反射数 | %反射 |
|---|
| Rfree | 0.2433 | 612 | 5.01 % |
|---|
| Rwork | 0.1854 | 11615 | - |
|---|
| obs | 0.1882 | 12227 | 99.85 % |
|---|
|
|---|
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL |
|---|
| 原子変位パラメータ | Biso max: 142.54 Å2 / Biso mean: 53.3997 Å2 / Biso min: 24.37 Å2 |
|---|
| 精密化ステップ | サイクル: final / 解像度: 2.124→47.691 Å
| タンパク質 | 核酸 | リガンド | 溶媒 | 全体 |
|---|
| 原子数 | 1357 | 0 | 27 | 97 | 1481 |
|---|
| Biso mean | - | - | 98.66 | 52.51 | - |
|---|
| 残基数 | - | - | - | - | 176 |
|---|
|
|---|
| 拘束条件 | | Refine-ID | タイプ | Dev ideal | 数 |
|---|
| X-RAY DIFFRACTION | f_bond_d| 0.008 | 1407 | | X-RAY DIFFRACTION | f_angle_d| 0.872 | 1899 | | X-RAY DIFFRACTION | f_chiral_restr| 0.053 | 207 | | X-RAY DIFFRACTION | f_plane_restr| 0.005 | 244 | | X-RAY DIFFRACTION | f_dihedral_angle_d| 6.336 | 1021 | | | | | |
|
|---|
| LS精密化 シェル | Refine-ID: X-RAY DIFFRACTION / Rfactor Rfree error: 0 | 解像度 (Å) | Rfactor Rfree | Num. reflection Rfree | Rfactor Rwork | Num. reflection Rwork | % reflection obs (%) |
|---|
| 2.124-2.3376 | 0.2864 | 148 | 0.2239 | 2799 | 99 | | 2.3376-2.6759 | 0.2686 | 150 | 0.221 | 2853 | 100 | | 2.6759-3.3712 | 0.2718 | 152 | 0.1985 | 2889 | 100 |
|
|---|
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION | ID | L11 (°2) | L12 (°2) | L13 (°2) | L22 (°2) | L23 (°2) | L33 (°2) | S11 (Å °) | S12 (Å °) | S13 (Å °) | S21 (Å °) | S22 (Å °) | S23 (Å °) | S31 (Å °) | S32 (Å °) | S33 (Å °) | T11 (Å2) | T12 (Å2) | T13 (Å2) | T22 (Å2) | T23 (Å2) | T33 (Å2) | Origin x (Å) | Origin y (Å) | Origin z (Å) |
|---|
| 1 | 1.9596 | 0.3959 | -0.2964 | 1.7018 | -0.531 | 5.5119 | 0.0258 | -0.1408 | 0.0188 | 0.0547 | -0.0291 | -0.1539 | 0.0922 | 0.2712 | 0.0207 | 0.2545 | 0.0143 | -0.0262 | 0.264 | -0.0068 | 0.3005 | 21.7445 | 105.169 | 127.942 | | 2 | 4.5392 | 3.5201 | -4.1184 | 2.864 | -3.231 | 3.8857 | 0.2486 | -0.9306 | -0.3944 | 0.4823 | -0.031 | -0.7814 | -0.3061 | 1.0166 | -0.4116 | 0.4707 | -0.0463 | -0.0839 | 0.6472 | -0.0907 | 0.5293 | 30.427 | 109.4009 | 149.5904 | | 3 | 4.3022 | 0.0731 | -4.8085 | 5.8289 | 0.3176 | 5.4233 | 0.0805 | 0.2946 | 0.2842 | -0.1302 | -0.0657 | -0.3725 | -0.3731 | 0.6482 | -0.0388 | 0.4341 | -0.029 | -0.1066 | 0.7484 | -0.0837 | 0.3807 | 28.082 | 108.5636 | 141.3123 |
|
|---|
| 精密化 TLSグループ | | ID | Refine-ID | Refine TLS-ID | Selection details | Auth asym-ID | Auth seq-ID |
|---|
| 1 | X-RAY DIFFRACTION | 1 | (chain 'A' and resid 1 through 121)A| 1 - 121 | | 2 | X-RAY DIFFRACTION | 2 | (chain 'B' and resid 0 through 26)B| 0 - 26 | | 3 | X-RAY DIFFRACTION | 3 | (chain 'C' and resid 0 through 27)C| 0 - 27 | | | | | | |
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報

 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.124 Å
分子置換 / 解像度: 2.124 Å  データ登録者
データ登録者 スロベニア, 3件
スロベニア, 3件  引用
引用 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2021
ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2021 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 7a4t.cif.gz
7a4t.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb7a4t.ent.gz
pdb7a4t.ent.gz PDB形式
PDB形式 7a4t.json.gz
7a4t.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/a4/7a4t
https://data.pdbj.org/pub/pdb/validation_reports/a4/7a4t ftp://data.pdbj.org/pub/pdb/validation_reports/a4/7a4t
ftp://data.pdbj.org/pub/pdb/validation_reports/a4/7a4t リンク
リンク 集合体
集合体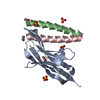

 要素
要素









 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SOLEIL
SOLEIL  / ビームライン: PROXIMA 2 / 波長: 0.980127 Å
/ ビームライン: PROXIMA 2 / 波長: 0.980127 Å 分子置換
分子置換 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj






