+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6xr4 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
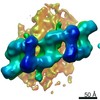
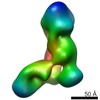
| タイトル | Integrative in situ structure of Parkinsons disease-linked human LRRK2 | |||||||||
 要素 要素 | Leucine-rich repeat serine/threonine-protein kinase 2 | |||||||||
 キーワード キーワード | SIGNALING PROTEIN / TRANSFERASE / kinase / GTPase / Parkinson's Disease / pseudo-kinase | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報caveola neck / negative regulation of protein processing involved in protein targeting to mitochondrion / Wnt signalosome assembly / beta-catenin destruction complex binding / regulation of branching morphogenesis of a nerve / regulation of kidney size / regulation of cell projection organization / tangential migration from the subventricular zone to the olfactory bulb / protein localization to endoplasmic reticulum exit site / GTP-dependent protein kinase activity ...caveola neck / negative regulation of protein processing involved in protein targeting to mitochondrion / Wnt signalosome assembly / beta-catenin destruction complex binding / regulation of branching morphogenesis of a nerve / regulation of kidney size / regulation of cell projection organization / tangential migration from the subventricular zone to the olfactory bulb / protein localization to endoplasmic reticulum exit site / GTP-dependent protein kinase activity / regulation of SNARE complex assembly / regulation of neuroblast proliferation / regulation of ER to Golgi vesicle-mediated transport / peroxidase inhibitor activity / negative regulation of late endosome to lysosome transport / regulation of mitochondrial depolarization / negative regulation of protein targeting to mitochondrion / positive regulation of dopamine receptor signaling pathway / amphisome / regulation of synaptic vesicle transport / regulation of CAMKK-AMPK signaling cascade / regulation of lysosomal lumen pH / co-receptor binding / negative regulation of GTPase activity / regulation of dopamine receptor signaling pathway / mitochondrion localization / regulation of neuron maturation / positive regulation of microglial cell activation / regulation of retrograde transport, endosome to Golgi / positive regulation of synaptic vesicle endocytosis / cytoplasmic side of mitochondrial outer membrane / negative regulation of excitatory postsynaptic potential / negative regulation of autophagosome assembly / JUN kinase kinase kinase activity / olfactory bulb development / neuron projection arborization / multivesicular body, internal vesicle / striatum development / regulation of dendritic spine morphogenesis / protein localization to mitochondrion / cellular response to dopamine / positive regulation of mitochondrial outer membrane permeabilization involved in apoptotic signaling pathway / endoplasmic reticulum organization / positive regulation of protein autoubiquitination / Wnt signalosome / positive regulation of programmed cell death / negative regulation of protein processing / GTP metabolic process / regulation of canonical Wnt signaling pathway / syntaxin-1 binding / regulation of reactive oxygen species metabolic process / lysosome organization / Golgi-associated vesicle / clathrin binding / protein kinase A binding / PTK6 promotes HIF1A stabilization / regulation of locomotion / negative regulation of macroautophagy / neuromuscular junction development / regulation of cAMP/PKA signal transduction / regulation of mitochondrial fission / Golgi organization / regulation of synaptic vesicle exocytosis / exploration behavior / microvillus / intracellular distribution of mitochondria / endoplasmic reticulum exit site / autolysosome / locomotory exploration behavior / negative regulation of Notch signaling pathway / MAP kinase kinase kinase activity / regulation of synaptic vesicle endocytosis / canonical Wnt signaling pathway / regulation of synaptic transmission, glutamatergic / negative regulation of endoplasmic reticulum stress-induced intrinsic apoptotic signaling pathway / presynaptic cytosol / Rho protein signal transduction / phagocytic vesicle / neuron projection morphogenesis / JNK cascade / cellular response to manganese ion / positive regulation of autophagy / tubulin binding / dendrite cytoplasm / GTPase activator activity / cellular response to starvation / positive regulation of protein ubiquitination / SNARE binding / determination of adult lifespan / cellular response to reactive oxygen species / regulation of membrane potential / excitatory postsynaptic potential / mitochondrion organization / trans-Golgi network / calcium-mediated signaling / mitochondrial membrane / regulation of protein stability / small GTPase binding / autophagy / endocytosis 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 電子顕微鏡法 / サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 14 Å | |||||||||
 データ登録者 データ登録者 | Villa, E. / Lasker, K. / Audagnotto, M. | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2020 ジャーナル: Cell / 年: 2020タイトル: The In Situ Structure of Parkinson's Disease-Linked LRRK2. 著者: Reika Watanabe / Robert Buschauer / Jan Böhning / Martina Audagnotto / Keren Lasker / Tsan-Wen Lu / Daniela Boassa / Susan Taylor / Elizabeth Villa /  要旨: Mutations in leucine-rich repeat kinase 2 (LRRK2) are the most frequent cause of familial Parkinson's disease. LRRK2 is a multi-domain protein containing a kinase and GTPase. Using correlative light ...Mutations in leucine-rich repeat kinase 2 (LRRK2) are the most frequent cause of familial Parkinson's disease. LRRK2 is a multi-domain protein containing a kinase and GTPase. Using correlative light and electron microscopy, in situ cryo-electron tomography, and subtomogram analysis, we reveal a 14-Å structure of LRRK2 bearing a pathogenic mutation that oligomerizes as a right-handed double helix around microtubules, which are left-handed. Using integrative modeling, we determine the architecture of LRRK2, showing that the GTPase and kinase are in close proximity, with the GTPase closer to the microtubule surface, whereas the kinase is exposed to the cytoplasm. We identify two oligomerization interfaces mediated by non-catalytic domains. Mutation of one of these abolishes LRRK2 microtubule-association. Our work demonstrates the power of cryo-electron tomography to generate models of previously unsolved structures in their cellular environment. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6xr4.cif.gz 6xr4.cif.gz | 3.6 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6xr4.ent.gz pdb6xr4.ent.gz | 2.4 MB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6xr4.json.gz 6xr4.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6xr4_validation.pdf.gz 6xr4_validation.pdf.gz | 2.7 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6xr4_full_validation.pdf.gz 6xr4_full_validation.pdf.gz | 2.8 MB | 表示 | |
| XML形式データ |  6xr4_validation.xml.gz 6xr4_validation.xml.gz | 59.4 KB | 表示 | |
| CIF形式データ |  6xr4_validation.cif.gz 6xr4_validation.cif.gz | 1005.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/xr/6xr4 https://data.pdbj.org/pub/pdb/validation_reports/xr/6xr4 ftp://data.pdbj.org/pub/pdb/validation_reports/xr/6xr4 ftp://data.pdbj.org/pub/pdb/validation_reports/xr/6xr4 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
| モデル数 | 53 |
- 要素
要素
| #1: タンパク質 | 分子量: 286450.719 Da / 分子数: 2 / 変異: I2020T / 由来タイプ: 天然 / 由来: (天然)  Homo sapiens (ヒト) / 細胞株: HEK293 Homo sapiens (ヒト) / 細胞株: HEK293参照: UniProt: Q5S007, non-specific serine/threonine protein kinase, 加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: CELL / 3次元再構成法: サブトモグラム平均法 |
- 試料調製
試料調製
| 構成要素 | 名称: HEK293 cell / タイプ: CELL 詳細: Dimer within the oligomer of microtubule-bound human LRRK2-I2020T in HEK cells Entity ID: all / 由来: NATURAL |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / Cell: HEK293 / 細胞内の位置: cytosol Homo sapiens (ヒト) / Cell: HEK293 / 細胞内の位置: cytosol |
| 緩衝液 | pH: 7 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES 詳細: FIB-milled cellular samples expressing Parkinson's disease-related mutant LRRK2 (I2020T) proteins in human embryonic kidney cells (HEK293 cells) |
| 試料支持 | グリッドの材料: GOLD / グリッドのサイズ: 200 divisions/in. / グリッドのタイプ: Quantifoil |
| 急速凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI POLARA 300 |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD |
| 撮影 | 電子線照射量: 2 e/Å2 / 検出モード: COUNTING フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) |
| 電子光学装置 | エネルギーフィルター名称: GIF Quantum SE / エネルギーフィルタースリット幅: 20 eV |
- 解析
解析
| EMソフトウェア |
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING ONLY | |||||||||||||||
| 対称性 | 点対称性: C1 (非対称) | |||||||||||||||
| 3次元再構成 | 解像度: 14 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 4307 詳細: Each model in the conformation ensemble represents a cluster. Models are arranged sequentially from lowest to highest cluster RMSD. Model 1 is the best representative model. Model-Cluster ...詳細: Each model in the conformation ensemble represents a cluster. Models are arranged sequentially from lowest to highest cluster RMSD. Model 1 is the best representative model. Model-Cluster correpondence is as follows: Model 1 = Cluster 20 Model 2 = Cluster 16 Model 3 = Cluster 24 Model 4 = Cluster 32 Model 5 = Cluster 14 Model 6 = Cluster 1 Model 7 = Cluster 44 Model 8 = Cluster 11 Model 9 = Cluster 3 Model 10 = Cluster 22 Model 11 = Cluster 4 Model 12 = Cluster 12 Model 13 = Cluster 19 Model 14 = Cluster 7 Model 15 = Cluster 31 Model 16 = Cluster 26 Model 17 = Cluster 52 Model 18 = Cluster 2 Model 19 = Cluster 45 Model 20 = Cluster 8 Model 21 = Cluster 5 Model 22 = Cluster 23 Model 23 = Cluster 42 Model 24 = Cluster 25 Model 25 = Cluster 43 Model 26 = Cluster 27 Model 27 = Cluster 39 Model 28 = Cluster 6 Model 29 = Cluster 13 Model 30 = Cluster 28 Model 31 = Cluster 21 Model 32 = Cluster 10 Model 33 = Cluster 53 Model 34 = Cluster 30 Model 35 = Cluster 35 Model 36 = Cluster 38 Model 37 = Cluster 33 Model 38 = Cluster 40 Model 39 = Cluster 37 Model 40 = Cluster 29 Model 41 = Cluster 51 Model 42 = Cluster 41 Model 43 = Cluster 15 Model 44 = Cluster 9 Model 45 = Cluster 17 Model 46 = Cluster 50 Model 47 = Cluster 48 Model 48 = Cluster 49 Model 49 = Cluster 18 Model 50 = Cluster 46 Model 51 = Cluster 36 Model 52 = Cluster 47 Model 53 = Cluster 34 対称性のタイプ: POINT | |||||||||||||||
| EM volume selection | Num. of tomograms: 12 / Num. of volumes extracted: 11508 | |||||||||||||||
| 原子モデル構築 | プロトコル: OTHER | |||||||||||||||
| 精密化 | 詳細: Each model in the conformation ensemble represents a cluster. Models are arranged sequentially from lowest to highest cluster RMSD. Model 1 is the best representative model. Model-Cluster ...詳細: Each model in the conformation ensemble represents a cluster. Models are arranged sequentially from lowest to highest cluster RMSD. Model 1 is the best representative model. Model-Cluster correpondence is as follows: Model 1 = Cluster 20 Model 2 = Cluster 16 Model 3 = Cluster 24 Model 4 = Cluster 32 Model 5 = Cluster 14 Model 6 = Cluster 1 Model 7 = Cluster 44 Model 8 = Cluster 11 Model 9 = Cluster 3 Model 10 = Cluster 22 Model 11 = Cluster 4 Model 12 = Cluster 12 Model 13 = Cluster 19 Model 14 = Cluster 7 Model 15 = Cluster 31 Model 16 = Cluster 26 Model 17 = Cluster 52 Model 18 = Cluster 2 Model 19 = Cluster 45 Model 20 = Cluster 8 Model 21 = Cluster 5 Model 22 = Cluster 23 Model 23 = Cluster 42 Model 24 = Cluster 25 Model 25 = Cluster 43 Model 26 = Cluster 27 Model 27 = Cluster 39 Model 28 = Cluster 6 Model 29 = Cluster 13 Model 30 = Cluster 28 Model 31 = Cluster 21 Model 32 = Cluster 10 Model 33 = Cluster 53 Model 34 = Cluster 30 Model 35 = Cluster 35 Model 36 = Cluster 38 Model 37 = Cluster 33 Model 38 = Cluster 40 Model 39 = Cluster 37 Model 40 = Cluster 29 Model 41 = Cluster 51 Model 42 = Cluster 41 Model 43 = Cluster 15 Model 44 = Cluster 9 Model 45 = Cluster 17 Model 46 = Cluster 50 Model 47 = Cluster 48 Model 48 = Cluster 49 Model 49 = Cluster 18 Model 50 = Cluster 46 Model 51 = Cluster 36 Model 52 = Cluster 47 Model 53 = Cluster 34 |
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj







