| #1: タンパク質 | Galactoside permease / Lactose permease / Lactose permease (Lactose-proton symport) / Lactose-proton symport / MFS ...Lactose permease / Lactose permease (Lactose-proton symport) / Lactose-proton symport / MFS transporter / NorA_1_M97169
分子量: 46789.207 Da / 分子数: 2 / 変異: G46W, G262W / 由来タイプ: 組換発現 / 由来: (組換発現)   Escherichia coli (大腸菌)遺伝子: lacY, lacY_1, lacY_2, lacY_3, A6V01_11495, A8C65_20655, A8M42_15285, ACN68_10080, ACN81_29445, ACU57_18760, AJ318_07830, AKG99_15095, AM270_02570, AM464_18655, AMK83_26120, AML07_16950, ...遺伝子: lacY, lacY_1, lacY_2, lacY_3, A6V01_11495, A8C65_20655, A8M42_15285, ACN68_10080, ACN81_29445, ACU57_18760, AJ318_07830, AKG99_15095, AM270_02570, AM464_18655, AMK83_26120, AML07_16950, AML35_24485, APU18_15875, APZ14_02895, AUQ13_18775, AUS26_08045, AW059_14270, AW106_12030, AWF59_002435, AWG78_006090, AZZ83_000123, B9M99_04475, B9T59_06225, BANRA_00777, BB545_15300, BHF46_20715, BIQ87_01935, BIU72_03550, BK373_01540, BK375_21010, BMT49_03020, BMT91_19335, BOH76_22050, BON66_09310, BON69_11830, BON71_09435, BON92_03420, BON94_14610, BON95_06740, BON96_22525, BUE81_15270, BvCms12BK_03300, BvCms2454_01726, BvCms28BK_00743, BvCmsA75A_04728, BvCmsHHP001_01494, BvCmsHHP019_04502, BvCmsHHP056_04283, BvCmsKKNP011_04619, BvCmsKKP036_04325, BvCmsKSNP019_03228, BvCmsKSNP120_04320, BvCmsKSP015_03661, BvCmsKSP026_02739, BvCmsKSP058_04814, BvCmsKSP083_00737, BvCmsNSNP036_04122, BvCmsNSP006_03638, BvCmsNSP007_00895, BvCmsNSP072_03352, BvCmsSINP011_04898, BvCmsSINP012_01842, BvCmsSIP019_03585, BvCmsSIP044_04786, BVL39_01120, BW690_22770, BWI87_08790, BWI89_12765, BXT93_23905, BZL31_08595, BZL69_13855, C2U48_08360, C3449_08540, C4K41_08290, C5715_13825, C5N07_24640, C5P44_12945, C6669_02995, C6B13_09720, C7235_19125, C7B02_04785, C7B06_24440, C7B07_24140, C7B08_05950, C9025_05365, C9063_01880, C9077_04180, C9078_02330, C9083_15570, C9212_10665, C9E25_04820, C9Y80_12290, C9Y91_05615, C9Y95_06770, C9Z87_09505, CA593_01420, CDL37_09235, CI694_24695, COD30_12260, CQP61_21130, CRD98_01430, CRT46_01825, CSB64_04040, CT143_18850, CT146_24550, CV83915_01231, CWS33_24300, D1912_21130, D2183_00410, D2188_07965, D3821_13115, D3822_04615, D3C88_02070, D3Y67_20835, D6W60_16025, D9D20_17940, D9D33_20360, D9D43_15040, D9F17_03720, D9G48_18410, D9H53_20330, D9H68_14985, D9H70_12020, D9H94_11975, D9I11_08840, D9I20_07450, D9I88_07865, D9I97_15245, D9J11_01945, D9J46_05685, D9J60_08355, D9K48_13755, D9K54_21075, DAH18_14735, DAH32_05885, DAH34_13895, DAH37_22385, DBQ99_19865, DEO04_04305, DIV22_00165, DL800_03635, DM102_12510, DNQ41_05610, DNR41_19640, DNX30_04510, DNX30_30030, DP277_21630, DQF57_07430, DS966_21110, DTL43_07655, DTL90_10485, DTM10_05840, DTM25_14170, DTM45_10720, DU321_19250, DU333_15155, DXT69_23380, DXT73_20905, E2112_14055, E2119_01930, E2127_11985, E2128_04640, E2129_15490, E2134_00125, E2855_00356, E2863_00381, E5P22_06110, E5P37_17460, E5S35_17515, E5S46_09030, E5S47_10100, E5S58_11240, EAI42_17215, EAI46_16235, EAI52_25350, EB575_09280, EC1094V2_3507, EC3234A_4c00200, EC3426_01189, EC382_22725, EC95NR1_04568, ECTO6_03753, ED600_13350, ED648_10345, EIA21_12990, EJH97_18755, EKI52_09385, EL75_3407, EL79_3502, EL80_3454, ELT20_08545, ELT33_21350, ELV05_17125, ELV08_19270, ELV28_08615, EO241_13610, EPS94_20330, EPS97_16790, EPT01_22725, EQ823_11820, EQ825_16155, EQ830_03870, ERS085365_01883, ERS085379_01258, ERS085386_01050, ERS085416_03243, ERS139211_01209, ERS150876_00526, EVY14_07340, EWK56_10075, ExPECSC022_00401, ExPECSC038_04855, EXX06_12625, EXX13_09890, EXX24_01665, EXX55_12100, EXX73_02505, EXX78_04900, EXX87_11790, EYD11_17665, EYX83_13280, FNJ69_23270, FNJ83_03490, FORC28_4807, FORC82_3704, FQ915_24920, FQR64_17860, FQU83_21025, FRV13_03655, FV293_06325, FWK02_14300, GJ11_02155, HmCmsJML079_00608, HmCmsJML122_00139, HmCmsJML146_04035, HmCmsJML204_03576, HMPREF3040_03340, HW43_05365, JD73_11990, MJ49_04715, MS6198_03470, NCTC10082_01120, NCTC10090_01929, NCTC10429_03820, NCTC10766_05668, NCTC13148_06751, NCTC8179_01591, NCTC8959_04986, NCTC8960_01355, NCTC9045_04482, NCTC9050_01807, NCTC9055_00742, NCTC9058_03073, NCTC9062_04425, NCTC9077_04847, NCTC9081_02389, NCTC9111_04021, NCTC9117_04856, NCTC9119_04042, NCTC9703_03267, RG28_03130, RK56_026970, RX35_02023, SAMEA3472055_05173, SAMEA3472056_02980, SAMEA3472080_03897, SAMEA3472090_01855, SAMEA3472110_02233, SAMEA3472112_02272, SAMEA3484427_03921, SAMEA3484429_03823, SAMEA3484434_02913, SAMEA3485101_01840, SAMEA3752372_02293, SAMEA3752557_00172, SAMEA3752559_04815, SAMEA3752620_00744, SAMEA3753164_00177, SAMEA3753300_00417, UC41_01885, UN86_08680, UN91_18970, YDC107_3615プラスミド Escherichia coli (大腸菌)遺伝子: lacY, lacY_1, lacY_2, lacY_3, A6V01_11495, A8C65_20655, A8M42_15285, ACN68_10080, ACN81_29445, ACU57_18760, AJ318_07830, AKG99_15095, AM270_02570, AM464_18655, AMK83_26120, AML07_16950, ...遺伝子: lacY, lacY_1, lacY_2, lacY_3, A6V01_11495, A8C65_20655, A8M42_15285, ACN68_10080, ACN81_29445, ACU57_18760, AJ318_07830, AKG99_15095, AM270_02570, AM464_18655, AMK83_26120, AML07_16950, AML35_24485, APU18_15875, APZ14_02895, AUQ13_18775, AUS26_08045, AW059_14270, AW106_12030, AWF59_002435, AWG78_006090, AZZ83_000123, B9M99_04475, B9T59_06225, BANRA_00777, BB545_15300, BHF46_20715, BIQ87_01935, BIU72_03550, BK373_01540, BK375_21010, BMT49_03020, BMT91_19335, BOH76_22050, BON66_09310, BON69_11830, BON71_09435, BON92_03420, BON94_14610, BON95_06740, BON96_22525, BUE81_15270, BvCms12BK_03300, BvCms2454_01726, BvCms28BK_00743, BvCmsA75A_04728, BvCmsHHP001_01494, BvCmsHHP019_04502, BvCmsHHP056_04283, BvCmsKKNP011_04619, BvCmsKKP036_04325, BvCmsKSNP019_03228, BvCmsKSNP120_04320, BvCmsKSP015_03661, BvCmsKSP026_02739, BvCmsKSP058_04814, BvCmsKSP083_00737, BvCmsNSNP036_04122, BvCmsNSP006_03638, BvCmsNSP007_00895, BvCmsNSP072_03352, BvCmsSINP011_04898, BvCmsSINP012_01842, BvCmsSIP019_03585, BvCmsSIP044_04786, BVL39_01120, BW690_22770, BWI87_08790, BWI89_12765, BXT93_23905, BZL31_08595, BZL69_13855, C2U48_08360, C3449_08540, C4K41_08290, C5715_13825, C5N07_24640, C5P44_12945, C6669_02995, C6B13_09720, C7235_19125, C7B02_04785, C7B06_24440, C7B07_24140, C7B08_05950, C9025_05365, C9063_01880, C9077_04180, C9078_02330, C9083_15570, C9212_10665, C9E25_04820, C9Y80_12290, C9Y91_05615, C9Y95_06770, C9Z87_09505, CA593_01420, CDL37_09235, CI694_24695, COD30_12260, CQP61_21130, CRD98_01430, CRT46_01825, CSB64_04040, CT143_18850, CT146_24550, CV83915_01231, CWS33_24300, D1912_21130, D2183_00410, D2188_07965, D3821_13115, D3822_04615, D3C88_02070, D3Y67_20835, D6W60_16025, D9D20_17940, D9D33_20360, D9D43_15040, D9F17_03720, D9G48_18410, D9H53_20330, D9H68_14985, D9H70_12020, D9H94_11975, D9I11_08840, D9I20_07450, D9I88_07865, D9I97_15245, D9J11_01945, D9J46_05685, D9J60_08355, D9K48_13755, D9K54_21075, DAH18_14735, DAH32_05885, DAH34_13895, DAH37_22385, DBQ99_19865, DEO04_04305, DIV22_00165, DL800_03635, DM102_12510, DNQ41_05610, DNR41_19640, DNX30_04510, DNX30_30030, DP277_21630, DQF57_07430, DS966_21110, DTL43_07655, DTL90_10485, DTM10_05840, DTM25_14170, DTM45_10720, DU321_19250, DU333_15155, DXT69_23380, DXT73_20905, E2112_14055, E2119_01930, E2127_11985, E2128_04640, E2129_15490, E2134_00125, E2855_00356, E2863_00381, E5P22_06110, E5P37_17460, E5S35_17515, E5S46_09030, E5S47_10100, E5S58_11240, EAI42_17215, EAI46_16235, EAI52_25350, EB575_09280, EC1094V2_3507, EC3234A_4c00200, EC3426_01189, EC382_22725, EC95NR1_04568, ECTO6_03753, ED600_13350, ED648_10345, EIA21_12990, EJH97_18755, EKI52_09385, EL75_3407, EL79_3502, EL80_3454, ELT20_08545, ELT33_21350, ELV05_17125, ELV08_19270, ELV28_08615, EO241_13610, EPS94_20330, EPS97_16790, EPT01_22725, EQ823_11820, EQ825_16155, EQ830_03870, ERS085365_01883, ERS085379_01258, ERS085386_01050, ERS085416_03243, ERS139211_01209, ERS150876_00526, EVY14_07340, EWK56_10075, ExPECSC022_00401, ExPECSC038_04855, EXX06_12625, EXX13_09890, EXX24_01665, EXX55_12100, EXX73_02505, EXX78_04900, EXX87_11790, EYD11_17665, EYX83_13280, FNJ69_23270, FNJ83_03490, FORC28_4807, FORC82_3704, FQ915_24920, FQR64_17860, FQU83_21025, FRV13_03655, FV293_06325, FWK02_14300, GJ11_02155, HmCmsJML079_00608, HmCmsJML122_00139, HmCmsJML146_04035, HmCmsJML204_03576, HMPREF3040_03340, HW43_05365, JD73_11990, MJ49_04715, MS6198_03470, NCTC10082_01120, NCTC10090_01929, NCTC10429_03820, NCTC10766_05668, NCTC13148_06751, NCTC8179_01591, NCTC8959_04986, NCTC8960_01355, NCTC9045_04482, NCTC9050_01807, NCTC9055_00742, NCTC9058_03073, NCTC9062_04425, NCTC9077_04847, NCTC9081_02389, NCTC9111_04021, NCTC9117_04856, NCTC9119_04042, NCTC9703_03267, RG28_03130, RK56_026970, RX35_02023, SAMEA3472055_05173, SAMEA3472056_02980, SAMEA3472080_03897, SAMEA3472090_01855, SAMEA3472110_02233, SAMEA3472112_02272, SAMEA3484427_03921, SAMEA3484429_03823, SAMEA3484434_02913, SAMEA3485101_01840, SAMEA3752372_02293, SAMEA3752557_00172, SAMEA3752559_04815, SAMEA3752620_00744, SAMEA3753164_00177, SAMEA3753300_00417, UC41_01885, UN86_08680, UN91_18970, YDC107_3615プラスミド: PT7-5 / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) / Variant (発現宿主) Escherichia coli BL21(DE3) (大腸菌) / Variant (発現宿主): C41 / 参照: UniProt: C6FW78, UniProt: P02920*PLUS      | #2: 抗体 | nanobody 9043
分子量: 13451.910 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)   Lama glama (ラマ) / プラスミド Lama glama (ラマ) / プラスミド: PMESY4 / 発現宿主:   Escherichia coli (大腸菌) / 株 (発現宿主) Escherichia coli (大腸菌) / 株 (発現宿主): WK6 | #3: 多糖 | beta-D-galactopyranose-(1-1)-1-thio-beta-D-galactopyranose
タイプ: oligosaccharide / 分子量: 358.362 Da / 分子数: 2 / 由来タイプ: 組換発現 | 記述子 | タイプ | プログラム |
|---|
| WURCS=2.0/1,2,1/[a2112h-1b_1-5]/1-1/a1-b1*S* | WURCS | PDB2Glycan 1.1.0 | | [][b-D-Galp1SH]{[(1+S)][b-D-Galp]{}} | LINUCS | PDB-CARE |
| #4: 糖 |
ChemComp-BNG / nonyl beta-D-glucopyranoside / Beta-NONYLGLUCOSIDE / nonyl beta-D-glucoside / nonyl D-glucoside / nonyl glucoside
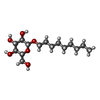  タイプ タイプ: D-saccharide / 分子量: 306.395 Da / 分子数: 9 / 由来タイプ: 組換発現 / 式: C 15H 30O 6 / コメント: 可溶化剤 *YM| 識別子 | タイプ | プログラム |
|---|
| b-nonylglucoside | IUPAC CARBOHYDRATE SYMBOL | PDB-CARE 1.0 |
| #5: 水 | ChemComp-HOH / water
 分子量 分子量: 18.015 Da / 分子数: 2 / 由来タイプ: 天然 / 式: H 2O |
|---|
| 研究の焦点であるリガンドがあるか | Y |
|---|
| Has protein modification | Y |
|---|
|
|---|
|
|---|
|
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報

 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.8 Å
分子置換 / 解像度: 2.8 Å  データ登録者
データ登録者 米国, 3件
米国, 3件  引用
引用 ジャーナル: Plos One / 年: 2020
ジャーナル: Plos One / 年: 2020 ジャーナル: Proc. Natl. Acad. Sci. U.S.A. / 年: 2018
ジャーナル: Proc. Natl. Acad. Sci. U.S.A. / 年: 2018 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6vbg.cif.gz
6vbg.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6vbg.ent.gz
pdb6vbg.ent.gz PDB形式
PDB形式 6vbg.json.gz
6vbg.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6vbg_validation.pdf.gz
6vbg_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6vbg_full_validation.pdf.gz
6vbg_full_validation.pdf.gz 6vbg_validation.xml.gz
6vbg_validation.xml.gz 6vbg_validation.cif.gz
6vbg_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/vb/6vbg
https://data.pdbj.org/pub/pdb/validation_reports/vb/6vbg ftp://data.pdbj.org/pub/pdb/validation_reports/vb/6vbg
ftp://data.pdbj.org/pub/pdb/validation_reports/vb/6vbg
 リンク
リンク 集合体
集合体


 要素
要素



 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ALS
ALS  / ビームライン: 8.3.1 / 波長: 1.115 Å
/ ビームライン: 8.3.1 / 波長: 1.115 Å 分子置換
分子置換 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj







