+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6uze | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
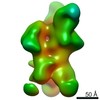
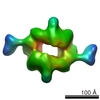
| タイトル | Anthrax toxin protective antigen channels bound to edema factor | ||||||||||||
 要素 要素 |
| ||||||||||||
 キーワード キーワード | TRANSLOCASE / anthrax toxin / protective antigen / edema factor | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont-mediated perturbation of host signal transduction pathway / symbiont-mediated cAMP intoxication of host cell / symbiont-mediated suppression of host MAPK cascade / calcium- and calmodulin-responsive adenylate cyclase activity / adenylate cyclase / cAMP biosynthetic process / adenylate cyclase activity / host cell cytosol / small molecule binding / Uptake and function of anthrax toxins ...symbiont-mediated perturbation of host signal transduction pathway / symbiont-mediated cAMP intoxication of host cell / symbiont-mediated suppression of host MAPK cascade / calcium- and calmodulin-responsive adenylate cyclase activity / adenylate cyclase / cAMP biosynthetic process / adenylate cyclase activity / host cell cytosol / small molecule binding / Uptake and function of anthrax toxins / catalytic complex / host cell endosome membrane / adenylate cyclase activator activity / protein homooligomerization / metallopeptidase activity / toxin activity / calmodulin binding / host cell plasma membrane / extracellular region / ATP binding / metal ion binding / identical protein binding / membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  | ||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | ||||||||||||
 データ登録者 データ登録者 | Hardenbrook, N.J. / Liu, S. / Zhou, K. / Zhou, Z.H. / Krantz, B.A. | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020タイトル: Atomic structures of anthrax toxin protective antigen channels bound to partially unfolded lethal and edema factors. 著者: Nathan J Hardenbrook / Shiheng Liu / Kang Zhou / Koyel Ghosal / Z Hong Zhou / Bryan A Krantz /  要旨: Following assembly, the anthrax protective antigen (PA) forms an oligomeric translocon that unfolds and translocates either its lethal factor (LF) or edema factor (EF) into the host cell. Here, we ...Following assembly, the anthrax protective antigen (PA) forms an oligomeric translocon that unfolds and translocates either its lethal factor (LF) or edema factor (EF) into the host cell. Here, we report the cryo-EM structures of heptameric PA channels with partially unfolded LF and EF at 4.6 and 3.1-Å resolution, respectively. The first α helix and β strand of LF and EF unfold and dock into a deep amphipathic cleft, called the α clamp, which resides at the interface of two PA monomers. The α-clamp-helix interactions exhibit structural plasticity when comparing the structures of lethal and edema toxins. EF undergoes a largescale conformational rearrangement when forming the complex with the channel. A critical loop in the PA binding interface is displaced for about 4 Å, leading to the weakening of the binding interface prior to translocation. These structures provide key insights into the molecular mechanisms of translocation-coupled protein unfolding and translocation. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6uze.cif.gz 6uze.cif.gz | 927.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6uze.ent.gz pdb6uze.ent.gz | 771.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6uze.json.gz 6uze.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6uze_validation.pdf.gz 6uze_validation.pdf.gz | 999.3 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6uze_full_validation.pdf.gz 6uze_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  6uze_validation.xml.gz 6uze_validation.xml.gz | 130.1 KB | 表示 | |
| CIF形式データ |  6uze_validation.cif.gz 6uze_validation.cif.gz | 203.2 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/uz/6uze https://data.pdbj.org/pub/pdb/validation_reports/uz/6uze ftp://data.pdbj.org/pub/pdb/validation_reports/uz/6uze ftp://data.pdbj.org/pub/pdb/validation_reports/uz/6uze | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 82768.828 Da / 分子数: 7 / 由来タイプ: 組換発現 / 由来: (組換発現)  発現宿主:  参照: UniProt: P13423 #2: タンパク質 | 分子量: 88955.578 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  発現宿主:  参照: UniProt: P40136, adenylate cyclase #3: 化合物 | ChemComp-CA / 研究の焦点であるリガンドがあるか | N | |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Anthrax toxin protective antigen channels bound to edema factor タイプ: COMPLEX / Entity ID: #1-#2 / 由来: RECOMBINANT |
|---|---|
| 分子量 | 値: 613 kDa/nm / 実験値: YES |
| 由来(天然) | 生物種:  |
| 由来(組換発現) | 生物種:  |
| 緩衝液 | pH: 8 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD |
| 撮影 | 電子線照射量: 62.9 e/Å2 / 検出モード: SUPER-RESOLUTION フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性: C1 (非対称) | ||||||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.4 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 73784 / 対称性のタイプ: POINT |
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj








