+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6tt7 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Ovine ATP synthase 1a state | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード | MEMBRANE PROTEIN / ATP synthase / mitochondrial / respiratory chain / mammalian | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / : / negative regulation of cell adhesion involved in substrate-bound cell migration / : / angiostatin binding / Formation of ATP by chemiosmotic coupling / Cristae formation / mitochondrial proton-transporting ATP synthase complex assembly / cellular response to interleukin-7 / proton channel activity ...: / : / negative regulation of cell adhesion involved in substrate-bound cell migration / : / angiostatin binding / Formation of ATP by chemiosmotic coupling / Cristae formation / mitochondrial proton-transporting ATP synthase complex assembly / cellular response to interleukin-7 / proton channel activity / Mitochondrial protein degradation / proton transmembrane transporter activity / proton motive force-driven ATP synthesis / MHC class I protein binding / proton-transporting two-sector ATPase complex, proton-transporting domain / mitochondrial nucleoid / proton motive force-driven mitochondrial ATP synthesis / proton-transporting ATPase activity, rotational mechanism / positive regulation of blood vessel endothelial cell migration / H+-transporting two-sector ATPase / proton-transporting ATP synthase complex / proton-transporting ATP synthase activity, rotational mechanism / proton transmembrane transport / aerobic respiration / regulation of intracellular pH / mitochondrial membrane / lipid metabolic process / angiogenesis / mitochondrial inner membrane / lipid binding / cell surface / mitochondrion / ATP binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Pinke, G. / Zhou, L. / Sazanov, L.A. | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2020 ジャーナル: Nat Struct Mol Biol / 年: 2020タイトル: Cryo-EM structure of the entire mammalian F-type ATP synthase. 著者: Gergely Pinke / Long Zhou / Leonid A Sazanov /  要旨: The majority of adenosine triphosphate (ATP) powering cellular processes in eukaryotes is produced by the mitochondrial F1Fo ATP synthase. Here, we present the atomic models of the membrane Fo domain ...The majority of adenosine triphosphate (ATP) powering cellular processes in eukaryotes is produced by the mitochondrial F1Fo ATP synthase. Here, we present the atomic models of the membrane Fo domain and the entire mammalian (ovine) F1Fo, determined by cryo-electron microscopy. Subunits in the membrane domain are arranged in the 'proton translocation cluster' attached to the c-ring and a more distant 'hook apparatus' holding subunit e. Unexpectedly, this subunit is anchored to a lipid 'plug' capping the c-ring. We present a detailed proton translocation pathway in mammalian Fo and key inter-monomer contacts in F1Fo multimers. Cryo-EM maps of F1Fo exposed to calcium reveal a retracted subunit e and a disassembled c-ring, suggesting permeability transition pore opening. We propose a model for the permeability transition pore opening, whereby subunit e pulls the lipid plug out of the c-ring. Our structure will allow the design of drugs for many emerging applications in medicine. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6tt7.cif.gz 6tt7.cif.gz | 1 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6tt7.ent.gz pdb6tt7.ent.gz | 825 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6tt7.json.gz 6tt7.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6tt7_validation.pdf.gz 6tt7_validation.pdf.gz | 1.8 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6tt7_full_validation.pdf.gz 6tt7_full_validation.pdf.gz | 1.9 MB | 表示 | |
| XML形式データ |  6tt7_validation.xml.gz 6tt7_validation.xml.gz | 147.8 KB | 表示 | |
| CIF形式データ |  6tt7_validation.cif.gz 6tt7_validation.cif.gz | 221.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/tt/6tt7 https://data.pdbj.org/pub/pdb/validation_reports/tt/6tt7 ftp://data.pdbj.org/pub/pdb/validation_reports/tt/6tt7 ftp://data.pdbj.org/pub/pdb/validation_reports/tt/6tt7 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-ATP synthase ... , 14種, 25分子 BCAEDFGHIJKM12345678NPQRS
| #1: タンパク質 | 分子量: 59779.492 Da / 分子数: 3 / 由来タイプ: 天然 / 由来: (天然)  #2: タンパク質 | 分子量: 56265.090 Da / 分子数: 3 / 由来タイプ: 天然 / 由来: (天然)  #3: タンパク質 | | 分子量: 33133.062 Da / 分子数: 1 / 由来タイプ: 天然 / 詳細: XP_004014229.1 / 由来: (天然)  #4: タンパク質 | | 分子量: 17580.924 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  #5: タンパク質 | | 分子量: 5779.863 Da / 分子数: 1 / 由来タイプ: 天然 / 詳細: XP_027832792.1 / 由来: (天然)  #6: タンパク質 | | 分子量: 23435.695 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  #7: タンパク質 | | 分子量: 28844.570 Da / 分子数: 1 / 由来タイプ: 天然 / 詳細: XP_004002367.2 / 由来: (天然)  #9: タンパク質 | | 分子量: 18747.508 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  #10: タンパク質 | 分子量: 14201.577 Da / 分子数: 8 / 由来タイプ: 天然 / 由来: (天然)  #11: タンパク質 | | 分子量: 24811.850 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  #13: タンパク質 | | 分子量: 6846.093 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  #14: タンパク質 | | 分子量: 7918.443 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  #15: タンパク質 | | 分子量: 10315.207 Da / 分子数: 1 / 由来タイプ: 天然 / 詳細: XP_004016938.3 / 由来: (天然)  #16: タンパク質 | | 分子量: 11428.407 Da / 分子数: 1 / 由来タイプ: 天然 / 詳細: XP_011950914.1 / 由来: (天然)  |
|---|
-タンパク質 , 3種, 3分子 LOT
| #8: タンパク質 | 分子量: 12490.296 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
|---|---|
| #12: タンパク質 | 分子量: 6443.579 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #17: タンパク質 | 分子量: 8336.688 Da / 分子数: 1 / 由来タイプ: 天然 / 詳細: XP_027827162.1 / 由来: (天然)  |
-非ポリマー , 6種, 19分子 




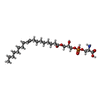





| #18: 化合物 | | #19: 化合物 | ChemComp-ADP / #20: 化合物 | ChemComp-LHG / #21: 化合物 | ChemComp-P5S / | #22: 化合物 | #23: 化合物 | ChemComp-S12 / | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | N |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Purified ATP synthase from sheep heart mitochondria / タイプ: COMPLEX / Entity ID: #1-#17 / 由来: NATURAL |
|---|---|
| 分子量 | 実験値: NO |
| 由来(天然) | 生物種:  |
| 緩衝液 | pH: 7.4 詳細: 20 mM HEPES pH7.4, 35 mM NaCl, 2 mM EDTA, 1% sucrose, 1 mM DTT, 0.1% LMNG, 0.2% CHAPS |
| 試料 | 濃度: 2.8 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 試料支持 | グリッドの材料: COPPER / グリッドのサイズ: 400 divisions/in. / グリッドのタイプ: Quantifoil R0.6/1 |
| 急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE-PROPANE / 湿度: 100 % / 凍結前の試料温度: 277 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(補正後): 131951 X / Calibrated defocus min: 1000 nm / 最大 デフォーカス(補正後): 2400 nm / Cs: 2.7 mm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 平均露光時間: 1 sec. / 電子線照射量: 106 e/Å2 / 検出モード: INTEGRATING フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 撮影したグリッド数: 1 / 実像数: 3053 / 詳細: 40 frames per movie |
| 画像スキャン | サンプリングサイズ: 14 µm / 横: 4096 / 縦: 4096 |
- 解析
解析
| ソフトウェア | 名称: PHENIX / バージョン: 1.12_2829: / 分類: 精密化 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 809000 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性: C1 (非対称) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 35000 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj














