| 登録情報 | データベース: PDB / ID: 6tj3
|
|---|
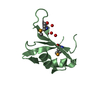
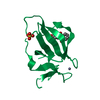
| タイトル | P. falciparum essential light chain, N-terminal domain |
|---|
 要素 要素 | PfELC |
|---|
 キーワード キーワード | MOTOR PROTEIN / motility / glideosome / light chain / myosin |
|---|
| 機能・相同性 | EF-hand domain pair / Myosin essential light chain ELC 機能・相同性情報 機能・相同性情報 |
|---|
| 生物種 |   Plasmodium falciparum 3D7 (マラリア病原虫) Plasmodium falciparum 3D7 (マラリア病原虫) |
|---|
| 手法 | 溶液NMR / simulated annealing |
|---|
 データ登録者 データ登録者 | Weininger, U. / Pazicky, S. / Loew, C. |
|---|
| 資金援助 |  ドイツ, ドイツ,  スウェーデン, 2件 スウェーデン, 2件 | 組織 | 認可番号 | 国 |
|---|
| Joachim Herz Foundation | 800026 |  ドイツ ドイツ | | Swedish Research Council | 621-2013-5905 |  スウェーデン スウェーデン |
|
|---|
 引用 引用 |  ジャーナル: Commun Biol / 年: 2020 ジャーナル: Commun Biol / 年: 2020
タイトル: Structural role of essential light chains in the apicomplexan glideosome.
著者: Pazicky, S. / Dhamotharan, K. / Kaszuba, K. / Mertens, H.D.T. / Gilberger, T. / Svergun, D. / Kosinski, J. / Weininger, U. / Low, C. |
|---|
| 履歴 | | 登録 | 2019年11月25日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2020年10月28日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2024年5月15日 | Group: Data collection / Database references / カテゴリ: chem_comp_atom / chem_comp_bond / database_2
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 データ登録者
データ登録者 ドイツ,
ドイツ,  スウェーデン, 2件
スウェーデン, 2件  引用
引用 ジャーナル: Commun Biol / 年: 2020
ジャーナル: Commun Biol / 年: 2020 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6tj3.cif.gz
6tj3.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6tj3.ent.gz
pdb6tj3.ent.gz PDB形式
PDB形式 6tj3.json.gz
6tj3.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/tj/6tj3
https://data.pdbj.org/pub/pdb/validation_reports/tj/6tj3 ftp://data.pdbj.org/pub/pdb/validation_reports/tj/6tj3
ftp://data.pdbj.org/pub/pdb/validation_reports/tj/6tj3




 リンク
リンク 集合体
集合体
 要素
要素

 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj
 SAXS
SAXS