| 登録情報 | データベース: PDB / ID: 6szc
|
|---|
| タイトル | NMR structure of repeat domain 13 of the fibrillar adhesin CshA from Streptococcus gordonii. |
|---|
 要素 要素 | Surface-associated protein CshA |
|---|
 キーワード キーワード | CELL ADHESION / Adhesin / Fibril / Novel / Repeat domain / Streptococcus |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
CshA domain / GEVED domain / Surface adhesin CshA repetitive domain / GEVED domain / YSIRK type signal peptide / YSIRK Gram-positive signal peptide / LPXTG cell wall anchor motif / Gram-positive cocci surface proteins LPxTG motif profile. / LPXTG cell wall anchor domain類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Streptococcus gordonii (バクテリア) Streptococcus gordonii (バクテリア) |
|---|
| 手法 | 溶液NMR / simulated annealing |
|---|
 データ登録者 データ登録者 | Higman, V.A. / Back, C. / Crump, M.P. / Race, P. |
|---|
| 資金援助 |  英国, 4件 英国, 4件 | 組織 | 認可番号 | 国 |
|---|
| National Institutes of Health/National Institute of Dental and Craniofacial Research (NIH/NIDCR) | DE016690 |  英国 英国 | | Biotechnology and Biological Sciences Research Council | BB/L01386X/1 |  英国 英国 | | Biotechnology and Biological Sciences Research Council | BB/M025624/1 |  英国 英国 | | Royal Society | UF080534 |  英国 英国 |
|
|---|
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2020 ジャーナル: J.Biol.Chem. / 年: 2020
タイトル: The streptococcal multidomain fibrillar adhesin CshA has an elongated polymeric architecture.
著者: Back, C.R. / Higman, V.A. / Le Vay, K. / Patel, V.V. / Parnell, A.E. / Frankel, D. / Jenkinson, H.F. / Burston, S.G. / Crump, M.P. / Nobbs, A.H. / Race, P.R. |
|---|
| 履歴 | | 登録 | 2019年10月2日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2020年4月8日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2020年5月20日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.journal_volume / _citation.page_first ..._citation.journal_volume / _citation.page_first / _citation.page_last / _citation_author.identifier_ORCID / _citation_author.name |
|---|
| 改定 1.2 | 2022年3月30日 | Group: Author supporting evidence / Database references / カテゴリ: database_2 / pdbx_audit_support
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_audit_support.funding_organization |
|---|
| 改定 1.3 | 2023年6月14日 | Group: Other / カテゴリ: pdbx_database_status / Item: _pdbx_database_status.status_code_nmr_data |
|---|
| 改定 1.4 | 2024年6月19日 | Group: Data collection / Database references / カテゴリ: chem_comp_atom / chem_comp_bond / database_2 / Item: _database_2.pdbx_DOI |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Streptococcus gordonii (バクテリア)
Streptococcus gordonii (バクテリア) データ登録者
データ登録者 英国, 4件
英国, 4件  引用
引用 ジャーナル: J.Biol.Chem. / 年: 2020
ジャーナル: J.Biol.Chem. / 年: 2020 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6szc.cif.gz
6szc.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6szc.ent.gz
pdb6szc.ent.gz PDB形式
PDB形式 6szc.json.gz
6szc.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6szc_validation.pdf.gz
6szc_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6szc_full_validation.pdf.gz
6szc_full_validation.pdf.gz 6szc_validation.xml.gz
6szc_validation.xml.gz 6szc_validation.cif.gz
6szc_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/sz/6szc
https://data.pdbj.org/pub/pdb/validation_reports/sz/6szc ftp://data.pdbj.org/pub/pdb/validation_reports/sz/6szc
ftp://data.pdbj.org/pub/pdb/validation_reports/sz/6szc リンク
リンク 集合体
集合体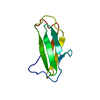
 要素
要素 Streptococcus gordonii (strain Challis / ATCC 35105 / BCRC 15272 / CH1 / DL1 / V288) (バクテリア)
Streptococcus gordonii (strain Challis / ATCC 35105 / BCRC 15272 / CH1 / DL1 / V288) (バクテリア)
 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー









 PDBj
PDBj gel filtration
gel filtration