1
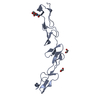
B: Agglutinin-like protein 5 概要 構成要素の詳細 対称操作
分子量 (理論値) 分子数 合計 (水以外) 22,489 36 ポリマ- 22,489 36 非ポリマー 0 0 水 649 36
タイプ 名称 対称操作 数 identity operation 1_555 x,y,z 1 crystal symmetry operation 1_515 x,y-4,z 1 crystal symmetry operation 1_525 x,y-3,z 1 crystal symmetry operation 1_535 x,y-2,z 1 crystal symmetry operation 1_545 x,y-1,z 1 crystal symmetry operation 1_565 x,y+1,z 1 crystal symmetry operation 1_575 x,y+2,z 1 crystal symmetry operation 1_585 x,y+3,z 1 crystal symmetry operation 1_595 x,y+4,z 1 crystal symmetry operation 1_415 x-1,y-4,z 1 crystal symmetry operation 1_425 x-1,y-3,z 1 crystal symmetry operation 1_435 x-1,y-2,z 1 crystal symmetry operation 1_445 x-1,y-1,z 1 crystal symmetry operation 1_455 x-1,y,z 1 crystal symmetry operation 1_465 x-1,y+1,z 1 crystal symmetry operation 1_475 x-1,y+2,z 1 crystal symmetry operation 1_485 x-1,y+3,z 1 crystal symmetry operation 1_495 x-1,y+4,z 1 crystal symmetry operation 2_415 -x-1,y-7/2,-z 1 crystal symmetry operation 2_425 -x-1,y-5/2,-z 1 crystal symmetry operation 2_435 -x-1,y-3/2,-z 1 crystal symmetry operation 2_445 -x-1,y-1/2,-z 1 crystal symmetry operation 2_455 -x-1,y+1/2,-z 1 crystal symmetry operation 2_465 -x-1,y+3/2,-z 1 crystal symmetry operation 2_475 -x-1,y+5/2,-z 1 crystal symmetry operation 2_485 -x-1,y+7/2,-z 1 crystal symmetry operation 2_495 -x-1,y+9/2,-z 1 crystal symmetry operation 2_315 -x-2,y-7/2,-z 1 crystal symmetry operation 2_325 -x-2,y-5/2,-z 1 crystal symmetry operation 2_335 -x-2,y-3/2,-z 1 crystal symmetry operation 2_345 -x-2,y-1/2,-z 1 crystal symmetry operation 2_355 -x-2,y+1/2,-z 1 crystal symmetry operation 2_365 -x-2,y+3/2,-z 1 crystal symmetry operation 2_375 -x-2,y+5/2,-z 1 crystal symmetry operation 2_385 -x-2,y+7/2,-z 1 crystal symmetry operation 2_395 -x-2,y+9/2,-z 1
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Candida albicans (酵母)
Candida albicans (酵母) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.2 Å
分子置換 / 解像度: 1.2 Å  データ登録者
データ登録者 引用
引用 ジャーナル: To be published
ジャーナル: To be published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6rhd.cif.gz
6rhd.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6rhd.ent.gz
pdb6rhd.ent.gz PDB形式
PDB形式 6rhd.json.gz
6rhd.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/rh/6rhd
https://data.pdbj.org/pub/pdb/validation_reports/rh/6rhd ftp://data.pdbj.org/pub/pdb/validation_reports/rh/6rhd
ftp://data.pdbj.org/pub/pdb/validation_reports/rh/6rhd リンク
リンク 集合体
集合体

 要素
要素 Candida albicans (酵母) / 参照: UniProt: O13368
Candida albicans (酵母) / 参照: UniProt: O13368 X線回折 / 使用した結晶の数: 2
X線回折 / 使用した結晶の数: 2  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ESRF
ESRF  / ビームライン: ID23-2 / 波長: 0.8729 Å
/ ビームライン: ID23-2 / 波長: 0.8729 Å 分子置換
分子置換 解析
解析 分子置換 / 解像度: 1.2→16.72 Å / Cor.coef. Fo:Fc: 0.99 / Cor.coef. Fo:Fc free: 0.987 / SU B: 1.424 / SU ML: 0.025 / SU R Cruickshank DPI: 0.0389 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.039 / ESU R Free: 0.035
分子置換 / 解像度: 1.2→16.72 Å / Cor.coef. Fo:Fc: 0.99 / Cor.coef. Fo:Fc free: 0.987 / SU B: 1.424 / SU ML: 0.025 / SU R Cruickshank DPI: 0.0389 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.039 / ESU R Free: 0.035  ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj
