| 登録情報 | データベース: PDB / ID: 6r5m
|
|---|
| タイトル | Crystal structure of toxin MT9 from mamba venom |
|---|
 要素 要素 | Dendroaspis polylepis MT9 |
|---|
 キーワード キーワード | TOXIN / mamba venom / aminergic toxin |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
Snake toxin, conserved site / CD59 / CD59 / Ribbon / Mainly Beta類似検索 - ドメイン・相同性 ACETYL GROUP / S-1,2-PROPANEDIOL / Dendroaspis polylepis MT9類似検索 - 構成要素 |
|---|
| 生物種 |  Dendroaspis polylepis (コブラ) Dendroaspis polylepis (コブラ) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.9 Å 分子置換 / 解像度: 1.9 Å |
|---|
 データ登録者 データ登録者 | Stura, E.A. / Tepshi, L. / Ciolek, J. / Triquigneaux, M. / Zoukimian, C. / De Waard, M. / Beroud, R. / Servent, D. / Gilles, N. / Legrand, P. / Ciccone, L. |
|---|
| 資金援助 |  フランス, 1件 フランス, 1件 | 組織 | 認可番号 | 国 |
|---|
| French National Research Agency | ANR-11-RPIB-022-04 venompicoscreen |  フランス フランス |
|
|---|
 引用 引用 |  ジャーナル: Biomed Pharmacother / 年: 2022 ジャーナル: Biomed Pharmacother / 年: 2022
タイトル: MT9, a natural peptide from black mamba venom antagonizes the muscarinic type 2 receptor and reverses the M2R-agonist-induced relaxation in rat and human arteries
著者: Ciolek, J. / Zoukimian, C. / Dhot, J. / Burban, M. / Triquigneaux, M. / Lauzier, B. / Guimbert, C. / Boturyn, D. / Ferron, M. / Ciccone, L. / Tepshi, L. / Stura, E. / Legrand, P. / Robin, P. ...著者: Ciolek, J. / Zoukimian, C. / Dhot, J. / Burban, M. / Triquigneaux, M. / Lauzier, B. / Guimbert, C. / Boturyn, D. / Ferron, M. / Ciccone, L. / Tepshi, L. / Stura, E. / Legrand, P. / Robin, P. / Mourier, G. / Schaack, B. / Fellah, I. / Blanchet, G. / Gauthier-Erfanian, C. / Beroud, R. / Servent, D. / De Waard, M. / Gilles, N. |
|---|
| 履歴 | | 登録 | 2019年3月25日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 置き換え | 2020年2月12日 | ID: 6F21 |
|---|
| 改定 1.0 | 2020年2月12日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2022年5月25日 | Group: Database references / カテゴリ: citation / citation_author / database_2
Item: _citation.journal_abbrev / _citation.journal_id_CSD ..._citation.journal_abbrev / _citation.journal_id_CSD / _citation.journal_volume / _citation.page_first / _citation.pdbx_database_id_DOI / _citation.title / _citation.year / _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Dendroaspis polylepis (コブラ)
Dendroaspis polylepis (コブラ) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.9 Å
分子置換 / 解像度: 1.9 Å  データ登録者
データ登録者 フランス, 1件
フランス, 1件  引用
引用 ジャーナル: Biomed Pharmacother / 年: 2022
ジャーナル: Biomed Pharmacother / 年: 2022 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6r5m.cif.gz
6r5m.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6r5m.ent.gz
pdb6r5m.ent.gz PDB形式
PDB形式 6r5m.json.gz
6r5m.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6r5m_validation.pdf.gz
6r5m_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6r5m_full_validation.pdf.gz
6r5m_full_validation.pdf.gz 6r5m_validation.xml.gz
6r5m_validation.xml.gz 6r5m_validation.cif.gz
6r5m_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/r5/6r5m
https://data.pdbj.org/pub/pdb/validation_reports/r5/6r5m ftp://data.pdbj.org/pub/pdb/validation_reports/r5/6r5m
ftp://data.pdbj.org/pub/pdb/validation_reports/r5/6r5m リンク
リンク 集合体
集合体
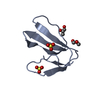


 要素
要素 Dendroaspis polylepis (コブラ) / 参照: UniProt: A0A4P1LYC9*PLUS
Dendroaspis polylepis (コブラ) / 参照: UniProt: A0A4P1LYC9*PLUS
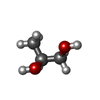







 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SOLEIL
SOLEIL  / ビームライン: PROXIMA 2 / 波長: 0.980097 Å
/ ビームライン: PROXIMA 2 / 波長: 0.980097 Å 解析
解析 分子置換 / 解像度: 1.9→19.45 Å / Cor.coef. Fo:Fc: 0.913 / Cor.coef. Fo:Fc free: 0.909 / SU R Cruickshank DPI: 0.17 / 交差検証法: THROUGHOUT / σ(F): 0 / SU R Blow DPI: 0.192 / SU Rfree Blow DPI: 0.156 / SU Rfree Cruickshank DPI: 0.146
分子置換 / 解像度: 1.9→19.45 Å / Cor.coef. Fo:Fc: 0.913 / Cor.coef. Fo:Fc free: 0.909 / SU R Cruickshank DPI: 0.17 / 交差検証法: THROUGHOUT / σ(F): 0 / SU R Blow DPI: 0.192 / SU Rfree Blow DPI: 0.156 / SU Rfree Cruickshank DPI: 0.146  ムービー
ムービー コントローラー
コントローラー









 PDBj
PDBj





