+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6qkk | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Aplysia californica AChBP in complex with 2-Fluoro-(carbamoylpyridinyl)deschloroepibatidine analogue (1) | ||||||
 要素 要素 | Soluble acetylcholine receptor | ||||||
 キーワード キーワード | SIGNALING PROTEIN / AChBP / Acetylcholine | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報excitatory extracellular ligand-gated monoatomic ion channel activity / transmitter-gated monoatomic ion channel activity involved in regulation of postsynaptic membrane potential / transmembrane signaling receptor activity / postsynapse / neuron projection / identical protein binding / membrane / metal ion binding 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.2 Å 分子置換 / 解像度: 2.2 Å | ||||||
 データ登録者 データ登録者 | Davis, S. / Bueno, R.V. / Dawson, A. / Hunter, W.N. | ||||||
 引用 引用 |  ジャーナル: Acta Crystallogr.,Sect.D / 年: 2022 ジャーナル: Acta Crystallogr.,Sect.D / 年: 2022タイトル: Interactions between 2'-fluoro-(carbamoylpyridinyl)deschloroepibatidine analogues and acetylcholine-binding protein inform on potent antagonist activity against nicotinic receptors 著者: Bueno, R.V. / Davis, S. / Dawson, A. / Ondachi, P.W. / Carroll, F.I. / Hunter, W.N. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6qkk.cif.gz 6qkk.cif.gz | 443.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6qkk.ent.gz pdb6qkk.ent.gz | 364.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6qkk.json.gz 6qkk.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6qkk_validation.pdf.gz 6qkk_validation.pdf.gz | 3.2 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6qkk_full_validation.pdf.gz 6qkk_full_validation.pdf.gz | 3.2 MB | 表示 | |
| XML形式データ |  6qkk_validation.xml.gz 6qkk_validation.xml.gz | 83.7 KB | 表示 | |
| CIF形式データ |  6qkk_validation.cif.gz 6qkk_validation.cif.gz | 116.2 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/qk/6qkk https://data.pdbj.org/pub/pdb/validation_reports/qk/6qkk ftp://data.pdbj.org/pub/pdb/validation_reports/qk/6qkk ftp://data.pdbj.org/pub/pdb/validation_reports/qk/6qkk | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 / 糖 , 2種, 14分子 ABCDEFGHIJ

| #1: タンパク質 | 分子量: 28326.750 Da / 分子数: 10 / 由来タイプ: 組換発現 詳細: all disulfide bonds have been represented as oxidised, as due to radiation damage the exact oxidation state of the disulfides in some chains is unclear. The sequence conflicts are because we ...詳細: all disulfide bonds have been represented as oxidised, as due to radiation damage the exact oxidation state of the disulfides in some chains is unclear. The sequence conflicts are because we have used the AChBP gene sequence provided in the Aplysia californica genome (http://genome-euro.ucsc.edu/cgi-bin/hgGateway?hgsid=589127391_t0Vv5jEv8KyWGt7MjPEaZKNxTL3u&redirect=manual&source=genome.cse.ucsc.edu), which differs from the UniProt Q8WSF8 Sequence by identifying both ALA 60 and and ALA 155 as VAL 由来: (組換発現)  発現宿主:  参照: UniProt: Q8WSF8 #5: 糖 | ChemComp-NAG / |
|---|
-非ポリマー , 5種, 1199分子 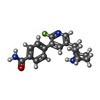


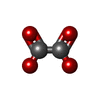





| #2: 化合物 | ChemComp-H92 / #3: 化合物 | ChemComp-PO4 / #4: 化合物 | ChemComp-EDO / #6: 化合物 | ChemComp-OXL / | #7: 水 | ChemComp-HOH / | |
|---|
-詳細
| 配列の詳細 | all disulfide bonds have been represented as oxidised, as due to radiation damage the exact ...all disulfide bonds have been represented as oxidised, as due to radiation damage the exact oxidation state of the disulfides in some chains is unclear |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.39 Å3/Da / 溶媒含有率: 63.68 % / 解説: Block |
|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 4.2 詳細: Reservoir: 0.2 M NaCl, 0.1 M Phosphate/citrate pH 4.2 12% PEG 8000 Buffer: 50 mM Tris, 250 mM NaCl pH 7.5 Protein concentration 4 mg/ml Microseeded |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  Diamond Diamond  / ビームライン: I24 / 波長: 0.96858 Å / ビームライン: I24 / 波長: 0.96858 Å |
| 検出器 | タイプ: DECTRIS PILATUS 6M-F / 検出器: PIXEL / 日付: 2017年7月3日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.96858 Å / 相対比: 1 |
| 反射 | 解像度: 2.2→48.88 Å / Num. obs: 177603 / % possible obs: 99.9 % / 冗長度: 3.4 % / Biso Wilson estimate: 15.5 Å2 / CC1/2: 0.95 / Rmerge(I) obs: 0.172 / Net I/σ(I): 6.6 |
| 反射 シェル | 解像度: 2.2→2.24 Å / 冗長度: 3.4 % / Rmerge(I) obs: 0.888 / Mean I/σ(I) obs: 2 / Num. unique obs: 8766 / CC1/2: 0.43 / % possible all: 98.4 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 2XYS 解像度: 2.2→48.88 Å / Cor.coef. Fo:Fc: 0.929 / Cor.coef. Fo:Fc free: 0.901 / SU B: 5.562 / SU ML: 0.133 / 交差検証法: THROUGHOUT / ESU R: 0.193 / ESU R Free: 0.167 / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 18.569 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: 1 / 解像度: 2.2→48.88 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー























 PDBj
PDBj





