| 登録情報 | データベース: PDB / ID: 6kor
|
|---|
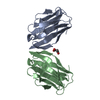
| タイトル | Crystal structure of the RRM domain of SYNCRIP |
|---|
 要素 要素 | Heterogeneous nuclear ribonucleoprotein Q |
|---|
 キーワード キーワード | RNA BINDING PROTEIN / RNA recognition motif |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
mRNA editing complex / : / CRD-mediated mRNA stability complex / negative regulation of nuclear-transcribed mRNA catabolic process, nonsense-mediated decay / mCRD-mediated mRNA stability complex / negative regulation of nuclear-transcribed mRNA catabolic process, deadenylation-dependent decay / CRD-mediated mRNA stabilization / mRNA modification / histone pre-mRNA 3'end processing complex / positive regulation of cytoplasmic translation ...mRNA editing complex / : / CRD-mediated mRNA stability complex / negative regulation of nuclear-transcribed mRNA catabolic process, nonsense-mediated decay / mCRD-mediated mRNA stability complex / negative regulation of nuclear-transcribed mRNA catabolic process, deadenylation-dependent decay / CRD-mediated mRNA stabilization / mRNA modification / histone pre-mRNA 3'end processing complex / positive regulation of cytoplasmic translation / GAIT complex / RNA processing / catalytic step 2 spliceosome / RNA splicing / cellular response to type II interferon / mRNA 5'-UTR binding / mRNA splicing, via spliceosome / osteoblast differentiation / negative regulation of translation / ribonucleoprotein complex / endoplasmic reticulum / RNA binding / nucleoplasm / membrane / nucleus / cytosol類似検索 - 分子機能 Heterogeneous nuclear ribonucleoprotein Q, RNA recognition motif 1 / Heterogeneous nuclear ribonucleoprotein Q, RNA recognition motif 2 / Heterogeneous nuclear ribonucleoprotein Q acidic domain / Heterogeneous nuclear ribonucleoprotein Q acidic domain / HnRNP R/Q splicing factor / RRM (RNA recognition motif) domain / RNA recognition motif / RNA recognition motif / Eukaryotic RNA Recognition Motif (RRM) profile. / RNA recognition motif domain ...Heterogeneous nuclear ribonucleoprotein Q, RNA recognition motif 1 / Heterogeneous nuclear ribonucleoprotein Q, RNA recognition motif 2 / Heterogeneous nuclear ribonucleoprotein Q acidic domain / Heterogeneous nuclear ribonucleoprotein Q acidic domain / HnRNP R/Q splicing factor / RRM (RNA recognition motif) domain / RNA recognition motif / RNA recognition motif / Eukaryotic RNA Recognition Motif (RRM) profile. / RNA recognition motif domain / RNA-binding domain superfamily / Nucleotide-binding alpha-beta plait domain superfamily / Alpha-Beta Plaits / 2-Layer Sandwich / Alpha Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 2.602 Å 分子置換 / 解像度: 2.602 Å |
|---|
 データ登録者 データ登録者 | Chen, Y. / Chan, J. / Chen, W. / Jobichen, C. |
|---|
 引用 引用 |  ジャーナル: Rna / 年: 2020 ジャーナル: Rna / 年: 2020
タイトル: SYNCRIP, a new player in pri-let-7a processing.
著者: Chen, Y. / Chan, J. / Chen, W. / Li, J. / Sun, M. / Kannan, G.S. / Mok, Y.K. / Yuan, Y.A. / Jobichen, C. |
|---|
| 履歴 | | 登録 | 2019年8月12日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2020年1月22日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2020年3月4日 | Group: Database references / カテゴリ: citation
Item: _citation.journal_volume / _citation.page_first / _citation.page_last |
|---|
| 改定 1.2 | 2023年11月22日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  分子置換 / 解像度: 2.602 Å
分子置換 / 解像度: 2.602 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Rna / 年: 2020
ジャーナル: Rna / 年: 2020 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6kor.cif.gz
6kor.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6kor.ent.gz
pdb6kor.ent.gz PDB形式
PDB形式 6kor.json.gz
6kor.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/ko/6kor
https://data.pdbj.org/pub/pdb/validation_reports/ko/6kor ftp://data.pdbj.org/pub/pdb/validation_reports/ko/6kor
ftp://data.pdbj.org/pub/pdb/validation_reports/ko/6kor
 リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: SYNCRIP, HNRPQ, NSAP1 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: SYNCRIP, HNRPQ, NSAP1 / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 回転陽極 / タイプ: RIGAKU MICROMAX-007 HF / 波長: 1.54187 Å
回転陽極 / タイプ: RIGAKU MICROMAX-007 HF / 波長: 1.54187 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj
