+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6klv | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Hyperthermophilic respiratory Complex III | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード | OXIDOREDUCTASE / Respiratory chain / Complex III / Hyperthermophilic mechanism / antimycin A | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ubiquinol-cytochrome-c reductase activity / respiratory electron transport chain / 2 iron, 2 sulfur cluster binding / electron transfer activity / oxidoreductase activity / heme binding / membrane / metal ion binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Aquifex aeolicus (バクテリア) Aquifex aeolicus (バクテリア) | |||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Fei, S. / Hartmut, M. / Yun, Z. / Guohong, P. / Guoliang, Z. / Hui, Z. / Shuangbo, Z. / Xiaoyun, P. / Yan, Z. | |||||||||
 引用 引用 |  ジャーナル: Angew Chem Int Ed Engl / 年: 2020 ジャーナル: Angew Chem Int Ed Engl / 年: 2020タイトル: A 3.3 Å-Resolution Structure of Hyperthermophilic Respiratory Complex III Reveals the Mechanism of Its Thermal Stability. 著者: Guoliang Zhu / Hui Zeng / Shuangbo Zhang / Jana Juli / Xiaoyun Pang / Jan Hoffmann / Yan Zhang / Nina Morgner / Yun Zhu / Guohong Peng / Hartmut Michel / Fei Sun /   要旨: Respiratory chain complexes convert energy by coupling electron flow to transmembrane proton translocation. Owing to a lack of atomic structures of cytochrome bc complex (Complex III) from ...Respiratory chain complexes convert energy by coupling electron flow to transmembrane proton translocation. Owing to a lack of atomic structures of cytochrome bc complex (Complex III) from thermophilic bacteria, little is known about the adaptations of this macromolecular machine to hyperthermophilic environments. In this study, we purified the cytochrome bc complex of Aquifex aeolicus, one of the most extreme thermophilic bacteria known, and determined its structure with and without an inhibitor at 3.3 Å resolution. Several residues unique for thermophilic bacteria were detected that provide additional stabilization for the structure. An extra transmembrane helix at the N-terminus of cyt. c was found to greatly enhance the interaction between cyt. b and cyt. c , and to bind a phospholipid molecule to stabilize the complex in the membrane. These results provide the structural basis for the hyperstability of the cytochrome bc complex in an extreme thermal environment. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6klv.cif.gz 6klv.cif.gz | 291.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6klv.ent.gz pdb6klv.ent.gz | 245.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6klv.json.gz 6klv.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6klv_validation.pdf.gz 6klv_validation.pdf.gz | 2 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6klv_full_validation.pdf.gz 6klv_full_validation.pdf.gz | 2 MB | 表示 | |
| XML形式データ |  6klv_validation.xml.gz 6klv_validation.xml.gz | 66.5 KB | 表示 | |
| CIF形式データ |  6klv_validation.cif.gz 6klv_validation.cif.gz | 88 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/kl/6klv https://data.pdbj.org/pub/pdb/validation_reports/kl/6klv ftp://data.pdbj.org/pub/pdb/validation_reports/kl/6klv ftp://data.pdbj.org/pub/pdb/validation_reports/kl/6klv | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-タンパク質 , 3種, 6分子 ADBECF
| #1: タンパク質 | 分子量: 19429.857 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)   Aquifex aeolicus (strain VF5) (バクテリア) Aquifex aeolicus (strain VF5) (バクテリア)株: VF5 / 参照: UniProt: O66460 #2: タンパク質 | 分子量: 47045.629 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)   Aquifex aeolicus (バクテリア) / 参照: UniProt: O66459*PLUS Aquifex aeolicus (バクテリア) / 参照: UniProt: O66459*PLUS#3: タンパク質 | 分子量: 27680.170 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)   Aquifex aeolicus (strain VF5) (バクテリア) Aquifex aeolicus (strain VF5) (バクテリア)株: VF5 / 参照: UniProt: O66458 |
|---|
-非ポリマー , 6種, 17分子 
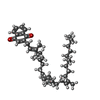

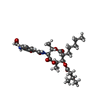







| #4: 化合物 | | #5: 化合物 | ChemComp-DLX / #6: 化合物 | ChemComp-HEM / #7: 化合物 | #8: 化合物 | #9: 化合物 | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | N |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: The cytochrome bc1 complex (respiratory complex III) タイプ: COMPLEX / Entity ID: #1-#3 / 由来: NATURAL |
|---|---|
| 由来(天然) | 生物種:   Aquifex aeolicus (バクテリア) Aquifex aeolicus (バクテリア) |
| 緩衝液 | pH: 7.4 |
| 試料 | 濃度: 1.5 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: OTHER FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: OTHER |
| 電子レンズ | モード: BRIGHT FIELD |
| 撮影 | 電子線照射量: 60 e/Å2 フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) |
- 解析
解析
| ソフトウェア | 名称: PHENIX / バージョン: 1.14_3260: / 分類: 精密化 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: NONE | ||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.2 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 81350 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj



















