[English] 日本語
 Yorodumi
Yorodumi- PDB-6kam: Crystal structure of FKRP in complex with Ba ion, CDP-ribtol, and... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 6kam | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Crystal structure of FKRP in complex with Ba ion, CDP-ribtol, and sugar acceptor | |||||||||
 Components Components | Fukutin-related protein | |||||||||
 Keywords Keywords | TRANSFERASE / glycosyltranferase / phospho ribitoyl tranferase | |||||||||
| Function / homology |  Function and homology information Function and homology informationpentitol metabolic process / filtration diaphragm assembly / pentose metabolic process / connective tissue development / creatine metabolic process / connective tissue replacement / Transferases; Transferring phosphorus-containing groups; Transferases for other substituted phosphate groups / oxygen metabolic process / phosphotransferase activity, for other substituted phosphate groups / localization of cell ...pentitol metabolic process / filtration diaphragm assembly / pentose metabolic process / connective tissue development / creatine metabolic process / connective tissue replacement / Transferases; Transferring phosphorus-containing groups; Transferases for other substituted phosphate groups / oxygen metabolic process / phosphotransferase activity, for other substituted phosphate groups / localization of cell / diaphragm development / maintenance of protein localization in endoplasmic reticulum / protein O-linked glycosylation via mannose / skeletal muscle fiber differentiation / basement membrane organization / dystroglycan binding / glial cell differentiation / reelin-mediated signaling pathway / respiratory system process / skeletal muscle tissue regeneration / protein import / camera-type eye development / response to alcohol / adult walking behavior / neuromuscular process / bone mineralization / skeletal muscle myofibril / heart morphogenesis / rough endoplasmic reticulum / laminin binding / response to glucocorticoid / muscle contraction / response to activity / glycolytic process / lipid metabolic process / protein tetramerization / phosphatidylinositol 3-kinase/protein kinase B signal transduction / sarcolemma / protein processing / brain development / neuron migration / in utero embryonic development / response to xenobiotic stimulus / inflammatory response / Golgi membrane / Golgi apparatus / extracellular space / nucleoplasm / metal ion binding / identical protein binding / cytosol Similarity search - Function | |||||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | |||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.46 Å MOLECULAR REPLACEMENT / Resolution: 2.46 Å | |||||||||
 Authors Authors | Kuwabara, N. | |||||||||
| Funding support |  Japan, 2items Japan, 2items
| |||||||||
 Citation Citation |  Journal: Nat Commun / Year: 2020 Journal: Nat Commun / Year: 2020Title: Crystal structures of fukutin-related protein (FKRP), a ribitol-phosphate transferase related to muscular dystrophy. Authors: Kuwabara, N. / Imae, R. / Manya, H. / Tanaka, T. / Mizuno, M. / Tsumoto, H. / Kanagawa, M. / Kobayashi, K. / Toda, T. / Senda, T. / Endo, T. / Kato, R. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  6kam.cif.gz 6kam.cif.gz | 848.2 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb6kam.ent.gz pdb6kam.ent.gz | 596.7 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  6kam.json.gz 6kam.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/ka/6kam https://data.pdbj.org/pub/pdb/validation_reports/ka/6kam ftp://data.pdbj.org/pub/pdb/validation_reports/ka/6kam ftp://data.pdbj.org/pub/pdb/validation_reports/ka/6kam | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  6kajC  6kakC  6kalC  6kanC  6l7sC  6l7tC  6l7uC C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||
| Unit cell |
|
- Components
Components
-Protein , 1 types, 4 molecules ABCD
| #1: Protein | Mass: 50026.805 Da / Num. of mol.: 4 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: FKRP / Cell line (production host): HEK293S GnT- / Production host: Homo sapiens (human) / Gene: FKRP / Cell line (production host): HEK293S GnT- / Production host:  Homo sapiens (human) / References: UniProt: Q9H9S5 Homo sapiens (human) / References: UniProt: Q9H9S5 |
|---|
-Sugars , 2 types, 8 molecules 
| #2: Polysaccharide | 2-acetamido-2-deoxy-beta-D-galactopyranose-(1-3)-2-acetamido-2-deoxy-beta-D-glucopyranose-(1-4)- ...2-acetamido-2-deoxy-beta-D-galactopyranose-(1-3)-2-acetamido-2-deoxy-beta-D-glucopyranose-(1-4)-alpha-D-mannopyranose Source method: isolated from a genetically manipulated source |
|---|---|
| #4: Sugar | ChemComp-NAG / |
-Non-polymers , 6 types, 211 molecules 
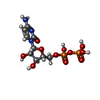
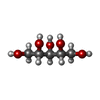








| #3: Chemical | ChemComp-ZN / #5: Chemical | ChemComp-CDP / #6: Chemical | ChemComp-RB0 / #7: Chemical | ChemComp-BA / #8: Chemical | #9: Water | ChemComp-HOH / | |
|---|
-Details
| Has ligand of interest | Y |
|---|---|
| Has protein modification | Y |
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.98 Å3/Da / Density % sol: 58.73 % |
|---|---|
| Crystal grow | Temperature: 293 K / Method: vapor diffusion, hanging drop / pH: 7.5 Details: 4-8 % PEG 4,000, 0.1 M HEPES-NaOH (pH7.5), 0.5 M LiCl2 and 20 mM BaCl2 |
-Data collection
| Diffraction | Mean temperature: 100 K / Serial crystal experiment: N |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  Photon Factory Photon Factory  / Beamline: BL-17A / Wavelength: 0.98 Å / Beamline: BL-17A / Wavelength: 0.98 Å |
| Detector | Type: DECTRIS PILATUS 2M / Detector: PIXEL / Date: May 10, 2017 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.98 Å / Relative weight: 1 |
| Reflection | Resolution: 2.46→45.69 Å / Num. obs: 86984 / % possible obs: 99.76 % / Redundancy: 6.5 % / Biso Wilson estimate: 55.27 Å2 / CC1/2: 0.999 / Rmerge(I) obs: 0.089 / Rpim(I) all: 0.051 / Rrim(I) all: 0.095 / Net I/σ(I): 19.8 |
| Reflection shell | Resolution: 2.46→2.51 Å / Rmerge(I) obs: 1.315 / Num. unique obs: 8055 / CC1/2: 0.745 / Rpim(I) all: 0.759 / Rrim(I) all: 1.424 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT / Resolution: 2.46→45.69 Å / SU ML: 0.3515 / Cross valid method: FREE R-VALUE / σ(F): 1.34 / Phase error: 31.2135 / Stereochemistry target values: GeoStd + Monomer Library MOLECULAR REPLACEMENT / Resolution: 2.46→45.69 Å / SU ML: 0.3515 / Cross valid method: FREE R-VALUE / σ(F): 1.34 / Phase error: 31.2135 / Stereochemistry target values: GeoStd + Monomer Library
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å / Solvent model: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 75.35 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 2.46→45.69 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller










 PDBj
PDBj





