| 登録情報 | データベース: PDB / ID: 6iej
|
|---|
| タイトル | The C2 domain of cytosolic phospholipase A2 alpha bound to phosphatidylcholine |
|---|
 要素 要素 | Cytosolic phospholipase A2 |
|---|
 キーワード キーワード | HYDROLASE / Lipid binding / Phospholipase / Calcium binding / C2 domain |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
phospho-PLA2 pathway / Acyl chain remodeling of CL / Acyl chain remodelling of PI / Acyl chain remodelling of PG / Hydrolysis of LPC / Synthesis of PA / Arachidonic acid metabolism / Platelet sensitization by LDL / Acyl chain remodelling of PC / Acyl chain remodelling of PS ...phospho-PLA2 pathway / Acyl chain remodeling of CL / Acyl chain remodelling of PI / Acyl chain remodelling of PG / Hydrolysis of LPC / Synthesis of PA / Arachidonic acid metabolism / Platelet sensitization by LDL / Acyl chain remodelling of PC / Acyl chain remodelling of PS / Acyl chain remodelling of PE / glycerophospholipid catabolic process / COPI-independent Golgi-to-ER retrograde traffic / phosphatidyl phospholipase B activity / lysophospholipase / phospholipase A2 activity => GO:0004623 / phospholipase A2 activity => GO:0004623 / ADP signalling through P2Y purinoceptor 1 / lysophospholipase activity / phospholipase A2 activity / calcium-dependent phospholipid binding / calcium-dependent phospholipase A2 activity / phospholipase A2 / cytoplasmic vesicle / calcium ion binding / Golgi apparatus / endoplasmic reticulum / nucleus / cytosol類似検索 - 分子機能 Lysophospholipase, catalytic domain / Cytosolic phospholipase A2, C2 domain / Lysophospholipase catalytic domain / PLA2c domain profile. / Cytoplasmic phospholipase A2, catalytic subunit / C2 domain / Acyl transferase/acyl hydrolase/lysophospholipase / C2 domain / Protein kinase C conserved region 2 (CalB) / C2 domain ...Lysophospholipase, catalytic domain / Cytosolic phospholipase A2, C2 domain / Lysophospholipase catalytic domain / PLA2c domain profile. / Cytoplasmic phospholipase A2, catalytic subunit / C2 domain / Acyl transferase/acyl hydrolase/lysophospholipase / C2 domain / Protein kinase C conserved region 2 (CalB) / C2 domain / C2 domain profile. / C2 domain superfamily / Immunoglobulin-like / Sandwich / Mainly Beta類似検索 - ドメイン・相同性 1,2-dihexanoyl-sn-glycero-3-phosphocholine / Cytosolic phospholipase A2類似検索 - 構成要素 |
|---|
| 生物種 |   Gallus gallus (ニワトリ) Gallus gallus (ニワトリ) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.206 Å 分子置換 / 解像度: 2.206 Å |
|---|
 データ登録者 データ登録者 | Hirano, Y. / Gao, Y.G. / Stephenson, D.J. / Vu, N.T. / Malinina, L. / Chalfant, C.E. / Patel, D.J. / Brown, R.E. |
|---|
| 資金援助 |  米国, 1件 米国, 1件 | 組織 | 認可番号 | 国 |
|---|
| National Institutes of Health/National Heart, Lung, and Blood Institute (NIH/NHLBI) | HL125353 |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: Elife / 年: 2019 ジャーナル: Elife / 年: 2019
タイトル: Structural basis of phosphatidylcholine recognition by the C2-domain of cytosolic phospholipase A2alpha.
著者: Hirano, Y. / Gao, Y.G. / Stephenson, D.J. / Vu, N.T. / Malinina, L. / Simanshu, D.K. / Chalfant, C.E. / Patel, D.J. / Brown, R.E. |
|---|
| 履歴 | | 登録 | 2018年9月14日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2019年5月22日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2022年3月23日 | Group: Author supporting evidence / Database references / Derived calculations
カテゴリ: database_2 / pdbx_audit_support ...database_2 / pdbx_audit_support / pdbx_struct_conn_angle / struct_conn
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_audit_support.funding_organization / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr2_auth_seq_id / _pdbx_struct_conn_angle.ptnr2_label_asym_id / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id / _struct_conn.ptnr2_symmetry |
|---|
| 改定 1.2 | 2023年11月22日 | Group: Data collection / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond / pdbx_initial_refinement_model |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.206 Å
分子置換 / 解像度: 2.206 Å  データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: Elife / 年: 2019
ジャーナル: Elife / 年: 2019 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6iej.cif.gz
6iej.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6iej.ent.gz
pdb6iej.ent.gz PDB形式
PDB形式 6iej.json.gz
6iej.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6iej_validation.pdf.gz
6iej_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6iej_full_validation.pdf.gz
6iej_full_validation.pdf.gz 6iej_validation.xml.gz
6iej_validation.xml.gz 6iej_validation.cif.gz
6iej_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/ie/6iej
https://data.pdbj.org/pub/pdb/validation_reports/ie/6iej ftp://data.pdbj.org/pub/pdb/validation_reports/ie/6iej
ftp://data.pdbj.org/pub/pdb/validation_reports/ie/6iej
 リンク
リンク 集合体
集合体
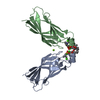
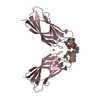
 要素
要素

 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 24-ID-C / 波長: 0.9792 Å
/ ビームライン: 24-ID-C / 波長: 0.9792 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj










