ソフトウェア 名称 バージョン 分類 データスケーリング 1.9_1692精密化 3.24 データ抽出 データ削減 位相決定
精密化 構造決定の手法 開始モデル 解像度 / SU ML / 交差検証法 / σ(F) / 位相誤差 Rfactor 反射数 %反射 Rfree 0.1823 2672 5.09 % Rwork 0.1612 - - obs 0.1622 52498 98.88 %
溶媒の処理 減衰半径 / VDWプローブ半径 原子変位パラメータ Biso max 2 / Biso mean 2 / Biso min 2 精密化ステップ サイクル / 解像度 タンパク質 核酸 リガンド 溶媒 全体 原子数 1894 0 0 358 2252 Biso mean - - - 35.41 - 残基数 - - - - 246
拘束条件 Refine-ID タイプ Dev ideal 数 X-RAY DIFFRACTION f_bond_d0.014 1994 X-RAY DIFFRACTION f_angle_d1.591 2706 X-RAY DIFFRACTION f_chiral_restr0.071 293 X-RAY DIFFRACTION f_plane_restr0.01 350 X-RAY DIFFRACTION f_dihedral_angle_d13.153 698
Refine LS restraints NCS Ens-ID Dom-ID Auth asym-ID 数 Refine-ID Rms タイプ 1 1 A1392 X-RAY DIFFRACTION 9.494 TORSIONAL1 2 B1392 X-RAY DIFFRACTION 9.494 TORSIONAL
LS精密化 シェル Refine-ID / Rfactor Rfree error / Total num. of bins used
大きな表を表示 (7 x 19) 大きな表を隠す 解像度 (Å)Rfactor Rfree Num. reflection Rfree Rfactor Rwork Num. reflection Rwork Num. reflection all % reflection obs (%)1.3-1.3236 0.2876 127 0.2648 2437 2564 94 1.3236-1.3491 0.2761 137 0.2605 2597 2734 99 1.3491-1.3766 0.2501 136 0.2336 2637 2773 100 1.3766-1.4066 0.2314 134 0.2111 2630 2764 100 1.4066-1.4393 0.2021 169 0.1924 2620 2789 100 1.4393-1.4753 0.2161 138 0.1755 2625 2763 100 1.4753-1.5152 0.1937 154 0.1759 2586 2740 100 1.5152-1.5598 0.2034 130 0.1736 2696 2826 100 1.5598-1.6101 0.1641 131 0.1627 2601 2732 100 1.6101-1.6677 0.1721 136 0.1556 2630 2766 100 1.6677-1.7344 0.1789 155 0.1542 2665 2820 100 1.7344-1.8134 0.2268 144 0.1556 2633 2777 100 1.8134-1.909 0.1729 155 0.1548 2617 2772 100 1.909-2.0286 0.1746 139 0.152 2653 2792 100 2.0286-2.1852 0.1546 133 0.1504 2660 2793 100 2.1852-2.4051 0.1753 148 0.1519 2669 2817 100 2.4051-2.753 0.1743 160 0.155 2652 2812 99 2.753-3.4682 0.1781 110 0.1493 2687 2797 99 3.4682-40.6081 0.1734 136 0.1593 2531 2667 91
精密化 TLS 手法 / Refine-ID
大きな表を表示 (25 x 14) 大きな表を隠す ID L11 (°2 )L12 (°2 )L13 (°2 )L22 (°2 )L23 (°2 )L33 (°2 )S11 (Å °)S12 (Å °)S13 (Å °)S21 (Å °)S22 (Å °)S23 (Å °)S31 (Å °)S32 (Å °)S33 (Å °)T11 (Å2 )T12 (Å2 )T13 (Å2 )T22 (Å2 )T23 (Å2 )T33 (Å2 )Origin x (Å)Origin y (Å)Origin z (Å)1 0.7342 -0.1325 1.3057 0.0662 -0.3244 2.5114 0.0215 0.3884 -0.3897 -0.3114 -0.0146 -0.2658 0.406 0.617 -0.1003 0.2666 -0.018 0.0797 0.2541 -0.0717 0.2247 15.3798 -3.7599 -1.4091 2 1.1556 0.0969 0.4567 1.3353 -0.6511 3.9995 -0.0769 -0.1065 -0.0149 0.1215 -0.1135 -0.3315 -0.2123 0.277 0.0939 0.1102 -0.0176 0.012 0.2004 -0.009 0.1589 18.8327 -2.8951 14.3912 3 2.1783 -0.6073 0.5993 1.2742 -0.471 2.6061 0.0134 -0.0272 -0.0066 -0.0496 0.0428 0.0263 -0.0205 -0.0958 -0.0587 0.1012 -0.0058 0.0197 0.1095 -0.0226 0.098 6.6364 1.7613 6.6646 4 3.08 0.3798 0.3997 2.5483 -0.1243 1.9304 -0.0845 -0.4223 0.1536 0.0763 0.0326 -0.0797 -0.3239 0.0179 0.0322 0.1601 0.0112 0.0134 0.1635 -0.0545 0.1585 9.657 5.7894 16.111 5 1.4949 0.0903 0.3081 1.5196 -0.4348 2.4831 0.023 -0.0895 0.0852 0.0917 0.0395 -0.2223 -0.2312 0.1178 -0.071 0.1303 -0.0111 0.0114 0.1489 -0.0339 0.1621 14.8878 1.5023 13.9576 6 1.4827 -0.1778 0.9268 1.1305 -0.8326 2.331 0.0741 0.014 -0.0326 -0.0141 -0.029 0.0157 0.01 0.0487 -0.0683 0.1155 -0.0026 0.0122 0.1083 -0.0178 0.1093 6.8002 -3.2269 6.671 7 6.7303 0.7872 6.1331 0.1566 0.837 5.8097 0.1664 -0.956 -0.4087 0.4414 -0.1079 0.0351 0.4624 -0.979 -0.1647 0.3002 -0.0644 -0.0229 0.2866 0.0509 0.2033 -16.5121 -4.9215 38.1815 8 1.2819 0.0355 1.078 0.9455 0.6179 5.7412 -0.0186 -0.0625 -0.135 0.0492 -0.1509 0.087 -0.2132 -0.3686 0.1051 0.1265 0.0034 0.0022 0.2129 -0.0274 0.163 -20.6327 -2.4486 23.3686 9 3.0393 0.3271 1.1982 2.4591 -0.2372 1.8627 0.0232 -0.2544 0.018 0.1981 -0.0024 -0.119 -0.1385 -0.187 -0.0216 0.132 0.0127 -0.0296 0.1181 -0.0019 0.1261 -11.2333 2.4045 36.0044 10 2.5639 0.8208 0.9474 2.8704 0.6699 3.8413 -0.1124 0.2796 -0.0914 -0.1491 0.0219 -0.1237 -0.1595 0.2885 0.0714 0.1116 -0.0091 -0.0162 0.1257 0.0001 0.1678 -7.7044 3.1755 27.2747 11 3.5213 -0.8397 0.623 2.9516 1.5589 3.0111 -0.2659 0.0616 0.3368 -0.0891 0.0872 -0.1396 -0.4839 0.3692 0.2029 0.202 -0.0159 -0.0231 0.1438 0.0185 0.1697 -12.796 6.7285 21.5416 12 1.6972 0.3681 -0.6062 1.1507 -0.1591 5.4772 -0.0335 -0.1596 0.1267 0.1107 -0.0591 0.2173 -0.4017 -0.7359 0.0857 0.1869 0.0516 0.0131 0.2505 -0.0492 0.1588 -20.0286 4.8593 31.0548 13 2.7086 -2.365 0.1484 3.3881 0.7156 1.6795 0.0334 0.5259 0.0543 -0.1826 -0.0098 -0.2907 -0.2504 0.1895 -0.1166 0.147 0.0162 0.0383 0.2312 -0.0579 0.1826 -13.8316 -2.5613 13.6537 14 1.2503 0.0827 0.7786 0.9706 0.4424 2.2413 0.1499 -0.0136 -0.2165 0.1675 -0.0397 -0.2298 0.1206 -0.0623 -0.0801 0.1333 0.0036 -0.0357 0.0944 0.0008 0.1889 -8.6041 -2.6432 30.6679
精密化 TLSグループ ID Refine-ID Refine TLS-ID Selection details Auth asym-ID Auth seq-ID 1 X-RAY DIFFRACTION 1 chain 'A' and (resid 1 through 7 )A1 - 7 2 X-RAY DIFFRACTION 2 chain 'A' and (resid 8 through 25 )A8 - 25 3 X-RAY DIFFRACTION 3 chain 'A' and (resid 26 through 60 )A26 - 60 4 X-RAY DIFFRACTION 4 chain 'A' and (resid 61 through 73 )A61 - 73 5 X-RAY DIFFRACTION 5 chain 'A' and (resid 74 through 91 )A74 - 91 6 X-RAY DIFFRACTION 6 chain 'A' and (resid 92 through 123 )A92 - 123 7 X-RAY DIFFRACTION 7 chain 'B' and (resid 1 through 7 )B1 - 7 8 X-RAY DIFFRACTION 8 chain 'B' and (resid 8 through 25 )B8 - 25 9 X-RAY DIFFRACTION 9 chain 'B' and (resid 26 through 39 )B26 - 39 10 X-RAY DIFFRACTION 10 chain 'B' and (resid 40 through 60 )B40 - 60 11 X-RAY DIFFRACTION 11 chain 'B' and (resid 61 through 73 )B61 - 73 12 X-RAY DIFFRACTION 12 chain 'B' and (resid 74 through 83 )B74 - 83 13 X-RAY DIFFRACTION 13 chain 'B' and (resid 84 through 91 )B
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.3 Å
分子置換 / 解像度: 1.3 Å  データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: J. Biol. Chem. / 年: 2018
ジャーナル: J. Biol. Chem. / 年: 2018 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6dba.cif.gz
6dba.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6dba.ent.gz
pdb6dba.ent.gz PDB形式
PDB形式 6dba.json.gz
6dba.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6dba_validation.pdf.gz
6dba_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6dba_full_validation.pdf.gz
6dba_full_validation.pdf.gz 6dba_validation.xml.gz
6dba_validation.xml.gz 6dba_validation.cif.gz
6dba_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/db/6dba
https://data.pdbj.org/pub/pdb/validation_reports/db/6dba ftp://data.pdbj.org/pub/pdb/validation_reports/db/6dba
ftp://data.pdbj.org/pub/pdb/validation_reports/db/6dba リンク
リンク 集合体
集合体


 要素
要素

 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  CLSI
CLSI  / ビームライン: 08ID-1 / 波長: 0.9795 Å
/ ビームライン: 08ID-1 / 波長: 0.9795 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー













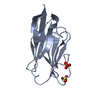
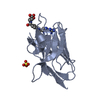


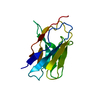
 PDBj
PDBj

