| 登録情報 | データベース: PDB / ID: 6d48
|
|---|
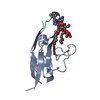
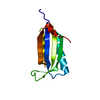
| タイトル | Cell Surface Receptor |
|---|
 要素 要素 | Myeloid cell surface antigen CD33 |
|---|
 キーワード キーワード | IMMUNE SYSTEM / Sialic Acid Binding / Transmembrane / Receptor / Siglec |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
immune response-inhibiting signal transduction / protein tyrosine phosphatase activator activity / negative regulation of monocyte activation / sialic acid binding / Fc-gamma receptor signaling pathway / negative regulation of interleukin-8 production / negative regulation of interleukin-1 beta production / tertiary granule membrane / negative regulation of tumor necrosis factor production / specific granule membrane ...immune response-inhibiting signal transduction / protein tyrosine phosphatase activator activity / negative regulation of monocyte activation / sialic acid binding / Fc-gamma receptor signaling pathway / negative regulation of interleukin-8 production / negative regulation of interleukin-1 beta production / tertiary granule membrane / negative regulation of tumor necrosis factor production / specific granule membrane / positive regulation of protein secretion / cell-cell adhesion / Immunoregulatory interactions between a Lymphoid and a non-Lymphoid cell / peroxisome / cell-cell signaling / signaling receptor activity / carbohydrate binding / protein phosphatase binding / cell adhesion / external side of plasma membrane / negative regulation of cell population proliferation / Neutrophil degranulation / cell surface / Golgi apparatus / signal transduction / plasma membrane類似検索 - 分子機能 : / Immunoglobulin / Immunoglobulin domain / Immunoglobulin V-set domain / Immunoglobulin V-set domain / Immunoglobulin subtype / Immunoglobulin / Ig-like domain profile. / Immunoglobulin-like domain / Immunoglobulin-like domain superfamily ...: / Immunoglobulin / Immunoglobulin domain / Immunoglobulin V-set domain / Immunoglobulin V-set domain / Immunoglobulin subtype / Immunoglobulin / Ig-like domain profile. / Immunoglobulin-like domain / Immunoglobulin-like domain superfamily / Immunoglobulin-like fold / Immunoglobulins / Immunoglobulin-like / Sandwich / Mainly Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.776 Å 分子置換 / 解像度: 1.776 Å |
|---|
 データ登録者 データ登録者 | Hermans, S.J. / Miles, L.A. / Parker, M.W. |
|---|
 引用 引用 |  ジャーナル: Iscience / 年: 2019 ジャーナル: Iscience / 年: 2019
タイトル: Small Molecule Binding to Alzheimer Risk Factor CD33 Promotes A beta Phagocytosis.
著者: Miles, L.A. / Hermans, S.J. / Crespi, G.A.N. / Gooi, J.H. / Doughty, L. / Nero, T.L. / Markulic, J. / Ebneth, A. / Wroblowski, B. / Oehlrich, D. / Trabanco, A.A. / Rives, M.L. / Royaux, I. / ...著者: Miles, L.A. / Hermans, S.J. / Crespi, G.A.N. / Gooi, J.H. / Doughty, L. / Nero, T.L. / Markulic, J. / Ebneth, A. / Wroblowski, B. / Oehlrich, D. / Trabanco, A.A. / Rives, M.L. / Royaux, I. / Hancock, N.C. / Parker, M.W. |
|---|
| 履歴 | | 登録 | 2018年4月17日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2019年4月17日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2019年10月30日 | Group: Data collection / Database references / カテゴリ: citation / citation_author
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year |
|---|
| 改定 1.2 | 2023年10月4日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / struct_conn
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_seq_id / _struct_conn.ptnr2_symmetry |
|---|
| 改定 1.3 | 2024年10月30日 | Group: Structure summary
カテゴリ: pdbx_entry_details / pdbx_modification_feature |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.776 Å
分子置換 / 解像度: 1.776 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Iscience / 年: 2019
ジャーナル: Iscience / 年: 2019 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6d48.cif.gz
6d48.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6d48.ent.gz
pdb6d48.ent.gz PDB形式
PDB形式 6d48.json.gz
6d48.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6d48_validation.pdf.gz
6d48_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6d48_full_validation.pdf.gz
6d48_full_validation.pdf.gz 6d48_validation.xml.gz
6d48_validation.xml.gz 6d48_validation.cif.gz
6d48_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/d4/6d48
https://data.pdbj.org/pub/pdb/validation_reports/d4/6d48 ftp://data.pdbj.org/pub/pdb/validation_reports/d4/6d48
ftp://data.pdbj.org/pub/pdb/validation_reports/d4/6d48 リンク
リンク 集合体
集合体




 要素
要素 Homo sapiens (ヒト) / 遺伝子: CD33, SIGLEC3 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: CD33, SIGLEC3 / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  Australian Synchrotron
Australian Synchrotron  / ビームライン: MX2 / 波長: 0.9537 Å
/ ビームライン: MX2 / 波長: 0.9537 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj





