[English] 日本語
 Yorodumi
Yorodumi- PDB-6c9j: AMP-activated protein kinase bound to pharmacological activator R734 -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 6c9j | |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Title | AMP-activated protein kinase bound to pharmacological activator R734 | |||||||||||||||||||||||||||
 Components Components |
| |||||||||||||||||||||||||||
 Keywords Keywords | TRANSFERASE / AMPK / activator | |||||||||||||||||||||||||||
| Function / homology |  Function and homology information Function and homology informationnegative regulation of glucosylceramide biosynthetic process / nail development / [hydroxymethylglutaryl-CoA reductase (NADPH)] kinase / [hydroxymethylglutaryl-CoA reductase (NADPH)] kinase activity / regulation of stress granule assembly / positive regulation of mitochondrial transcription / negative regulation of tubulin deacetylation / histone H2BS36 kinase activity / cold acclimation / cAMP-dependent protein kinase regulator activity ...negative regulation of glucosylceramide biosynthetic process / nail development / [hydroxymethylglutaryl-CoA reductase (NADPH)] kinase / [hydroxymethylglutaryl-CoA reductase (NADPH)] kinase activity / regulation of stress granule assembly / positive regulation of mitochondrial transcription / negative regulation of tubulin deacetylation / histone H2BS36 kinase activity / cold acclimation / cAMP-dependent protein kinase regulator activity / AMP-activated protein kinase activity / lipid droplet disassembly / Lipophagy / regulation of carbon utilization / positive regulation of skeletal muscle tissue development / CAMKK-AMPK signaling cascade / import into nucleus / regulation of vesicle-mediated transport / nucleotide-activated protein kinase complex / negative regulation of hepatocyte apoptotic process / Energy dependent regulation of mTOR by LKB1-AMPK / positive regulation of T cell mediated immune response to tumor cell / tau-protein kinase / protein kinase regulator activity / negative regulation of TOR signaling / Activation of PPARGC1A (PGC-1alpha) by phosphorylation / response to caffeine / regulation of glycolytic process / : / cAMP-dependent protein kinase activity / tau-protein kinase activity / protein localization to lipid droplet / AMP binding / cellular response to stress / Macroautophagy / cholesterol biosynthetic process / lipid biosynthetic process / fatty acid oxidation / motor behavior / positive regulation of protein kinase activity / cellular response to ethanol / fatty acid homeostasis / negative regulation of lipid catabolic process / response to UV / cellular response to glucose starvation / energy homeostasis / Activation of AMPK downstream of NMDARs / positive regulation of protein localization / positive regulation of adipose tissue development / negative regulation of TORC1 signaling / cellular response to nutrient levels / positive regulation of autophagy / positive regulation of gluconeogenesis / negative regulation of insulin receptor signaling pathway / cellular response to calcium ion / regulation of microtubule cytoskeleton organization / positive regulation of glycolytic process / response to activity / response to gamma radiation / TP53 Regulates Metabolic Genes / Translocation of SLC2A4 (GLUT4) to the plasma membrane / cellular response to glucose stimulus / positive regulation of cholesterol biosynthetic process / neuron cellular homeostasis / ADP binding / regulation of circadian rhythm / autophagy / response to estrogen / cellular response to xenobiotic stimulus / tau protein binding / glucose metabolic process / Wnt signaling pathway / cellular response to hydrogen peroxide / positive regulation of T cell activation / fatty acid biosynthetic process / rhythmic process / cellular response to prostaglandin E stimulus / glucose homeostasis / positive regulation of cold-induced thermogenesis / cellular response to oxidative stress / spermatogenesis / cellular response to hypoxia / Regulation of TP53 Activity through Phosphorylation / response to hypoxia / protein phosphorylation / protein kinase activity / non-specific serine/threonine protein kinase / apical plasma membrane / regulation of cell cycle / cilium / nuclear speck / ciliary basal body / negative regulation of gene expression / axon / protein serine kinase activity / protein serine/threonine kinase activity / neuronal cell body / positive regulation of cell population proliferation / chromatin binding / dendrite Similarity search - Function | |||||||||||||||||||||||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | |||||||||||||||||||||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 3.05 Å MOLECULAR REPLACEMENT / Resolution: 3.05 Å | |||||||||||||||||||||||||||
 Authors Authors | Yan, Y. / Zhou, X.E. / Novick, S. / Shaw, S.J. / Li, Y. / Brunzelle, J.S. / Hitoshi, Y. / Griffin, P.R. / Xu, H.E. / Melcher, K. | |||||||||||||||||||||||||||
| Funding support |  United States, United States,  China, 8items China, 8items
| |||||||||||||||||||||||||||
 Citation Citation |  Journal: J. Biol. Chem. / Year: 2019 Journal: J. Biol. Chem. / Year: 2019Title: Structures of AMP-activated protein kinase bound to novel pharmacological activators in phosphorylated, non-phosphorylated, and nucleotide-free states. Authors: Yan, Y. / Zhou, X.E. / Novick, S.J. / Shaw, S.J. / Li, Y. / Brunzelle, J.S. / Hitoshi, Y. / Griffin, P.R. / Xu, H.E. / Melcher, K. | |||||||||||||||||||||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  6c9j.cif.gz 6c9j.cif.gz | 366.9 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb6c9j.ent.gz pdb6c9j.ent.gz | 295.9 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  6c9j.json.gz 6c9j.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/c9/6c9j https://data.pdbj.org/pub/pdb/validation_reports/c9/6c9j ftp://data.pdbj.org/pub/pdb/validation_reports/c9/6c9j ftp://data.pdbj.org/pub/pdb/validation_reports/c9/6c9j | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  6c9fC  6c9gC  6c9hC  4rerS S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| Unit cell |
|
- Components
Components
-Protein , 1 types, 1 molecules A
| #1: Protein | Mass: 57245.625 Da / Num. of mol.: 1 / Mutation: S108D Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: PRKAA1, AMPK1 / Production host: Homo sapiens (human) / Gene: PRKAA1, AMPK1 / Production host:  References: UniProt: Q13131, non-specific serine/threonine protein kinase, EC: 2.7.11.27, [hydroxymethylglutaryl-CoA reductase (NADPH)] kinase, tau-protein kinase |
|---|
-5'-AMP-activated protein kinase subunit ... , 2 types, 2 molecules BC
| #2: Protein | Mass: 23071.316 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: PRKAB1, AMPK / Production host: Homo sapiens (human) / Gene: PRKAB1, AMPK / Production host:  |
|---|---|
| #3: Protein | Mass: 37027.598 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: PRKAG1 / Production host: Homo sapiens (human) / Gene: PRKAG1 / Production host:  |
-Non-polymers , 3 types, 3 molecules 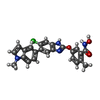



| #4: Chemical | ChemComp-R34 / |
|---|---|
| #5: Chemical | ChemComp-STU / |
| #6: Chemical | ChemComp-AMP / |
-Details
| Has protein modification | Y |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 3.77 Å3/Da / Density % sol: 67.39 % |
|---|---|
| Crystal grow | Temperature: 293 K / Method: vapor diffusion / Details: 0.1 M tri-ammonium citrate pH7, 12% w/v PEG 3350 |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  APS APS  / Beamline: 21-ID-D / Wavelength: 1 Å / Beamline: 21-ID-D / Wavelength: 1 Å |
| Detector | Type: DECTRIS EIGER X 9M / Detector: PIXEL / Date: Nov 11, 2016 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1 Å / Relative weight: 1 |
| Reflection | Resolution: 3.05→50 Å / Num. obs: 32841 / % possible obs: 91.1 % / Redundancy: 34.4 % / CC1/2: 0.997 / Rmerge(I) obs: 0.807 / Net I/σ(I): 10 |
| Reflection shell | Resolution: 3.05→3.22 Å / Rmerge(I) obs: 0.0897 / CC1/2: 0.722 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 4RER Resolution: 3.05→49.646 Å / SU ML: 0.3 / Cross valid method: FREE R-VALUE / σ(F): 1.35 / Phase error: 22.37 / Stereochemistry target values: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.8 Å / VDW probe radii: 1 Å / Solvent model: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 3.05→49.646 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller












 PDBj
PDBj












