+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6btm | |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of Alternative Complex III from Flavobacterium johnsoniae (Wild Type) | |||||||||||||||||||||||||||
 要素 要素 | (Alternative Complex III subunit ...) x 6 | |||||||||||||||||||||||||||
 キーワード キーワード | MEMBRANE PROTEIN / Electron transport chain / triacylated cysteine / heme c domain / iron-sulfur cluster | |||||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報membrane => GO:0016020 / electron transfer activity / heme binding / metal ion binding / plasma membrane 類似検索 - 分子機能 | |||||||||||||||||||||||||||
| 生物種 |  Flavobacterium johnsoniae UW101 (バクテリア) Flavobacterium johnsoniae UW101 (バクテリア) | |||||||||||||||||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||||||||||||||||||||
 データ登録者 データ登録者 | Sun, C. / Benlekbir, S. / Venkatakrishnan, P. / Yuhang, W. / Tajkhorshid, E. / Rubinstein, J.L. / Gennis, R.B. | |||||||||||||||||||||||||||
| 資金援助 |  米国, 米国,  カナダ, 8件 カナダ, 8件
| |||||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2018 ジャーナル: Nature / 年: 2018タイトル: Structure of the alternative complex III in a supercomplex with cytochrome oxidase. 著者: Chang Sun / Samir Benlekbir / Padmaja Venkatakrishnan / Yuhang Wang / Sangjin Hong / Jonathan Hosler / Emad Tajkhorshid / John L Rubinstein / Robert B Gennis /   要旨: Alternative complex III (ACIII) is a key component of the respiratory and/or photosynthetic electron transport chains of many bacteria. Like complex III (also known as the bc complex), ACIII ...Alternative complex III (ACIII) is a key component of the respiratory and/or photosynthetic electron transport chains of many bacteria. Like complex III (also known as the bc complex), ACIII catalyses the oxidation of membrane-bound quinol and the reduction of cytochrome c or an equivalent electron carrier. However, the two complexes have no structural similarity. Although ACIII has eluded structural characterization, several of its subunits are known to be homologous to members of the complex iron-sulfur molybdoenzyme (CISM) superfamily , including the proton pump polysulfide reductase. We isolated the ACIII from Flavobacterium johnsoniae with native lipids using styrene maleic acid copolymer, both as an independent enzyme and as a functional 1:1 supercomplex with an aa-type cytochrome c oxidase (cyt aa). We determined the structure of ACIII to 3.4 Å resolution by cryo-electron microscopy and constructed an atomic model for its six subunits. The structure, which contains a [3Fe-4S] cluster, a [4Fe-4S] cluster and six haem c units, shows that ACIII uses known elements from other electron transport complexes arranged in a previously unknown manner. Modelling of the cyt aa component of the supercomplex revealed that it is structurally modified to facilitate association with ACIII, illustrating the importance of the supercomplex in this electron transport chain. The structure also resolves two of the subunits of ACIII that are anchored to the lipid bilayer with N-terminal triacylated cysteine residues, an important post-translational modification found in numerous prokaryotic membrane proteins that has not previously been observed structurally in a lipid bilayer. | |||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6btm.cif.gz 6btm.cif.gz | 442.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6btm.ent.gz pdb6btm.ent.gz | 347.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6btm.json.gz 6btm.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6btm_validation.pdf.gz 6btm_validation.pdf.gz | 1.4 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6btm_full_validation.pdf.gz 6btm_full_validation.pdf.gz | 1.4 MB | 表示 | |
| XML形式データ |  6btm_validation.xml.gz 6btm_validation.xml.gz | 61.5 KB | 表示 | |
| CIF形式データ |  6btm_validation.cif.gz 6btm_validation.cif.gz | 94.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/bt/6btm https://data.pdbj.org/pub/pdb/validation_reports/bt/6btm ftp://data.pdbj.org/pub/pdb/validation_reports/bt/6btm ftp://data.pdbj.org/pub/pdb/validation_reports/bt/6btm | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-Alternative Complex III subunit ... , 6種, 6分子 ABCDEF
| #1: タンパク質 | 分子量: 48236.250 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Flavobacterium johnsoniae UW101 (バクテリア) Flavobacterium johnsoniae UW101 (バクテリア)株: ATCC 17061 / DSM 2064 / UW101 / 参照: UniProt: A5FJF1 |
|---|---|
| #2: タンパク質 | 分子量: 102911.984 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Flavobacterium johnsoniae UW101 (バクテリア) Flavobacterium johnsoniae UW101 (バクテリア)株: ATCC 17061 / DSM 2064 / UW101 / 参照: UniProt: A5FJF2 |
| #3: タンパク質 | 分子量: 53269.992 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Flavobacterium johnsoniae UW101 (バクテリア) Flavobacterium johnsoniae UW101 (バクテリア)株: ATCC 17061 / DSM 2064 / UW101 / 参照: UniProt: A5FJF3 |
| #4: タンパク質 | 分子量: 19720.820 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Flavobacterium johnsoniae UW101 (バクテリア) Flavobacterium johnsoniae UW101 (バクテリア)株: ATCC 17061 / DSM 2064 / UW101 / 参照: UniProt: A5FJF4 |
| #5: タンパク質 | 分子量: 18179.270 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Flavobacterium johnsoniae UW101 (バクテリア) Flavobacterium johnsoniae UW101 (バクテリア)株: ATCC 17061 / DSM 2064 / UW101 / 参照: UniProt: A5FJF5 |
| #6: タンパク質 | 分子量: 52926.332 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  Flavobacterium johnsoniae UW101 (バクテリア) Flavobacterium johnsoniae UW101 (バクテリア)株: ATCC 17061 / DSM 2064 / UW101 / 参照: UniProt: A5FJE2 |
-非ポリマー , 5種, 12分子 


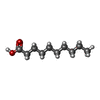
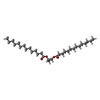




| #7: 化合物 | ChemComp-HEC / #8: 化合物 | ChemComp-F3S / | #9: 化合物 | ChemComp-SF4 / | #10: 化合物 | #11: 化合物 | |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: ACIII-cyt aa3 supercomplex / タイプ: COMPLEX 詳細: Alternative complex III-- cytochrome aa3 oxidase supercomplex purified with styrene maleic acid copolymer Entity ID: #1, #3-#4, #6 / 由来: NATURAL |
|---|---|
| 由来(天然) | 生物種:  Flavobacterium johnsoniae UW101 (バクテリア) Flavobacterium johnsoniae UW101 (バクテリア) |
| 緩衝液 | pH: 8 |
| 試料 | 濃度: 3 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 試料支持 | グリッドの材料: GOLD |
| 急速凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD |
| 撮影 | 平均露光時間: 10 sec. / 電子線照射量: 61 e/Å2 / 検出モード: COUNTING フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 撮影したグリッド数: 2 / 実像数: 2017 |
| 画像スキャン | 横: 3838 / 縦: 3710 / 動画フレーム数/画像: 50 / 利用したフレーム数/画像: 1-40 |
- 解析
解析
| ソフトウェア | 名称: PHENIX / バージョン: dev_2722: / 分類: 精密化 | ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 3044 | ||||||||||||||||||||||||||||
| 対称性 | 点対称性: C1 (非対称) | ||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.4 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 164239 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: OTHER / 空間: REAL | ||||||||||||||||||||||||||||
| 精密化 | 最高解像度: 3.4 Å | ||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj


















