| #1: タンパク質 | C-6' aminotransferase / Gentamicin (Hexosaminyl-6-) aminotransferase I / GntW / Putative glutamate-1-semialdehyde aminotransferase
分子量: 47174.406 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)  Micromonospora echinospora (バクテリア)遺伝子 Micromonospora echinospora (バクテリア)遺伝子: gacK, genB1, gntW / 発現宿主:   Escherichia coli (大腸菌) / 参照 Escherichia coli (大腸菌) / 参照: UniProt: Q70KD9 | #2: 化合物 | ChemComp-PLP / PYRIDOXAL-5'-PHOSPHATE / VITAMIN B6 Phosphate / ピリドキサ-ル5′-りん酸
  分子量 分子量: 247.142 Da / 分子数: 2 / 由来タイプ: 合成 / 式: C 8H 10NO 6P / タイプ: SUBJECT OF INVESTIGATION | #3: 糖 | ChemComp-XXX / (1R,2R,3S,4R,6S)-4,6-diamino-2,3-dihydroxycyclohexyl 2,6-diamino-2,6-dideoxy-alpha-D-glucopyranoside / (2R,3S,4R,5R,6R)-6-((1R,2R,3S,4R,6S)-4,6-DIAMINO-2,3-DIHYDROXYCYCLOHEXYLOXY)-5-AMINO-2-(AMINOMETHYL)-TETRAHYDRO-2H-PYRA N-3,4-DIOL / NEOMYCIN A / NEAMINE / (1R,2R,3S,4R,6S)-4,6-diamino-2,3-dihydroxycyclohexyl 2,6-diamino-2,6-dideoxy-alpha-D-glucoside / (1R,2R,3S,4R,6S)-4,6-diamino-2,3-dihydroxycyclohexyl 2,6-diamino-2,6-dideoxy-D-glucoside / (1R,2R,3S,4R,6S)-4,6-diamino-2,3-dihydroxycyclohexyl 2,6-diamino-2,6-dideoxy-glucoside / ネアミン
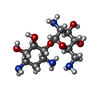  タイプ タイプ: D-saccharide / 分子量: 322.358 Da / 分子数: 2 / 由来タイプ: 合成 / 式: C 12H 26N 4O 6 / タイプ: SUBJECT OF INVESTIGATION / コメント: 抗生剤 *YM| #4: 水 | ChemComp-HOH / water
 分子量 分子量: 18.015 Da / 分子数: 626 / 由来タイプ: 天然 / 式: H 2O |
|---|
|
|---|
|
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Micromonospora echinospora (バクテリア)
Micromonospora echinospora (バクテリア) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.759 Å
分子置換 / 解像度: 1.759 Å  データ登録者
データ登録者 韓国, 1件
韓国, 1件  引用
引用 ジャーナル: Nat. Chem. Biol. / 年: 2019
ジャーナル: Nat. Chem. Biol. / 年: 2019 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5z8k.cif.gz
5z8k.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5z8k.ent.gz
pdb5z8k.ent.gz PDB形式
PDB形式 5z8k.json.gz
5z8k.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5z8k_validation.pdf.gz
5z8k_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5z8k_full_validation.pdf.gz
5z8k_full_validation.pdf.gz 5z8k_validation.xml.gz
5z8k_validation.xml.gz 5z8k_validation.cif.gz
5z8k_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/z8/5z8k
https://data.pdbj.org/pub/pdb/validation_reports/z8/5z8k ftp://data.pdbj.org/pub/pdb/validation_reports/z8/5z8k
ftp://data.pdbj.org/pub/pdb/validation_reports/z8/5z8k リンク
リンク 集合体
集合体
 要素
要素 Micromonospora echinospora (バクテリア)
Micromonospora echinospora (バクテリア)
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト: PAL/PLS
シンクロトロン / サイト: PAL/PLS  / ビームライン: 5C (4A) / 波長: 0.9796 Å
/ ビームライン: 5C (4A) / 波長: 0.9796 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj




