| 登録情報 | データベース: PDB / ID: 5z80
|
|---|
| タイトル | Solution structure for the 1:1 complex of a platinum(II)-based tripod bound to a hybrid-1 human telomeric G-quadruplex |
|---|
 要素 要素 | G-quadruplex DNA (26-MER) |
|---|
 キーワード キーワード | DNA / Intramolecular G-quadruplex / Human Telomeric G-quadruplex / Platinum(II) complex / Ligand-DNA complex |
|---|
| 機能・相同性 | Chem-9F0 / DNA / DNA (> 10) 機能・相同性情報 機能・相同性情報 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 | 溶液NMR / simulated annealing / DGSA-distance geometry simulated annealing / molecular dynamics |
|---|
 データ登録者 データ登録者 | Liu, W.T. / Zhong, Y.F. / Liu, L.Y. / Zeng, W.J. / Wang, F.Y. / Yang, D.Z. / Mao, Z.W. |
|---|
| 資金援助 |  中国, 4件 中国, 4件 | 組織 | 認可番号 | 国 |
|---|
| 973 program | 2014CB845604 and 2015CB856301 |  中国 中国 | | National Science Foundation of China | 21231007 and 21572282 |  中国 中国 | | Ministry of Education of China | IRT-17R111 |  中国 中国 | | Science and Technology Planning Project of Guangdong Province | 2013B051000047 and 207999 |  中国 中国 |
|
|---|
 引用 引用 |  ジャーナル: Nat Commun / 年: 2018 ジャーナル: Nat Commun / 年: 2018
タイトル: Solution structures of multiple G-quadruplex complexes induced by a platinum(II)-based tripod reveal dynamic binding
著者: Liu, W. / Zhong, Y.F. / Liu, L.Y. / Shen, C.T. / Zeng, W. / Wang, F. / Yang, D. / Mao, Z.W. |
|---|
| 履歴 | | 登録 | 2018年1月30日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2018年9月19日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2024年5月1日 | Group: Data collection / Database references
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_nmr_software
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_software.name |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 中国, 4件
中国, 4件  引用
引用 ジャーナル: Nat Commun / 年: 2018
ジャーナル: Nat Commun / 年: 2018 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5z80.cif.gz
5z80.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5z80.ent.gz
pdb5z80.ent.gz PDB形式
PDB形式 5z80.json.gz
5z80.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5z80_validation.pdf.gz
5z80_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5z80_full_validation.pdf.gz
5z80_full_validation.pdf.gz 5z80_validation.xml.gz
5z80_validation.xml.gz 5z80_validation.cif.gz
5z80_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/z8/5z80
https://data.pdbj.org/pub/pdb/validation_reports/z8/5z80 ftp://data.pdbj.org/pub/pdb/validation_reports/z8/5z80
ftp://data.pdbj.org/pub/pdb/validation_reports/z8/5z80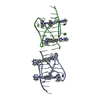
 リンク
リンク 集合体
集合体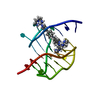
 要素
要素 Homo sapiens (ヒト)
Homo sapiens (ヒト) 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj







































 X-PLOR
X-PLOR