[English] 日本語
 Yorodumi
Yorodumi- PDB-5wiy: Kelch domain of human Keap1 bound to small molecule inhibitor fra... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 5wiy | ||||||
|---|---|---|---|---|---|---|---|
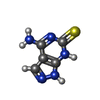
| Title | Kelch domain of human Keap1 bound to small molecule inhibitor fragment: 4-amino-1,7-dihydro-6H-pyrazolo[3,4-d]pyrimidine-6-thione | ||||||
 Components Components | Kelch-like ECH-associated protein 1 | ||||||
 Keywords Keywords | TRANSCRIPTION/INHIBITOR / Scaffold / Inhibitor / Fragment / TRANSCRIPTION / TRANSCRIPTION-INHIBITOR complex | ||||||
| Function / homology |  Function and homology information Function and homology informationregulation of epidermal cell differentiation / Nuclear events mediated by NFE2L2 / negative regulation of response to oxidative stress / Cul3-RING ubiquitin ligase complex / transcription regulator inhibitor activity / ubiquitin-like ligase-substrate adaptor activity / inclusion body / cellular response to interleukin-4 / regulation of autophagy / actin filament ...regulation of epidermal cell differentiation / Nuclear events mediated by NFE2L2 / negative regulation of response to oxidative stress / Cul3-RING ubiquitin ligase complex / transcription regulator inhibitor activity / ubiquitin-like ligase-substrate adaptor activity / inclusion body / cellular response to interleukin-4 / regulation of autophagy / actin filament / centriolar satellite / disordered domain specific binding / KEAP1-NFE2L2 pathway / Antigen processing: Ubiquitination & Proteasome degradation / positive regulation of proteasomal ubiquitin-dependent protein catabolic process / Neddylation / cellular response to oxidative stress / midbody / ubiquitin-dependent protein catabolic process / Potential therapeutics for SARS / in utero embryonic development / proteasome-mediated ubiquitin-dependent protein catabolic process / RNA polymerase II-specific DNA-binding transcription factor binding / Ub-specific processing proteases / protein ubiquitination / negative regulation of transcription by RNA polymerase II / endoplasmic reticulum / nucleoplasm / identical protein binding / cytoplasm / cytosol Similarity search - Function | ||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.23 Å MOLECULAR REPLACEMENT / Resolution: 2.23 Å | ||||||
 Authors Authors | Carolan, J.P. / Lynch, A.J. / Allen, K.N. | ||||||
| Funding support |  United States, 1items United States, 1items
| ||||||
 Citation Citation |  Journal: Biochemistry / Year: 2020 Journal: Biochemistry / Year: 2020Title: Interaction Energetics and Druggability of the Protein-Protein Interaction between Kelch-like ECH-Associated Protein 1 (KEAP1) and Nuclear Factor Erythroid 2 Like 2 (Nrf2). Authors: Zhong, M. / Lynch, A. / Muellers, S.N. / Jehle, S. / Luo, L. / Hall, D.R. / Iwase, R. / Carolan, J.P. / Egbert, M. / Wakefield, A. / Streu, K. / Harvey, C.M. / Ortet, P.C. / Kozakov, D. / ...Authors: Zhong, M. / Lynch, A. / Muellers, S.N. / Jehle, S. / Luo, L. / Hall, D.R. / Iwase, R. / Carolan, J.P. / Egbert, M. / Wakefield, A. / Streu, K. / Harvey, C.M. / Ortet, P.C. / Kozakov, D. / Vajda, S. / Allen, K.N. / Whitty, A. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  5wiy.cif.gz 5wiy.cif.gz | 237.9 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb5wiy.ent.gz pdb5wiy.ent.gz | 191.1 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  5wiy.json.gz 5wiy.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/wi/5wiy https://data.pdbj.org/pub/pdb/validation_reports/wi/5wiy ftp://data.pdbj.org/pub/pdb/validation_reports/wi/5wiy ftp://data.pdbj.org/pub/pdb/validation_reports/wi/5wiy | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  5wflSC  5wfvC  5wg1C  5whlC  5whoC S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| Unit cell |
|
- Components
Components
| #1: Protein | Mass: 36899.176 Da / Num. of mol.: 2 / Mutation: E540A, E542A, C613S Source method: isolated from a genetically manipulated source Details: His-tagged variant including mutations for altered crystallographic packing Source: (gene. exp.)  Homo sapiens (human) / Gene: KEAP1, INRF2, KIAA0132, KLHL19 / Production host: Homo sapiens (human) / Gene: KEAP1, INRF2, KIAA0132, KLHL19 / Production host:  #2: Chemical | #3: Chemical | ChemComp-1XM / | #4: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.64 Å3/Da / Density % sol: 53.4 % |
|---|---|
| Crystal grow | Temperature: 290 K / Method: vapor diffusion, hanging drop Details: 1.2 - 1.5 M Ammonium sulfate, 0.1 M Bis-Tris pH = 6.0 - 6.5 PH range: 6.0 - 6.5 |
-Data collection
| Diffraction | Mean temperature: 100 K / Ambient temp details: Nitrogen stream |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  NSLS NSLS  / Beamline: X29A / Wavelength: 1.075 Å / Beamline: X29A / Wavelength: 1.075 Å |
| Detector | Type: ADSC QUANTUM 315 / Detector: CCD / Date: Jul 21, 2014 Details: 12 V servo motors utilizing a CAMAC-based E500 stepper controller |
| Radiation | Monochromator: Double crystals monochromator / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1.075 Å / Relative weight: 1 |
| Reflection | Resolution: 2.23→51.93 Å / Num. obs: 37651 / % possible obs: 99.41 % / Redundancy: 8.7 % / CC1/2: 0.909 / Rmerge(I) obs: 0.8581 / Net I/σ(I): 28.5 |
| Reflection shell | Resolution: 2.23→2.31 Å / Redundancy: 8.2 % / Rmerge(I) obs: 2.115 / Mean I/σ(I) obs: 2.46 / Num. unique obs: 3724 / CC1/2: 0.225 / % possible all: 98.37 |
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 5WFL Resolution: 2.23→51.926 Å / SU ML: 0.32 / Cross valid method: FREE R-VALUE / σ(F): 1.34 / Phase error: 25.42 / Stereochemistry target values: ML
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å / Solvent model: FLAT BULK SOLVENT MODEL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 2.23→51.926 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller










 PDBj
PDBj